前面我更新了针对生信工程师R语言入门指南,见:生信分析人员如何系统入门R(2019更新版) ,广受好评,反响热烈,趁热打铁我应该把剩余的3个知识点也认真系统更新一下,恰好昨天授课讲解的就是linux学习路线图!
在生信分析人员如何系统入门R(2019更新版) 里面,我提到过Linux基本上几十年都没有怎么变动过基础知识的,哪怕你现在搜索到十几年前的Linux教学视频,也不会觉得尴尬。而且Linux属于IT工程师必备技能,IT的发展程度远超于我们,再加上各种马哥鸟叔,还有黑马训练营公开30天完整教学视频,按照道理我是没有必要在他们IT专业人士面前班门弄虎的, 毕竟他们随便拿几个偏门知识点就可以问倒我了!不过我们生信技能树的特色是主打生物信息学方向技能建设,而它作为一个典型的教交叉学科,想在此领域成为一个专业靠谱的生信工程师,我们实在是做不到在任何一个非核心知识点投入过多的时间和精力。
写在前面
可以把Linux的学习过程分成6个阶段 ,一般来说,每个阶段都需要至少一天以上的学习:
第1阶段:把linux系统玩得跟Windows或者MacOS那样的桌面操作系统一样顺畅,主要目的就是去可视化,熟悉黑白命令行界面,可以仅仅以键盘交互模式完成常规文件夹及文件管理工作。
第2阶段:做到文本文件的表格化处理,类似于以键盘交互模式完成Excel表格的排序、计数、筛选、去冗余,查找,切割,替换,合并,补齐,熟练掌握awk,sed,grep这文本处理的三驾马车。
第3阶段:元字符,通配符及shell中的各种扩展,从此linux操作不在神秘!
第4阶段:高级目录管理:软硬链接,绝对路径和相对路径,环境变量
第5阶段:任务提交及批处理,脚本编写解放你的双手
第6阶段:软件安装及conda管理,让linux系统实用性放飞自我
仅仅是以自己多年处理生物信息学数据经验给大家总结一下Linux该如何学,希望这个给初学者的知识点路线图能帮助到你。
第一阶段:常规文件夹及文件管理工作
大家这个时候可以回忆一下自己在Windows或者MacOS这样的桌面操作系统日常学习办公主要是做什么,比如会在C盘D盘存放不同的资料,不同的文件夹分门别类,处理各种文件类型,pdf的文献,word的手稿,txt的文本等等,整理资料,文件夹挪动,删除不需要的东西,发送到桌面快捷方式等等,这些都是大家通过鼠标在视窗操作系统,所见即所得的模式完成。
而在LINUX的命令行,一切都不一样,传统意义的鼠标几乎失去了功能,只能是靠键盘发送具有特定规则的命令来与我们的LINUX电脑进行交互。你首先需要根据IP和用户名密码连接远程服务器(使用Xshell,finalshell ,SecureCRT,Putty,VNC等具有ssh功能的软件均可),这一步就会拦住很多人。
如果你是Windows电脑,这个时候可以选择安装一个git软件,默认安装后就可以右键点击进入bash here环境,可以小范围的测试一些常规文件夹及文件管理命令,如果你是MAC电脑,那么自带的终端(terminal) 也可以练习这些命令。
这个时候我们需要学习的常规文件夹及文件管理基础命令不多:
ls ## list 列出当前路径下信息
pwd ## print working directory 打印工作目录,即当前所在目录
cd ## change directory 切换目录
mkdir ## make directory建立一个新的目录
touch ## 创建文本
mv ## Move 更改文件或目录,移动目录或文件
rm ## ReMove 删除目录或文件
cp ## copy and paste 将给出的文件或目录复制到另一个文件或目录中 tar ## Tape archive 解压文件
ln ## LINk 链接文件
可以看到,大部分命令都是其英文单词的缩写,毕竟计算机大部分基础建设都是基于英文体系的,虽然命令不多,但是参数组合不少。
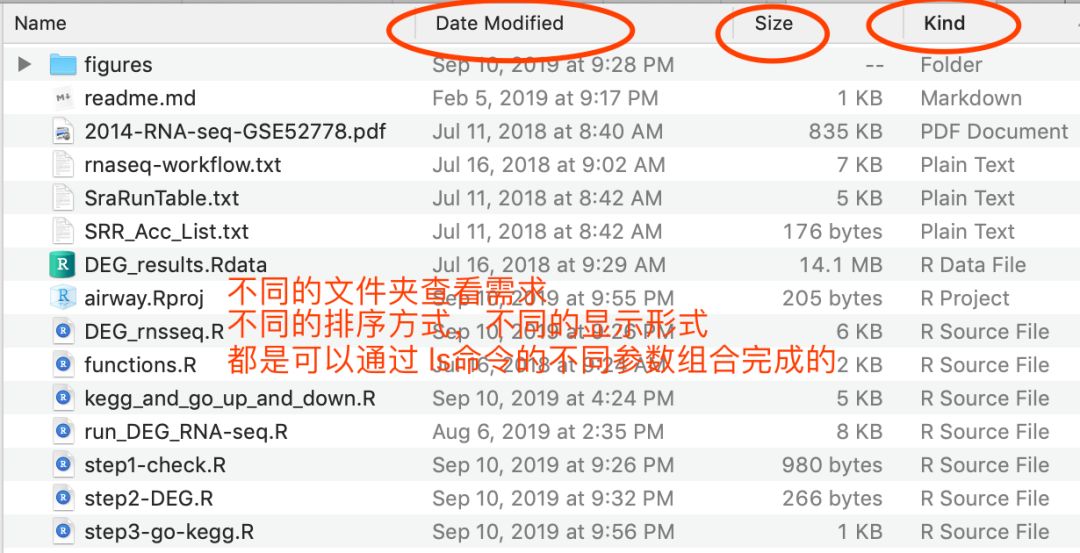
比如ls命令,就是lst , 可以列出当前路径下信息,就有大量的参数:
## 常见参数
-a :全部的目录,连同隐藏文件夹一起列出来
-h :将目录容量转换为以易读的方式(例如 GB, KB 等等) -l :列出目录的详细信息
-S :以文件大小排序
-t :以时间排序
-R : 递归目录列出文件
-d : 显示目录本身,而非目录下文件
你需要结合你自己在Windows或者MacOS这样的桌面操作系统日常学习办公实际需求来理解和学习它,你可以查看文件夹下面的文件信息,也可以查看详情,也可以按照文件大小,文件修改时间排序,那么这些功能就通通是可以命令行化!
当然,你也可以不在自己电脑练习,比如选择使用腾讯云实验室的linux服务器:https://cloud.tencent.com/developer/labs/lab/10000 或者干脆购买一个最低配版本云服务器,应该是十块钱一个月, 就可以练习上面的命令。
具体的每个命令,这里就不展开讲解了,大家可以对着命令的帮助文档或者示例演练教程慢慢练习和体会,不得不提的 man 命令可以查看大部分 文件夹及文件管理基础命令的标准文档。
第二阶段:文本文件处理大全
提到文本文件,在Windows或者MacOS这样的桌面操作系统里面,通常是txt后缀和Excel文件,最常见的需求就是打开看了,这里必须强调一下,在linux命令行环境下面,只有普通的txt文本文件是可以直接查看的,这个时候我们需要熟练使用3个配对操作命令:
head # 功能:显示文档的开头至标准输出中,默认显示十行。
tail # 功能:显示文档的末尾至标准输出中,默认显示十行。
less # 功能:逐页查看文档内容。
more # 也是逐页查看文档内容,跟less类似
cat # cat 命令 (concatenate) 查看文本,输出到屏幕
tac # tac 命令 反向查看,是cat命令的补充
同样的,每个命令都是有着不同的参数,来扩展它们的功能,比如cat命令就可以加上
-A:显示全部内容,包括特殊字符,可列出一些特殊字符而不是空白而已;
-b:列出行号,仅针对非空白行做行号显示,空白行不标行号!
-E:将结尾的断行字符$显示出来;
-n:打印出行号,连同空白行也会有行号,与-b的选项不同:
-T:将[tab]按键以I显示出来;
-v:列出一些看不出来的特殊字符
也是需要初学者花费时间去一个个体验学习,其中值得一提的less命令是一个交互式命令,假设你开始学习它了,就需要知道如何退出。
因为在命令行是没有Excel这样的软件的,但是我们文本文件的表格化处理的需求是仍然存在的,所以就需要学习大量的类似于Excel表格功能的命令,主要是排序、提取列,计数、筛选、去冗余,查找,切割,替换,合并,补齐。
sort #排序, sort lines of text files 对文件的数据进行排序(默认根据ASCII表升序排列)
cut # 提取列,可以以列(字段)为单位处理数据
wc # 计数
grep # 筛选
uniq # 去冗余
grep # 查找
awk # 切割
tr,sed # 替换
cat, paste # 合并,补齐
这些命令也很好练习,还是根据帮助文档和配套习题即可,在linux系统里面,甚至可以针对一个文件就玩转全部的这些命令。
初学者需要花费时间和精力来体验它们的各种参数效果。比如grep就可以进行查找和筛选,提供它这些功能的参数主要是:
-v ## 逆向匹配
-w ## 匹配上整个words
-B ## 输出匹配行之前的指定的行数 -A ## 输出匹配行之后的指定的行数 -E ## 指定支持扩展表达式
-C ## 对匹配到的行计数
-n ##输出计数后的一行
-o ## -n形式输出匹配的内容
值得一提的是,grep,awk,sed被我称为linux下的文本处理三驾马车,其中awk和sed命令, 还专门有一个400页的书籍来进行讲解,这里就不强调大家称为两个命令高手了,一定要是有需求再学习,但是需要对它有一定的基础认知,做到随学随用!
第三阶段:元字符,通配符及shell中的各种扩展
完成了前面两个阶段的学习,可以得心应手的的操作各种文件夹及文件,还能对文本文件进行类Excel表格探索, 就差不多是半只脚踏入linux大门了。很明显这样仍然是不够的,充其量你只是换了一个linux环境来完成你之前在Windows或者MacOS这样的桌面操作系统里面的日常办公罢了,linux去可视化的魅力你完全没有体会到。比如你想通过鼠标点击的方式新建10个,100个,甚至1000个文件夹,可能是会到猴年马月才能完成,但是在linux命令行,只需要短小精悍的一句话即可:
mkdir -p dir{1..1000}
我们想要的1000个文件夹就瞬间生成,而且还可以在文件夹下面继续嵌套,甚至都不需要进入该文件夹。
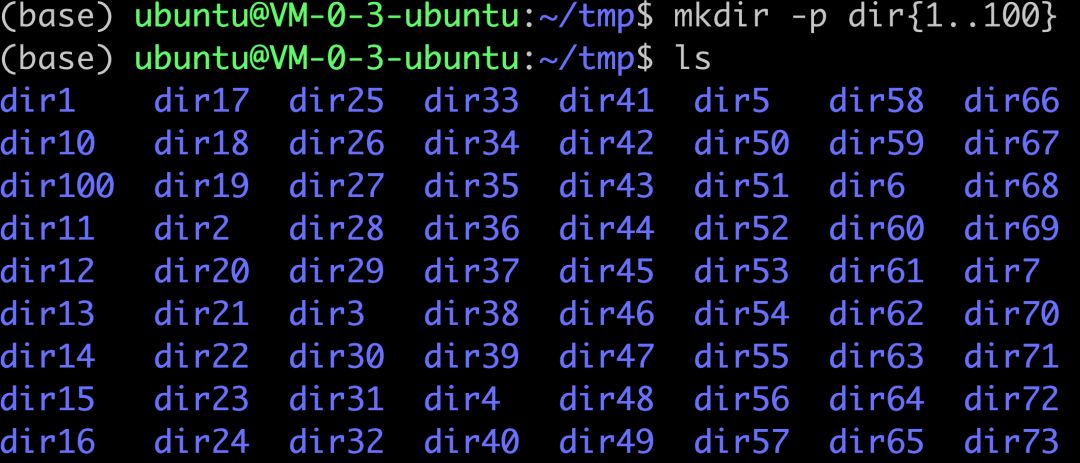
我们之所以可以在linux达到如此惊人的工作效率,得益于我们对其高级知识的掌握。这里就是{1..100}语法,是shell的扩展,shell扩展有以下几种,并按以下顺序处理,当然如果没找到匹配的扩展格式,那就不处理
brace expansion 大括号({})扩展
tilde expansion ~字符扩展
parameter and variable expansion 参数和变量扩展
arithmetic expansion 算术扩展
command substitution 命令替换
process substitution 过程替换
word splitting
Filename Expansion 通配符扩展
以上扩展中,只有brace expansion,word splitting,filename expansion 三种扩展可以改变token个数,我们演示的{1..100}语法就是这个大括号扩展(brace expansion)的序列输出功能,其中两个点是进行序列输出,然后外面套的大括号是进行扩展,这样我们就一下子新建了成百上千个文件夹。
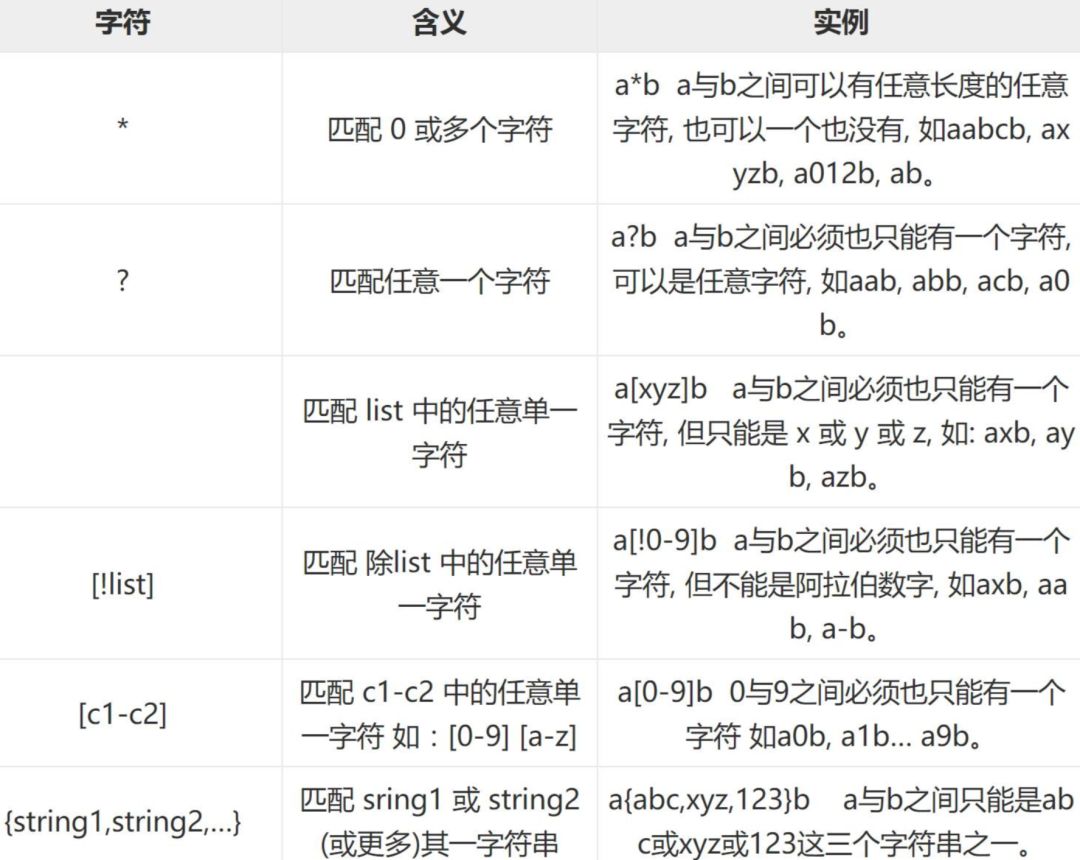
然后需要掌握的知识点,是linux shell通配符(wildcard),实际上就是一种shell实现的路径扩展功能,就是前面提到的Filename Expansion 通配符扩展。在 通配符被处理后, shell会先完成该命令的重组,然后再继续处理重组后的命令,直至执行该命令。
shell常见通配符不多,如下图:
加快效率的最后一个必备知识点就是shell元字符(特殊字符 Meta),就是一系列自己的其他特殊字符。比如我们在演示 cd 这个命令的时候,提到过一系列高级操作,如下:
cd ## 回到用户家目录
cd ~ ## 回到用户家目录
cd - # 回到前个目录
cd .. ## 切换到上层目录,相对路径
cd ../.. ## 切换到上上层目录
cd / ## 切换到根目录
cd /teach/ ## 切换到根目录下的teach,绝对路径
这里面的特殊字符就是shell元字符啦,还有一系列其它shell元字符,如下表:

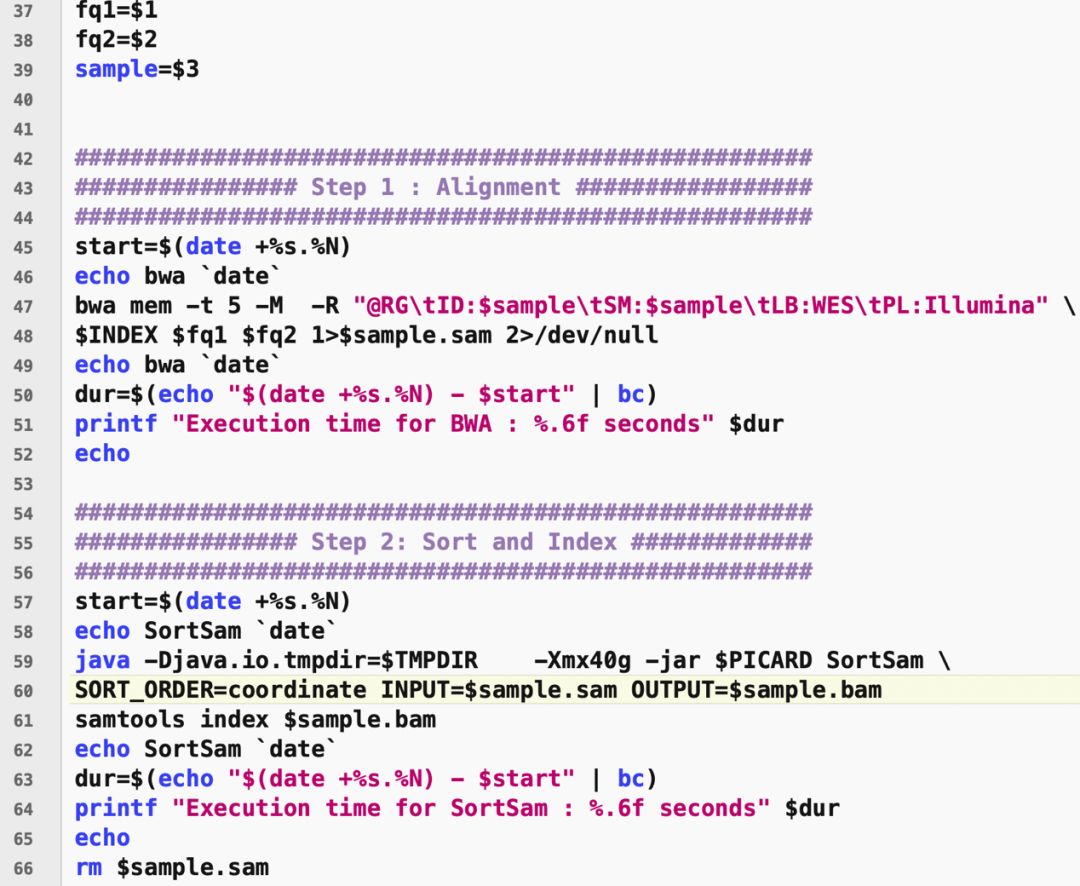
熟练掌握这些特殊字符,就可以很容易看懂大神那些看起来是鬼画符一样的NGS流程啦,比如:
第四阶段:高级目录管理
主要是理解软硬链接,绝对路径和相对路径,环境变量这些概念,以前你在Windows或者MacOS这样的桌面操作系统里面是接触不到这些概念的,因为那些电脑是需要卖给千家万户,各个阶层的普通人,要足够傻瓜化的操作才符合大众的定位和认知。但是我们生信工程师作为某种程度的专业人士,要学的更深入,看的更远。
比如,你可以在视窗操作系统打开QQ或者微信这样的界面版软件,在上面谈天说地,打开网络浏览器畅游知识的海洋,你肯定不会去思考,为什么我点击桌面的QQ图表,电脑就自动打开了QQ这个软件呢?同样的,你也不会想到,为什么我是linux的命令行交互界面输入 ls 就能列出我当前文件夹里面的内容呢?
用type命令用来显示指定命令的类型,判断给出的指令是内部指令还是外部指令。主要的命令类型:
alias:别名。
keyword:关键字,Shell保留字。
function:函数,Shell函数。
builtin:内建命令,Shell内建命令。
file:文件,磁盘文件,外部命令。
unfound:没有找到。
你会发现很多命令,都存在于下面的目录:
/usr/local/sbin
/usr/local/bin
/usr/sbin
/usr/bin
/sbin
/bin
这难道是一种巧合吗?当然不是啦, 如果你 echo $PATH 就能发现这些目录都是存储在 PATH 这个变量,在shell里面的变量需要使用 这个美元符合来进行标识。这个PATH变量就是我们的环境变量啦,掌握它是linux的一个分水岭,我们生物信息学领域大名鼎鼎的conda软件,就是通过修改你的环境变量PATH值来进行软件管理。
不仅仅是PATH,linux系统还预设了大量的变量,大全如下:
$SHELL 默认Shell
$HOME 当前用户家目录
$IFS 内部字段分隔符
$LANG 默认语言
$PATH 默认可执行程序路径
$PWD 当前目录
$UID 当前用户ID
$USER 当前用户
$HISTSIZE 历史命令大小,可通过HISTTIMEFORMAT变量设置命令执行时间
$RANDOM 随机生成一个0至32767的整数
$HOSTNAME 主机名
$0:保存当前程序或脚本的名称
$*:保存传递给脚本或进程的所有参数
$$:当前进程给脚本的PID号
$!:后台运行的最后一个进程的PID号
$?:用于返回上一条命令是否成功执行。如果成功执行,将返回数字0,否则返回非零数字(通常情况下都返回数字1)。
$#:用于保存脚本的参数个数
其实归根结底,是对shell的变量这个概念的掌握。
然后两个比较容易被忽略,但是也确实非常重要的概念是绝对路径和相对路径:
虽然路径这个知识点简单,但是确实大家容易犯错的,比如:
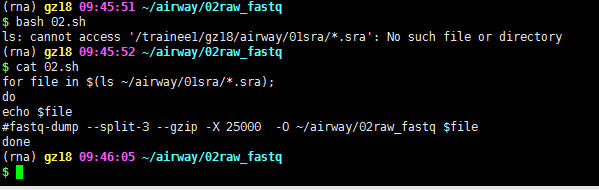
学员就会以为是自己的脚本有问题,实际上报错写的清清楚楚,找不到文件,就是绝对路径和相对路径的问题,有时候其它文件夹路径太深,我们会采取软链接的方式来减轻代码量或者某个文件夹需要经常访问,也可以链接到主目录下面,避免每次cd太多层目录。
其实就是大家桌面的快捷方式,点击就可以直接进入你存放在D盘某个文件夹深处,避免每次频繁点击进入查找。
第五阶段:任务提交及批处理,脚本编写
任务提交在生物信息学领域是一个刚需,因为大量的NGS数据处理流程耗时都很长,如果仅仅是运行命令会面临电脑的命令行交互界面被占用几个小时以上时间,需要提交任务到其它节点,取决于大家的实际计算机资源,或者在单机上面直接提交到后台,就是格式是 nohup (要提交的任务) & , 比如我们提交一个命令
sleep 100
# 这个命令提交后系统进入睡眠状态,时间是100秒,它会占用我们的命令行,再输入任何命令也不起作用。
# 我们可以用下面的命令,来把任务提交到后台运行,这样我们就可以继续使用命令行。
nohup sleep 100 &
# 提交任务后可以通过 ps -ef | grep sleep 来查看后台sleep任务的运行情况
# 也可以提供fg等命令把挂在后台的命令拿回来
批处理更多的时候属于编程的概念了,我们前面演示的短小精悍的一句话即可1000个文件夹瞬间生成
mkdir -p dir{1..1000}
就是一种批处理的概念,等价于循环,我们这里只推荐大家学习两个语法;
for i in {1..1000};do (mkdir -p dir${i});done
echo {1..1000}|tr ' ' '\n'| while read id;do ( mkdir -p dir${id});done
基本上来说,掌握了 for和while就足够了,脚本编写就更为复杂了,大纲如下:
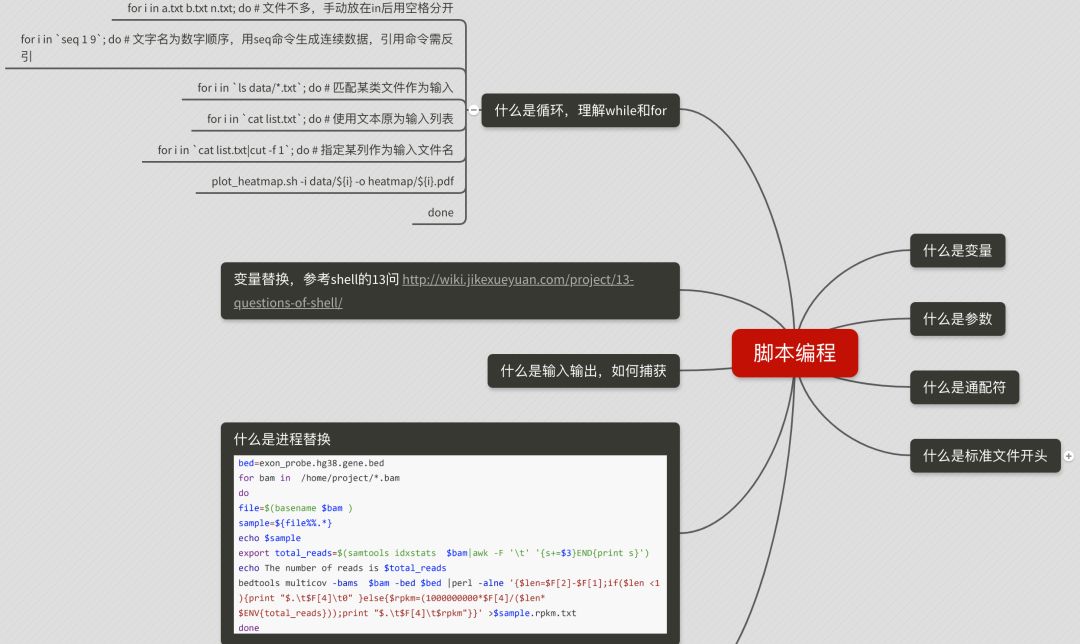
第六阶段:软件安装及conda管理
虽然是,现在有了小白福音,conda来管理大量的生物信息学软件,但大量关于conda使用的疑问都是大家对linux基础知识掌握不够牢固造成的。我还是推荐大家参考 生物信息学常见1000个软件的安装代码! 来安装部分常用软件,至少安装100+软件,力图掌握其中的规律。最后才推荐可以平稳过渡到conda管理软件。
软件安装的规律我总结如下:
二进制可执行程序,解压即可使用
https://www.ncbi.nlm.nih.gov/sra/docs/toolkitsoft/
ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
http://hgdownload.soe.ucsc.edu/admin/exe/linux.x86_64/blat/
系统级别的,演示ubuntu系统的apt功能
编程语言级别
C源代码
java/python/perl/R
conda的下载和安装见conda官网,我们也在生信技能树写过系列推文conda管理生信软件一文就够 , 主要是需要理清楚下面这些概念:
miniconda安装
miniconda配置镜像
创建小环境
查看小环境
进入小环境
查找软件
安装软件
指定软件安装版本
更新软件
查看已安装软件
退出小环境
移除小环境
大部分人的疑问集中在使用conda安装软件结束后,无法得心应手的管理它们,尤其有些需要调py的一些包的时候,会出错,或者python版本不兼容,在使用软件的时候,软件即会出现异常报错。
写在后面
Linux的学习肯定不止我提到的这6个阶段知识点,虽然说大部分人其实连这6点都做的很差,但也有少量生信人员会进阶到服务器管理,运维工作,如果确实条件受限团队无法纳入专职运维,自己也可以在上面知识点基础上面更新自己的思维层次,从更深入的书籍或者教学视频中继续学习,我这里仅仅是列出我工作过程接触到的少量高级知识点:
w/last/top/qsub/condor/apache/socket/IO/ps/who/uid/
磁盘挂载/格式化/重启系统/文件清理/IP查看/网络管理/用户管理/目录结构了解/计划任务/各种库文件
用户组,目录权限管理等等
牢记“不懂的名词,感觉谷歌搜索,多记笔记”。在学习Linux基础知识的同时,就可以开始项目实战,在实战的过程中要随时思考记录如何应用Linux知识辅助生物信息数据处理,并整理学习笔记以及经验分享。
引用:
(公众号推文) linux命令行文本操作一文就够
(公众号推文)linux系统环境变量一文就够
(公众号推文)构建shell脚本一文就够
(公众号推文) conda管理生信软件一文就够
shell中的扩展(Expansions) https://opengers.github.io/linux/linux-shell-brace-parameter-command-pathname-expansion/
bash脚本的参数扩展 (parameter expansion) :https://www.ibm.com/developerworks/cn/linux/l-bash-parameters.html
shell通配符(wildcard): https://cloud.tencent.com/developer/article/1114732
type命令:https://man.linuxde.net/type
字符串操作:https://my.oschina.net/aiguozhe/blog/41557