
本文介绍莱斯计算机科学家托德·特雷根的生物信息学研究小组本月发表在Nature Communication期刊的一篇综述型文章,“Current progress and open challenges for applying deep learning across the biosciences”。文章从蛋白质结构预测、蛋白质功能预测、基因组工程、系统生物学与数据集成、系统发生学等5个方面讨论了生物信息学的研究进展、局限性和前景。对于这5个方面,相对于经典机器学习的改进,文章评估了深度学习在计算生物学中取得了不同程度的进展(图1)。
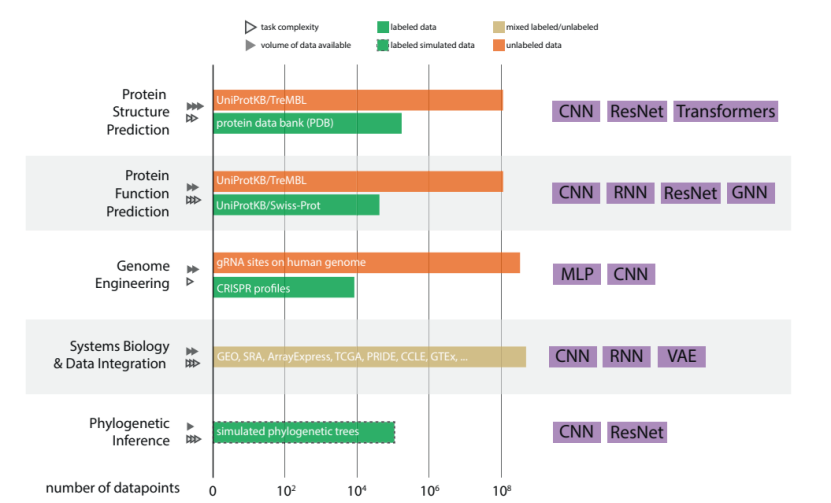
图1.主要标记和非标记数据集的总结观点,以及在计算生物学的深度学习方法中使用的架构。对于本文中考虑的每个领域,它总结了关键数据集和数据库的估计规模,以及这些数据集和数据库的预计增长率。此外,最右边的一栏总结了应用于生物科学相应领域的最流行的深度学习架构。
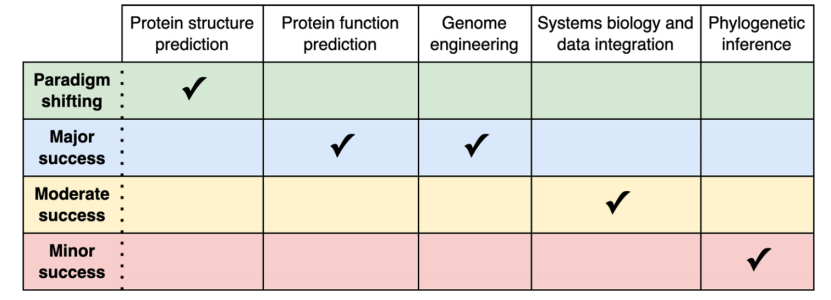
以下将围绕计算生物学的五个广泛而独特的领域展开讨论:蛋白质结构预测、蛋白质功能预测、基因组工程、系统生物学和数据集成,以及系统发育推断(表1)。这些领域从主要范式转换(AlphaFold2)到婴儿时期深度学习应用(系统发育推断)反应了深度学习在计算生物学的影响程度,分别是 (i) 范式转换(深度学习明显优于其他机器学习和经典方法,并占据了一个全领域的影响),(ii)主要成功(深度学习的表现通常高于其他机器学习和经典方法),(iii)中等成功(深度学习的表现通常可与其他机器学习和经典方法相媲美),(iv)次要成功(深度学习方法未被广泛采用或与其他机器学习和经典方法相比表现不佳)。
表1.深度学习对计算生物学的影响
1. 深度学习在不同领域的成功应用
1.1 深度学习的范式转换
蛋白质结构预测 蛋白质结构预测可以说是深度学习在计算生物学中最成功的应用之一。蛋白质的氨基酸序列决定了其三维结构,而三维结构又直接关系到其功能(如化学反应催化、信号传导等)。x射线晶体学是蛋白质结构测定的金标准实验方法,也被认为是验证蛋白质结构预测计算模型的参考。人类基因组计划之后获得了越来越多的生物序列,考虑到x射线晶体学的高成本和技术限制,根据序列预测蛋白质的3D结构成为计算生物学的难题。问题最初集中使用生物物理上精确的能量函数和基于知识的统计推理,但最近由于方法转移到深度学习,取得了更快的进展。深度学习在这一领域取得成功的关键原因之一是以多重序列比对(MSA)形式出现的大量无监督数据,它使学习蛋白质的非线性进化表征成为可能。DeepMind团队设计的AlphaFold和AlphaFold2是将深度学习和MSA联合使用,成功展示了一种基于深度学习的高精度蛋白质结构预测。DeepMind还与欧洲分子生物学实验室(EMBL)合作,创建了一个以AlphaFold2为模型的开放蛋白质结构数据库。该数据库已经覆盖了98.5%的人类蛋白质,其中至少36%的氨基酸残基得到了高可信度的预测。虽然AlphaFold2成功预测了蛋白质的静态结构,但关于蛋白质生物学功能的许多关键见解都来自于其动态构象。此外,多种蛋白质相互作用的动力学在该领域仍然是一个开放的挑战。今后,监测深度学习在这些后续研究领域的应用将是重要的。
1.2 深度学习的主要成功应用
1.2.1 蛋白质功能预测
蛋白质功能预测是继蛋白质结构预测之后的下一步工作。蛋白质功能预测包括将目标蛋白质映射到组织本体,如基因本体术语、生物过程、分子功能和细胞成分。蛋白质结构可以传递关于这些本体的大量信息,然而,两者之间并没有直接的映射关系,而且这种映射关系往往非常复杂。深度学习是解决这个问题最成功的方法之一。第一个基于深度学习的模型DeepGO及其改进版本DeepGOPlus模型表明,将卷积的输出与基于同源性的预测相结合可以获得更好的预测精度。DeepNF使用多模态DAE从多个异构交互网络中提取特征,其性能优于基于矩阵分解和线性回归的方法。总的来说,之前的结果表明,集成来自多模态数据类型(例如,序列、结构、PPI等)的特征的模型更有可能优于那些依赖于单一数据类型的模型。来自文献的趋势表明,依赖于特定任务的架构可以增强数据的特征表示。该方向的未来工作可以集中在基于序列的表示的结合DAEs和RNN模型,以及基于结构和PPI的信息表示的图卷积网络。将这些表示结合到层次分类器中,如多任务DNN和生物相关的正则化方法中可以为蛋白质功能预测提供一个可解释的、计算上可行的深度学习结构。1.2.2 基因组工程
生物医学工程,特别是基因组工程,是生物学的一个重要领域,应用深度学习模型解决领域内问题是当下热点。应用深度学习技术CRISPR是原核生物基因组中发现的一类DNA序列,近年来被用于识别和切割人类基因组中的特定位置。当20个核苷酸的gRNA序列与基因组互补时,Cas9在靶基因上产生双链断裂(DSB)。但是,Cas9可以切断基因组上不需要的位置(脱靶事件),这对细胞的功能和完整性有致病性的影响。因此,由于Cas9在治疗方面的效率、可靠性和可控性不足,Cas9的全面临床部署一直缓慢。因此,减少脱靶同时提高靶内效率已成为利用深度学习技术实现基因组工程目标的重要最终目标。事实上,深度在预测人类细胞中腺嘌呤碱基编辑器(ABEs)和胞嘧啶碱基编辑器(CBEs)的效率以及主编辑器2 (PE2)的活性方面已经显示出前景。未来的挑战在于理解这些模型,CRISPRLand是一个最新的框架,已经向深度模型的高阶交互解释和可视化迈出了第一步。除了可解释性,文章推测,能够对预测结果进行不确定性估计的方法在基因组编辑中变得更加普遍。此外,由于显著的细胞类型效应对CRISPR实验的效率,在基因组工程中部署深度学习模型时,意识到分布变化是至关重要的。集成领域自适应方法来限制这种分布偏移的影响是未来的重要方向之一。
1.3 深度学习的中等成功应用
系统生物学和数据集成 系统生物学以整体的观点建模复杂的生物过程,最终解开基因型和表现型之间的联系。多种组学数据的整合能够建立稳健的预测模型,已在基础生物学和精准医学领域取得多项突破。
数据集成任务主要分为两大类:1)跨不同平台和单一数据类型的集成;2)不同组学数据类型之间的集成。在数据整合方面,传统的机器学习通常是使用自动特征提取技术的无监督学习。近来,自动编码器已被用作初始数据处理步骤,以生成低维嵌入,然后连接在一起作为下游模型的特征。也许集成多模态数据最直接的方法是训练单个数据模态模型,然后通过组合来自单个模型的结果来集成它们,这称为基于模型的集成。在某种程度上,这类似于经典机器学习中经常使用的集成方法。经典方法试图通过找到一个公共的建模空间来连接数据模式,深度学习可以识别公共的空间并联合建模,从而避免了单独建模和集成步骤的需要。虽然很明显,深度神经网络可能会在数据集成任务中带来更好的性能,但记住深度神经网络的局限性也很重要,这也是继续研究的重要领域。另一方面,在数据集成任务中,批处理效应可能很普遍,而且经常很容易在训练集和测试集之间产生偏差,所有这些都可能导致夸大的性能估计。这些偏差可能会导致虚假的关联,这种关联困扰着所有的机器学习方法,也可能包括深度学习。
1.4 深度学习的次要成功应用
系统发生学 系统发生学是一种进化树,模拟了一组分类单元的进化历史。系统发育推断问题涉及从所研究的类群中获得的数据(通常是分子序列)建立系统发育。图3说明了四个类群的系统发育推理问题;在这种情况下,它可以被视为三种可能的拓扑之间的分类问题。然而,分类方法有一个主要的限制,即它们不能推断分支长度,也不能扩展到非常少的分类单元,因为可能的拓扑(类)的数量随着这个变量以超指数增长。但也许更重要的是,像深度学习模型这样的分类器需要训练数据,而在这个领域中几乎不可能获得已知真正系统发育的基准数据。对于复杂版本的系统发育推理问题,需要更真实的仿真协议。最后,从某种意义上说,对单个基因的系统发育推理本身就是一个简化的问题:从全基因组数据推断单个系统发育引入了复杂性,即不同的基因可能有不同的历史,或者真正的系统发育可能是一个网络,而不是一棵树。由于这些原因,深度学习要么成功有限,要么局限于主要推理任务之外的小的子问题。

图2.系统发育推断的标准和DL方法
尽管如此,还是有人尝试将深度学习用于上述的分类任务。基于距离的方法是一类常用的系统发育推理技术,其中邻居连接方法是最常用的一种,而深度学习的CNN模型则被用于改进距离表示,然而,这些方法的关键局限性是树不能可靠地嵌入到低维的欧几里德空间中。另一方面,双曲空间已被证明更适合表示具有层次潜在结构的数据。深度学习可以用来验证系统发育推断,以及指导下游分析,如假设生成和检验。总的来说,一些相关的成功是令人印象深刻的,但考虑到一系列的局限性,很难构想一个端到端深度学习模型直接建立原始数据的系统发育树。而且,如果要开发出一种基于(可能是模拟的)训练数据的方法,那么在传统的系统发育方法被取代之前,需要仔细验证其对实际生物序列的适用性。
2.深度学习在生物科学领域面临的普遍挑战
并非所有深度学习的应用在计算生物学上都同样成功。而在一些领域,如蛋白质结构预测和基因组编辑深度学习取得了很大成就,在其他领域如系统发育推断,深度学习已经面临重大障碍(表1)。目前两个需要改进的关键领域:(i)可解释性和(ii)训练效率。
可解释性,是目前最关键的局限性之一。开发dnn解释工具仍处于初级阶段,并正在迅速成长;在生物学中,要想建立一个完全可解释的系统,仍然面临许多挑战。关键问题是,目前解释深度学习模型的通用方法不够充分。为了让科学家和临床医生相信这些黑盒子模型,他们需要能够以一种人类可以理解的方式,可量化地解释、总结的原因,并增加额外的步骤(例如,实验、临床研究等等)加以证明结论。文章推测,新一代可解释的方法专注于帮助这些黑盒模型从假设生成机器过渡到假设测试机器,从而更容易与医疗从业者沟通。
训练效率,降低深度学习方法训练成本最直接的方法是对现有的预训练通用模型进行迁移学习,而不是从头开始训练新模型。这是自然语言处理任务训练模型的一种常用方法,而且已有研究表明,通用语言知识模型对于各种不同的自然语言处理任务来说都是一个很好的起点。如果所有的下游任务都可以从生物数据的通用模型开始,那么这种方法就可以在计算生物学中采用。另一种方法是设计效率更高的深度学习模型体系结构。利用先前的领域专家知识来帮助修剪注意力区域;在训练过程中改变模型的结构,以自适应地提高训练效率;模型的剪枝,从模型中删除不重要的参数,部署轻量级深度学习模型。
3.结论
总而言之,深度学习在蛋白质结构预测等领域的成功是范式的转变,而在功能预测、基因组工程和多组学等其他领域,与传统方法相比,也在性能上取得了快速进展。对于其他领域,如系统发育学,经典计算方法似乎在这些领域占了上风。在生物科学挑战中应用深度学习在其他领域取得进一步进展的同时也要努力增强可解释性和提高效率。
参考资料
Sapoval, N., Aghazadeh, A., Nute, M.G. et al. Current progress and open challenges for applying deep learning across the biosciences. Nat Commun 13, 1728 (2022).
DOI: https://doi.org/10.1038/s41467-022-29268-7