本文将介绍大连理工大学都健教授团队近期在AIChE期刊上发表的论文“De novo drug design framework based on mathematical programming method and deep learning model”,其团队开发了一个深度学习模型用于识别具有高结合亲和力的靶标-配体复合物,并将其与数学规划模型进行集成,实现了高通量反向优化设计小分子药物目标,进一步以治疗心血管疾病的利伐沙班替代药设计和治疗肿瘤疾病的阿昔替尼替代药设计为例,证明了本文所提出的从头药物设计框架的通用性和有效性。
通常,从头药物设计方法包括遗传算法与深度生成方法。然而,这两种方法均容易陷入局部最优解。数学规划方法是系统工程领域常用的一种数学优化方法,其也可用于分子设计问题。该方法通过建立由目标函数、分子结构约束和分子性质约束组成的混合整数非线性规划 (MINLP) 模型并进行有效求解,可实现高通量反向优化设计目标性质最优的分子结构。具体而言,MINLP模型能够通过利用显式的数学公式(如八隅体规则、价键规则等)与基于梯度的确定性优化算法 (如BARON算法) 来优化组合基团,该模型无需遍历评估所有基团组合的情况,即可快速获得满足结构和性质约束的所有可行解并确定具有最大或最小目标性质的最优解。然而,当MINLP模型中的非线性方程过于复杂时(即模型非凸性较强时),直接求解MINLP模型是非常困难的。为了解决这个问题,有学者提出一种分解式求解算法来求解非凸性极强的MINLP模型。理论上,当MINLP模型性质约束相互独立时,通过使用分解式求解算法顺利求解MINLP模型,可在MINLP创建的化学空间范围内找到全局最优解。
尽管数学规划方法在设计小分子溶剂上已取得了巨大的成功,但其在应用于候选药物设计时仍存在两个挑战。首先,药物的结构(尤其是环结构)相比于小分子溶剂更大且更复杂,因此,需要更多数量的环基团来建立MINLP模型以用于候选药物设计,这将增加MINLP模型的问题规模和求解难度。即使MINLP模型成功求解,第二个挑战是传统MINLP模型容易产生一些结构上可行但反常的环结构。例如,如果为MINLP模型选择了环状基团“aC-C#CH”,则将设计类似于“CC(C)c1c(C#C)c(C#C)c(C#C)c(C#C)c1C#C”的分子。
为了解决上述两个挑战,本文使用Bemis–Murcko算法批量提取DrugBank数据库中药物分子的骨架结构,共得到2,898个药物骨架,并将骨架引入传统MINLP模型,以确保设计的候选药物结构的合理性。然而,如果使用所有2,898个药物骨架来设计候选药物,MINLP模型的问题规模仍然较大。考虑到具有相似骨架的候选药物可能具有相似的特性,因此在建立MINLP模型之前,本文使用基于骨架的相似性算法来识别与目标上市药物骨架相似的骨架子集,从而大幅减少MINLP模型的问题规模。
此外,本文也建立了一个预测结合亲和力的深度学习模型作为MINLP模型的目标函数,使MINLP模型具备设计结合亲和力最优的小分子候选药物的能力。
本文首先构建了一个可预测高结合亲和力概率的深度学习分类模型,模型的输入是配体的SMILES文本与靶标的氨基酸序列,模型的输出是高/低结合亲和力的概率大小,如图1所示。
本文将配体SMILES转化为Mol2vec描述符来表示配体分子的结构特征,并将配体特征矩阵送至基于门增强的注意力层进行特征提取。同理,将靶标氨基酸序列转化为高阶氨基酸序列来表示靶标的结构特征,并将靶标特征矩阵送至卷积神经网络进行关键氨基酸序列的识别,进一步将降维后的氨基酸序列送至基于门增强的注意力层进行特征提取。随后,将配体与靶标矩阵进行拼接,送至全连接层来预测高/低结合亲和力的概率。
随后,本文提出了一个基于优化的从头药物设计框架,该框架集成了预测结合亲和力的深度学习模型与MINLP模型,如图2所示。
该框架构建步骤包括:
(a)建立药物数据库。
(b)通过使用RDKit中的Bemis–Murcko算法从DrugBank药物数据库中提取药物骨架。在建立MINLP模型之前,针对目标药物的骨架结构,使用基于骨架的相似性算法从骨架数据库中搜索与目标药物骨架相似的骨架子集G1,同时选定一组常用的基团子集G2,G1和G2作为MINLP模型的输入。
(c)建立由目标函数、药物结构约束、药物性质约束组成的MINLP模型。MINLP模型细节如下:
目标函数:
约束条件:
深度学习约束:通用方程(1)表示用于识别具有高结合亲和力的靶标-配体复合物的深度学习模型。
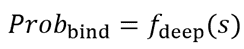
药物结构约束:通用方程(2)表示八隅体规则m1、价键规则m2和化学复杂性m3的结构约束,通过骨架和基团的组合能够生成结构合理的分子。

药物性质约束:通用方程(3)与(4)表示“里宾斯基五规则”性质(相对分子质量MW、氢键受体的数量HBA、氢键供体的数量HBD、辛醇-水分配系数logP、可旋转角数ROT(ROTfrag)),以及合成可行性分数SA和合成复杂性分数SC。
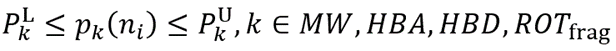
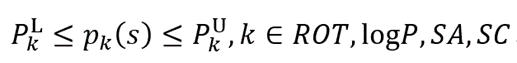
其它约束:通用方程(5)表示改进的基于SMILES的异构体生成算法,该算法用于将候选药物的骨架-基团向量自动转换为对应的药物SMILES字符串。
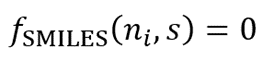

(d)采用分解式求解算法求解MINLP模型。如果没有符合所有约束的最佳候选药物,则返回(c)松弛约束范围并重新求解MINLP模型。
(e)MINLP模型的最优解通过分子对接和分子动力学模拟进行进一步验证。
训练集/验证集/测试集的损失函数(CEL)和监督函数(AUC)分别为0.369/0.442/0.401和0.914/0.880/0.901。
深度学习模型的训练过程与分类性能如图3所示。图3(a)和图3(b)分别记录了深度学习模型训练集和验证集的CEL和AUC随Epochs的变化情况。图3(c)表示基于二元分类的混淆矩阵,用于评估深度学习模型的分类性能,图中测试集的真阴性和真阳性样本数远大于假阴性和假阳性样本数,表明深度学习模型能够较好区分高/低结合亲和力。图3(d)给出了测试集的ROC曲线(实线),测试集AUC=0.901表明深度学习模型具有良好的分类性能。
进一步,通过集成深度学习模型与MINLP模型,实现了从头药物设计目标。以治疗心血管疾病的利伐沙班替代药设计为例。首先,使用基于骨架的相似性算法,从包含2,898个药物骨架的骨架数据库中寻找与利伐沙班骨架相似的药物骨架子集(G1),结果共获得14个骨架。随后,这14个骨架搭配一组基团子集(G2)输入MINLP模型,并设定MINLP模型的目标函数、结构约束上下限和性质约束上下限。然后,使用分解式求解策略对MINLP模型进行求解。在子问题1中,通过数学规划方法,在结构和线性性质的约束下,高通量反向设计得到N1=17,659个可行解(由骨架-基团向量表示),该过程在台式机上耗时116秒(Intel(R) Core (TM)i7-10700F CPU @ 2.90GHz 24.0 GB RAM)。
在子问题2~3中,利用改进的基于SMILES的异构体生成算法(其它约束),基于N1个骨架-基团向量生成N2=159,170个候选药物SMILES字符串(2,161秒),并利用非线性性质约束,剔除不满足性质约束的候选药物,剩余N3=42,932个候选药物SMILES用于进一步分析。首先,将本文设计得到的42,932个化合物SMILES在PubChem数据库中搜索,发现有2,261个(5.25%)设计得到的结构存在于PubChem中,这表明基于MINLP的药物设计模型不仅能够找到现有的候选药物,还能够设计全新的候选药物(94.75%)。其次,使用ECFP指纹和主成分分析方法(PCA)创建42,932个设计候选药物的化学空间图,如图4所示。右侧图例中的整数(0~7)代表8个骨架,点“4”代表利伐沙班。图4展示了设计的候选药物在化学空间中的广泛分布,表明基于MINLP的药物设计模型在设计与利伐沙班相似的结构多样的候选药物方面具有强大的潜力。
在子问题4中,使用深度学习模型预测设计得到的42,932种候选药物的高结合亲和力的概率,并按降序排列。排名结果表明,有四个设计得到的候选药物SMILES在高结合亲和力的概率方面优于利伐沙班(98.76%)。与现有药物(利伐沙班:SC=4.7152)相比,最佳设计的候选药物不仅具有高结合亲和力,而且具有较低合成复杂性(SC=3.1661)。最后,选择具有98.78%高结合亲和力概率的最佳设计候选药物进行分子对接(台式机(Intel Core i5-10500 CPU @ 3.10 GHz)上为414秒)和分子动力学模拟(Advanced超级计算中心(AMD EPYC 7502 CPU @ 2.5GHz 64cores)上约8小时)来验证本文开发的深度学习模型的可靠性和MINLP模型在候选药物设计中的有效性。
在分子对接中,靶标为Xa因子(PDB条目2w26),配体为MINLP模型最优设计结果,对接得到的结合能为∆Gbind=-42.39 kJ/mol。通过∆Gbind=RTlnKi/d公式计算结合亲和力Ki/d=0.037 μmol/L(<1 μmol/L代表高结合亲和力),这表明设计得到的候选药物与Xa因子具有高结合亲和力。此外,本文通过分子对接方法评估了设计得到的高结合亲和力概率排名最差的(高结合亲和力概率为25.82%)候选药物,得到∆Gbind=-29.97 kJ/mol和Ki/d=5.61 μmol/L(≥1μmol/L表示低结合亲和力)。这个结果表明本文的深度学习模型能够可靠地区分靶标-配体复合物之间是具有高结合亲和力还是低结合亲和力。分子动力学模拟结果如图5和图6所示。
综上,本文提出了一个基于优化的从头药物设计框架,该框架首先开发了一个深度学习模型预测靶标-配体复合物的高/低结合亲和力概率,并将其集成于数学规划模型,实现了高通量反向优化设计结合亲和力最优且满足“里宾斯基五规则”、合成可行性分数与合成复杂性分数性质要求的全新候选药物,最后对最优设计结果进行了分子对接和分子动力学验证。
Yujing Zhao, Qilei Liu*, Xinyuan Wu, Lei Zhang, Jian Du*, Qingwei Meng. De novo drug design framework based on mathematical programming method and deep learning model. AIChE Journal. 2022, e17748. https://doi.org/10.1002/aic.17748