——介绍——
目前的分子动力学模拟技术(MD)仍难以满足研究人员对重大微观事件深入探究的要求。即使发展了一系列的增强采样方法,但似乎仍难以解决MD的时间尺度限制问题,甚至可能导致不可避免的精度折损问题。来自西湖大学的李子青团队发展了基于分数的模型ScoreMD来尝试利用深度学习模型,在保持精度的同时,从技术层面突破目前MD的时间尺度瓶颈。
——背景——
基于深度学习的MD(DLMD)为揭示大分子体系重要微观事件提供了新的思路。这类模型之所以受欢迎,来源于其准确性可由基于物理偏执约束以及神经网络拟合大数据的能力所保证,以及当前AI硬件的并行加速。尽管这类模型取得了一定的成功,但是目前的DLMD模型主要有以下三个问题: 1. 大多数DLMD模型依赖于中间变量,并以多阶段方式生成生物分子构象。这大大增加了计算开销,而且阻碍了推理的效率,因为逆Hessian标度与原子数呈三次方复杂度; 2. 现有的DLMD是预测原子属性的黑箱模型,大多数是针对静态结构设计的,而不适用于动态体系。如何预测速度,加速度和力等物理量,使得模型显式地满足牛顿运动方程还需要不断的摸索。
——方法——
大多数的DLMD模型依赖于势能,以获取原子的作用力和位置的更新,因此需要额外的反向传播计算,显著增加了计算开销。即使最近有模型抛弃这类双阶段范式,直接预测原子作用力,但还是不可避免地使用了额外的积分器来更新原子的位置。而该工作试图开发马尔可夫状态模型,利用前一帧直接预测微观体系的三维坐标,无需额外的积分器。
1.Score-based model
ScoreMD的架构如下图1所示。作者使用去噪分数匹配技术(Denoising score matching, DSM)训练分数模型。DSM (或者称扩散模型) 算法(图2)详情可以参考[2]快速了解,在此不进行赘述。作者认为,从直觉上而言,去噪扩散模型与目前的增强采样方法存在相似性。模型嵌入时间信息后,在有限的时间步数内迭代地加入先验噪音,进行前向扩散,从而缓慢地破坏分布中的结构。而在逆向去噪阶段,模型需要尽可能地最小化体系总能量。
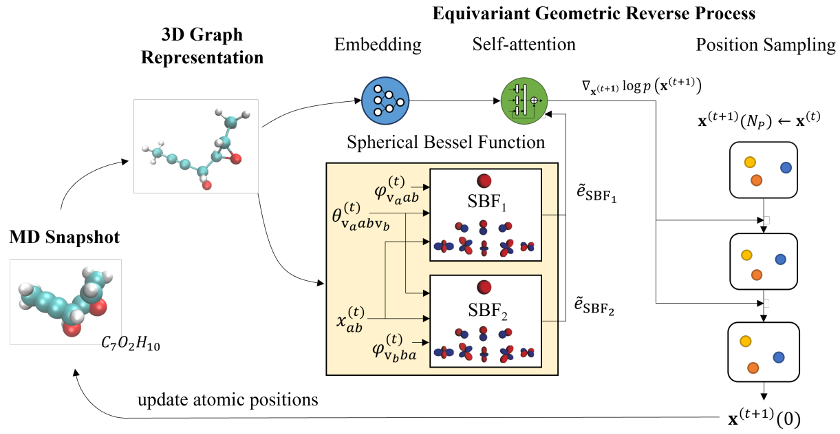
图1. ScoreMD的总体架构
作者意识到直接将图像生成领域的扩散模型应用到MD中,直接做深度学习技术应用的扩展是不合适的。显然,对于MD轨迹生成这类问题,我们期待扩散模型采样的最终轨迹至少在结构上是合理的,并且是依赖于前一帧轨迹的。ScoreMD采取的方案是在扩散时,噪声规划方案不是标准的,而是自适应地依赖于前一帧的加速度来获得当前帧的噪声强度 (Conditional noise),然后按照规划进行扩散,从而能够采样得到下一帧的原子位置更新,注入MD轨迹前后帧RMSD相差不大的偏执,保证一定的结构相关性。因此,简单来说,ScoreMD是,在基于分数的模型的基础上,使用前一帧轨迹加速度作为先验的条件生成模型。具体到每一帧,还是需要按照一般的扩散模型去做采样。
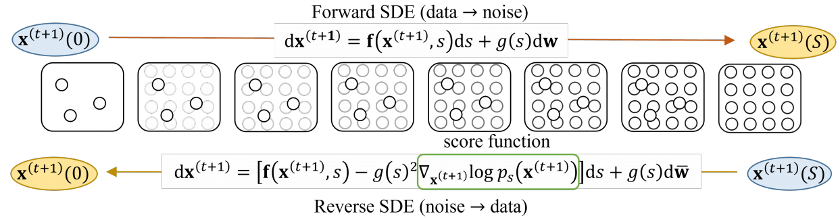
图2. 前向和逆向扩散示意图
与原始的DSM
模型的噪音规划方案相比,ScoreMD下一帧的原子位置更新的SDE方程参考了GaMD的boost potential,修订的方程如下所示:其中是谐加速度常数,表示加速度阈值。一旦系统具有缓慢的运动变化趋势(即系统能量较低),系统将被提供大量噪声。因此,噪音与前一帧的加速度成反比,继承了增强采样的优点。
由此,由于每一帧的目标扩散分布并不是标准的高斯分布,SDE的transition kernel需要重现表述为如下公式2:2.Equivariant geometric transformer (EGT)
作者还提出了EGT架构,在保持E(3)-equivariance的同时,利用任意两个成键原子的速度与共价键形成的两个夹角和二面角来考虑成对原子的相互作用以及未来的位置。另外,为了整合所有的几何信息,在网络输入的初始阶段会计算spherical Fourier-Bessel bases,并整合到特征当中,具体的计算公式请见文章。
——模型比较——
为了检验模型的性能,作者在两个任务上进行了评价: 1) 短程到长程 (Short-term-to-long-term, S2L) 轨迹生成。在这个任务中模型以每一个的分子的短程轨迹作为训练集,期望模型能够在长时间尺度下生成的轨迹的多样性更高; 2) 一到多 (One-to-others, O2O) 轨迹生成。在这个任务中,以部分分子的轨迹作为训练集,期望模型能够针对训练集未出现的分子产生合理的轨迹。两个任务的评价指标均为累积均方根误差 (ARMSE),仅衡量生成的轨迹的构象变异程度。
表1. 在MD17数据集上以ARMSE为指标的评价结果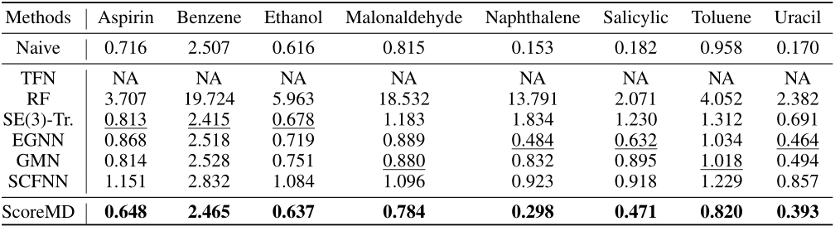
表2. 在MD17数据集上以ARMSE为指标的评价结果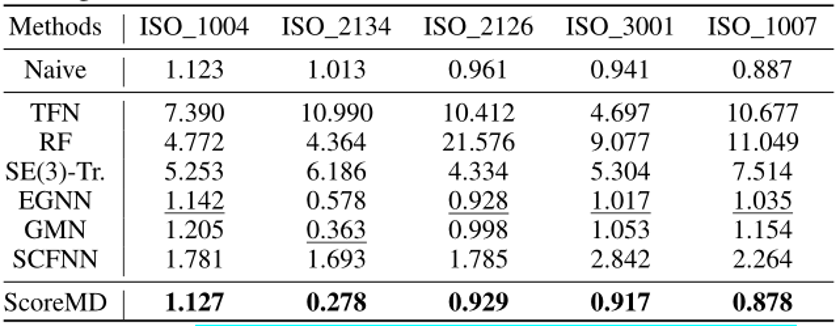
从上述的两个任务以及相应的数据集上不难发现,ScoreMD达到了SOTA的水平。
——总结
——
在这篇工作中,作者提出ScoreMD模型,将扩散模型与几何等变网络相结合,并利用原子间距离之外的其他矢量信息来加入物理偏执。在不同数据集上对S2L和O2O任务进行的大量实验表明,ScoreMD是目前的SOTA模型。这项研究可能有助于加速新药和材料的发现。当然,这篇工作目前只挂在arxiv上,可能仍有一些工作未完成:
1.与传统的MD相比,采样速度在这篇文章中未得到评价。扩散模型要求前向的迭代步数一般在1000以上,将扩散模型直接应用到MD中以突破时间尺度限制甚至可能会导致此类DLMD模型比传统MD的速度还慢。尽管目前扩散模型采样速度低的不足已经得到关注及解决,但试想一下,用目前最好的逆向扩散采样方法 (粗略的按照10步估计),如果我们希望采样1000条轨迹,那么我们需要花额外的9000步模型计算量 (如果是按照1000-2000步,那采样效率可能更低),更何况目前的采样方法在结构生成上的表现仍是未知的;
2.ARMSE只意味着采样的轨迹的构象差异,但是作者并未评价结构的合理性。去噪过程中只有最后一帧可能是有用的,而中间轨迹可能几乎是不可利用的。如何保证去噪过程的每一个结构都是潜在有用的,或至少保证结构是合理的,是扩散模型应用到这个领域的一个核心问题;
3.相较于其他的模型,也许ScoreMD真的实现了让分子"动起来",但是作者并未进一步分析构象的势能面,并未真正回答到背景部分试图说明的模型意义,ScoreMD的采样效率还有待探究;
即使在本篇文章中作者仅将模型应用到小分子数据集上,但笔者认为ScoreMD也为应用在生物大分子体系的DLMD模型提供了新的思路,特别是基于深度学习做增强采样问题。
参考文献:
1.Fang Wu, et al. A score-based geometric model for molecular dynamics simulations. arXiv:2204.08672.
2.https://yang-song.net/blog/2021/score/
点击左下角的"阅读原文"即可查看原文章。
作者:幻 幻
审稿:顾仲晖
编辑:黄志贤
GoDesign
ID:Molecular_Design_Lab
( 扫描下方二维码可以订阅哦!)

本文为GoDesign原创编译,如需转载,请在公众号后台留言。