可重复性研究一直是学术发表希望实现的方式,更有助于判断研究结果的准确性和帮助后人以此为基础进行深入研究。计算分析类型的研究,是能够比较容易实现可重复性的 - 只要分享代码和数据就可以 (当然配置运行环境也需要一定的功底),如使用R markdown或Bookdown 或者 Jupyter notebook等,像宏基因组主编刘永鑫老师的植物根系菌群分析的NBT文章那样,把代码存放在Github上面也是一种比较好的方式。
CodeOcean是Nature推荐过的一个可重现性代码共享平台。其实质类似于一个云平台,可以直接在上面配置环境、开展项目分析,合作者之间协作并存放数据和代码,发表时提供给杂志社或读者,实现从原始数据到文章图表的整个可重复过程,并且可以进行衍生分析,或者上传自己的文件数据测试。而且有部分杂志要求作者发表时要把自己的代码和数据上传到CodeOcean上面。

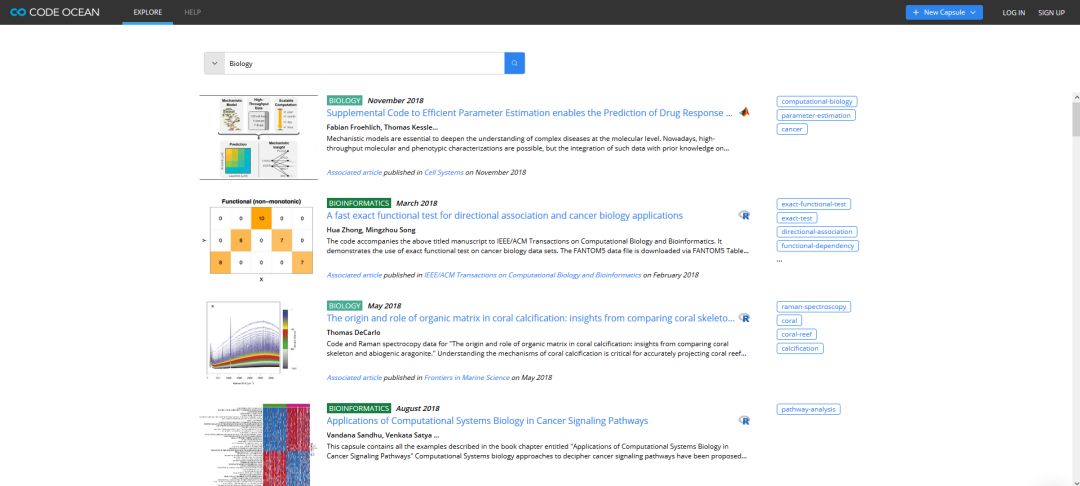
我们以2019年9月份发表在Nature microbiology上的一篇文章Life cycle progression and sexual development of the apicomplexan parasite Cryptosporidium parvum为例,查看下其代码的可重用性使用方式。作者在CodeOcean上面提供了其从原始数据到基因定量、差异分析、火山图、热图、功能注释用到的Bash和R的代码,只需要在右侧点一下Reproducible Run按钮就可以重现分析。(Note: 从Rmarkdown中可以看到Bash相关代码默认没有执行,因为没有存放原始数据,可以自己从NCBI的SRA数据库下载,修改eval=T再继续分析)
具体可见下面的图和录制的视频。
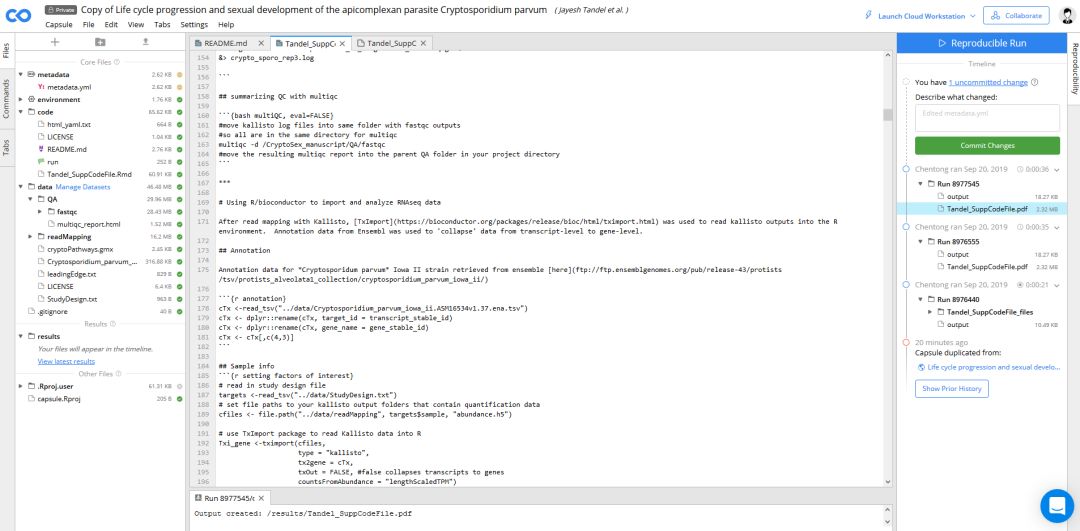
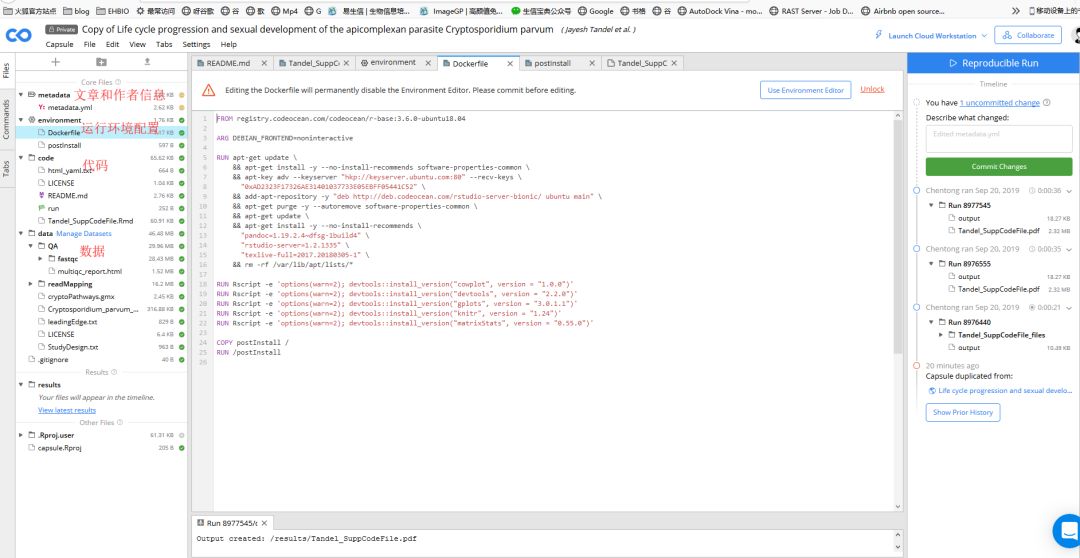
从视频中可以看到,CodeOccean不是一个简单的代码存放平台,是可以根据自己的需求配置环境、安装软件、R/Python包、存储数据和运行程序的平台,相当于一台共享的云服务器。所有的配置信息也都在对应的文件中有记录,如果我们想用于分析自己的数据,就在自己服务器配置对应的环境、下载代码和数据进行运行即可。
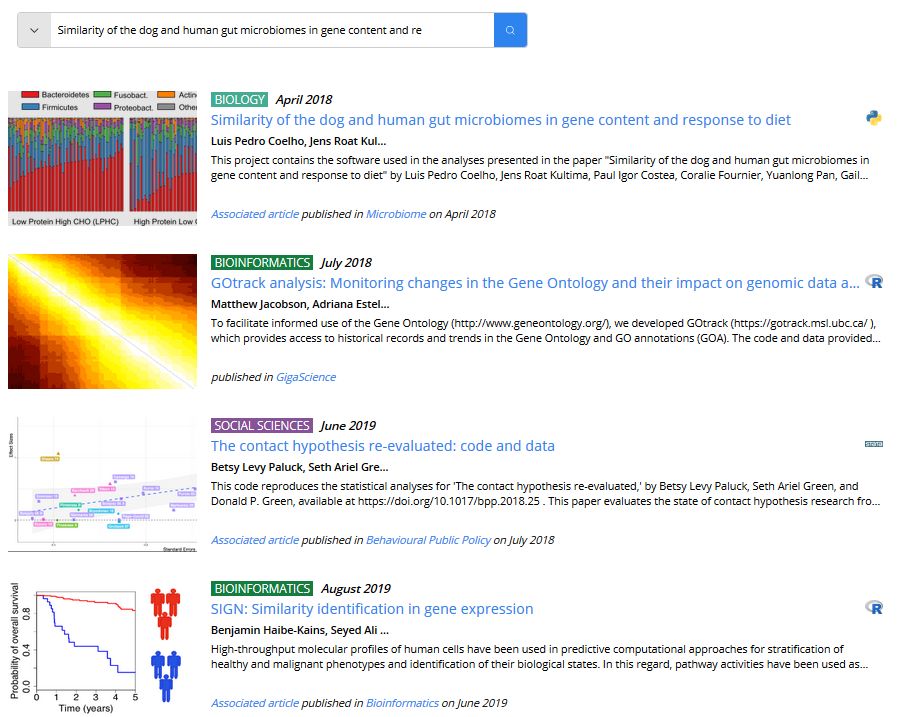
浏览过程中,发现一篇Microbiome上发表的人和狗肠道菌群的比较文章,作者提供了全部分析的Python代码,脚本很多,是学习宏基因组分析和Python的好文档。
还有2019年发表在eLife上的一篇单细胞分析的文章,Identifying Gene Expression Programs of Cell-type Identity and Cellular Activity with Single-Cell RNA-Seq,用Jupyter notebook的形式列出了各个分析部分的所有代码,还贴心的提供了目录。

更多文章和代码等着你去探索了。
https://codeocean.com/
实用网站文献
更多阅读
画图三字经 生信视频 生信系列教程
心得体会 TCGA数据库 Linux Python
高通量分析 免费在线画图 测序历史 超级增强子
生信学习视频 PPT EXCEL 文章写作 ggplot2
海哥组学 可视化套路 基因组浏览器
色彩搭配 图形排版 互作网络
自学生信 2019影响因子 GSEA 单细胞
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集

