
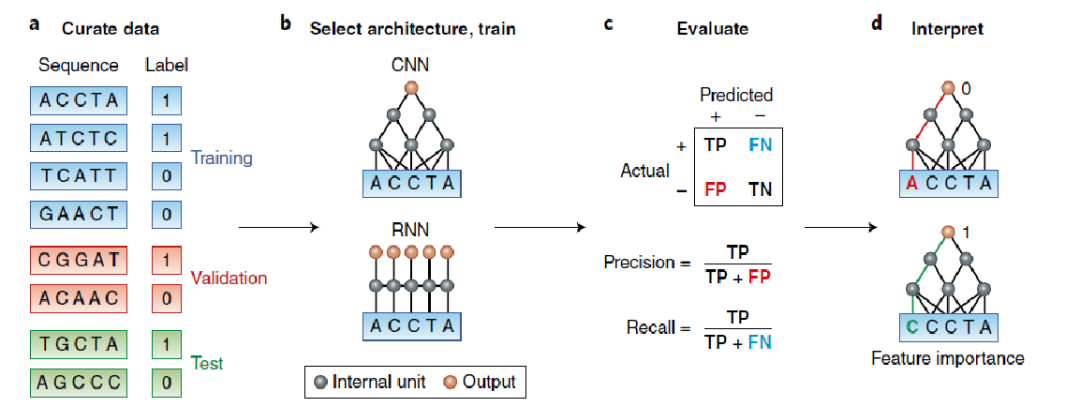
如何用深度学习研究组学?一、什么是深度学习?1、主要策略2、数据集的划分3、如何保证深度学习高效?1、合适的训练集2、合理的评估标准3、对应学科的知识4、在大多数基因组学应用中,少于五层就足够了二、常见网络1、全连接层 (DNN)2、卷积神经网络 (CNN)3、循环神经网络 (RNN)4、图卷积神经网络 (GCN)5、自编码器 (AE)三、基因组深度学习案例三、深度资源1、常用的框架2、课程资源参考文献
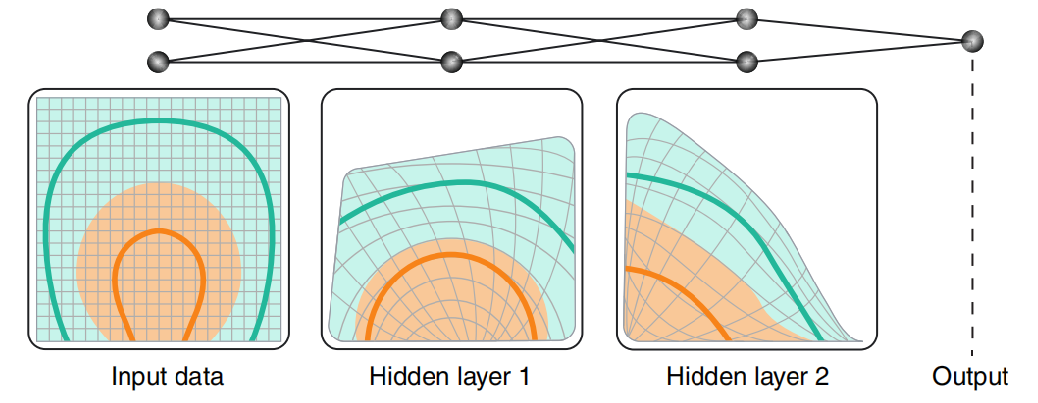
深度学习是表示学习的一种。
上图能够看到数据经过不同的隐藏层,数据的表示形式不断的改变,直到线性可分或者变成具体的可能性。
监督学习:预测样本的标签
无监督学习:学习数据固有模式(转化数据)
例如样本平衡,这就像有99个女人和1个男人,你只要预测样本是女生,正确率就是99%,但你无法预测男人。
例如,不平衡的数据集使用召回率(Recall)更为可信
例如,调控motif一般小于20nt,说明卷积过滤核也应该小于20。还有一般增强子位置一般在几百到两千的位置,那么序列的输入应该不大于两千。
在大多数基因组学应用中,少于五层就足够了
即使在5层网络中,也包含上百万的参数,关键在于数据集是否足够和有效(一般上千个样本效果会很好)
注意
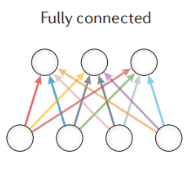
最常见的就是全连接层,这个是最常用的神经网络,处理一般生物学问题几乎都用它,例如PWM matrix(位置权重矩阵)和k-mer等。
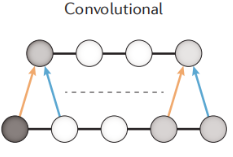
在图片识别中,CNN在语音识别,图像识别,图像分割,自然语言处理等领域取得了巨大的成功,已经有很多卷积深度学习应用于分析生物学数据,例如DeepCpG、 DeepBind、DeepSEA、Basset等,它适用于带有固定位置的数据,例如DNA序列,氨基酸序列和图片。
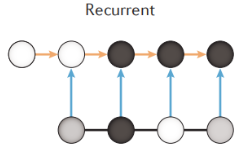
自然语言处理(NLP)是计算机科学、语言学和机器学习的交叉点,它关注计算机与人类之间使用自然语言中的沟通交流。DNA序列也是一种语言,但是其中奥秘尚未可知。它适用于DNA序列,氨基酸序列和时序性数据。
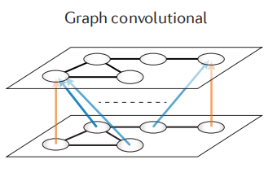
图卷积神经网络,顾名思义就是在图上使用卷积运算,目前在基因组上应用较少,它适用用于蛋白互作网络、蛋白质结构等。
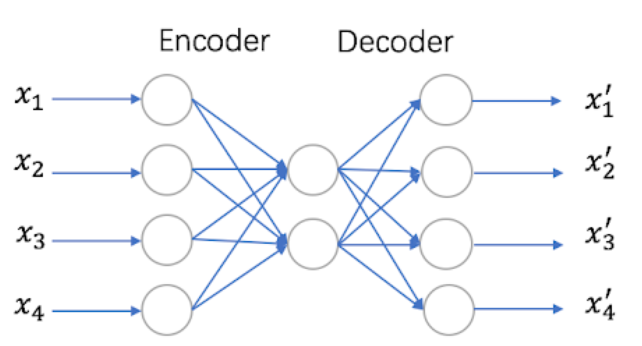
自编码器是一种利用反向传播算法使得输出值等于输入值的神经网络,它先将输入压缩成潜在空间表征,然后通过这种表征来重构输出。
 将DNA序列转换为"图片"
将DNA序列转换为"图片"
# 方法源于https://colab.research.google.com/drive/17E4h5aAOioh5DiTo7MZg4hpL6Z_0FyWr#scrollTo=IA9FJeQkr1Ze
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import requests
from sklearn.preprocessing import LabelEncoder, OneHotEncoder
# 数据集
# 特征集地址
SEQUENCES_URL = 'https://raw.githubusercontent.com/abidlabs/deep-learning-genomics-primer/master/sequences.txt'
# 标签集地址
LABELS_URL = 'https://raw.githubusercontent.com/abidlabs/deep-learning-genomics-primer/master/labels.txt'
# 下载数据集
sequences = requests.get(SEQUENCES_URL).text.split('\n')
sequences = list(filter(None, sequences)) # This removes empty sequences.
labels = requests.get(LABELS_URL).text.split('\n')
labels = list(filter
(None, labels)) # removes empty sequences
# one-hot编码
one_hot_encoder = OneHotEncoder(categories='auto')
labels = np.array(labels).reshape(-1, 1)
input_labels = one_hot_encoder.fit_transform(labels).toarray()
'''
DNA Sequence #1:
CCGAGGGCTA ... CGCGGACACC
One hot encoding of Sequence #1:
[[0. 0. 0. ... 1. 0. 0.]
[1. 1. 0. ... 0. 1. 1.]
[0. 0. 1. ... 0. 0. 0.]
[0. 0. 0. ... 0. 0. 0.]]
'''
# 这个转换很有意思, 行变成了C:0100,成功的把序列数据集转换成了“图片”!!!
fast.ai: http://www.fast.ai/
Coursera: https://www.coursera.org/specializations/deep-learning/
Textbook:
http://neuralnetworksanddeeplearning.com/
参考文献
Deep learning: new computational modelling techniques for genomics
A primer on deep learning in genomics
A guide to deep learning in healthcare