进化树以树状结构形象的展示各个节点的进化关系,在物种进化,亲缘关系研究领域广泛应用。在biopython中,通过Bio.Phylo子模块,可以方便的访问和展示树状结构中的信息1. 读取文件
树状结构最常见的文件格式是newick, 读取方法如下>>> from Bio import Phylo
>>> tree = Phylo.read('tree.newick', 'newick')
>>> tree
Tree(rooted=False, weight=1.0)
2. 查看树状结构
print方法是最简单的查看树状结构的方法,示例如下
>>> print(tree)
Tree(rooted=False, weight=1.0)
Clade()
Clade()
Clade()
Clade(name='A')
Clade(name='B')
Clade()
Clade(name='C')
Clade(name='D')
Clade()
Clade(name='E')
Clade(name='F')
Clade(name='G')
3. 可视化
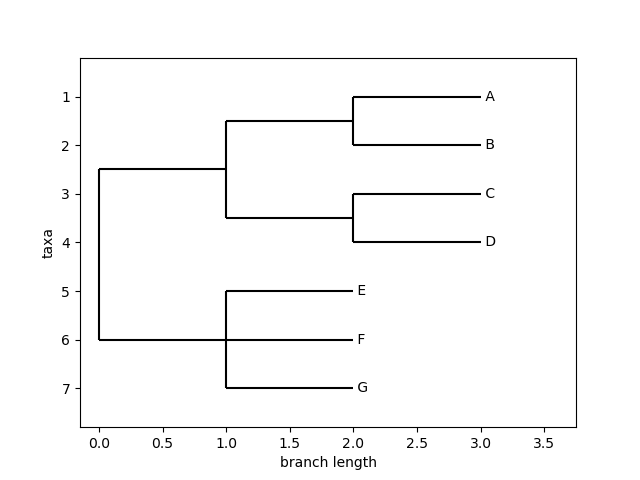
对于需要导出图片的可视化需求,可以通过draw方法来实现,示例如下
>>> tree.rooted=True
>>> Phylo.draw(tree)
输出结果如下
4. 订制分支颜色
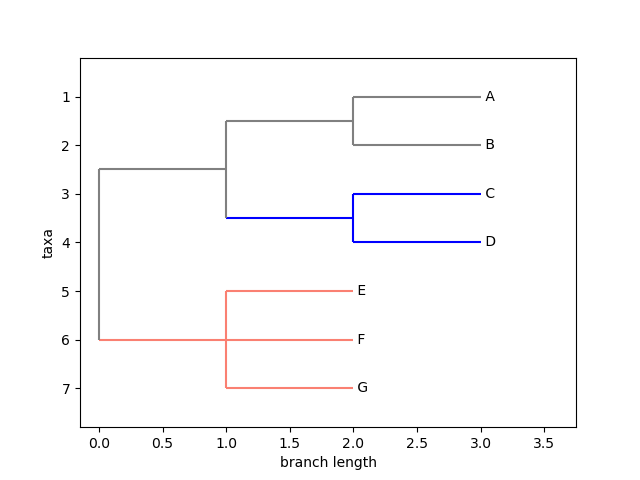
在biopython中,将tree文件转换为xml格式之后,可以详细订制每个分支的颜色,示例如下
>>> tree = tree.as_phyloxml()
>>> tree.root.color = "gray"
>>> mcra = tree.common_ancestor({"name":"E"}, {"name":"F"})
>>> mcra.color = "salmon"
>>> tree.clade[0, 1].color = "blue"
>>> Phylo.draw(tree)
输出结果如下
xml格式的结果也可以输出到文件中,方便后续使用,保存的方式如下
>>> Phylo.write(tree, "tree.xml", "phyloxml")
相比ggtree等专业的树状结构可视化程序,biopython的功能显得有点简陋,对于完全使用python生态的开发者,提供了最基础的展示功能,其最大亮点是分支颜色的高度订制,可以方便的指定各个分支的颜色。
原创不易,欢迎收藏,点赞,转发!生信知识浩瀚如海,在生信学习的道路上,让我们一起并肩作战!
本公众号深耕耘生信领域多年,具有丰富的数据分析经验,致力于提供真正有价值的数据分析服务,擅长个性化分析,欢迎有需要的老师和同学前来咨询。转发本文至朋友圈,后台私信截图即可加入生信交流群,和小伙伴一起学习交流。扫描下方二维码,关注我们,解锁更多精彩内容!