
类固醇(steroid)是一类具有特定四环结构排列的多环化合物,其生物学功能极为广泛。作为调节认知、免疫、代谢和其他生理过程的重要信号分子,类固醇失调会招致众多疾病,如神经退行性疾病、各类炎症和癌症等。此外,化学合成的类固醇还被广泛应用于制药行业(如抗炎剂氢化泼尼松、地塞米松)。因而在科研、工业和临床等领域中,人们对类固醇的鉴定和测量的需求日益增长,其中最主流的分析技术就是质谱(mass spectrometry)。质谱凭借其灵敏性和高通量数据采集的特点,是目前研究生命体内所有小分子代谢物(代谢组)的主要手段。近日,英属哥伦比亚大学(The University of British Columbia)还涛教授课题组提出了一种基于卷积神经网络(CNN)深度学习的生物学驱动代谢组学工作流程,SteroidXtract,可实现在非靶向代谢组学数据集中对类固醇化合物二级质谱谱图(MS2)的自动化快速索取。该方法突破了传统的统计驱动的代谢组学数据处理过程,是一种高灵敏度、高特异性提取类固醇化合物谱图的有效工具。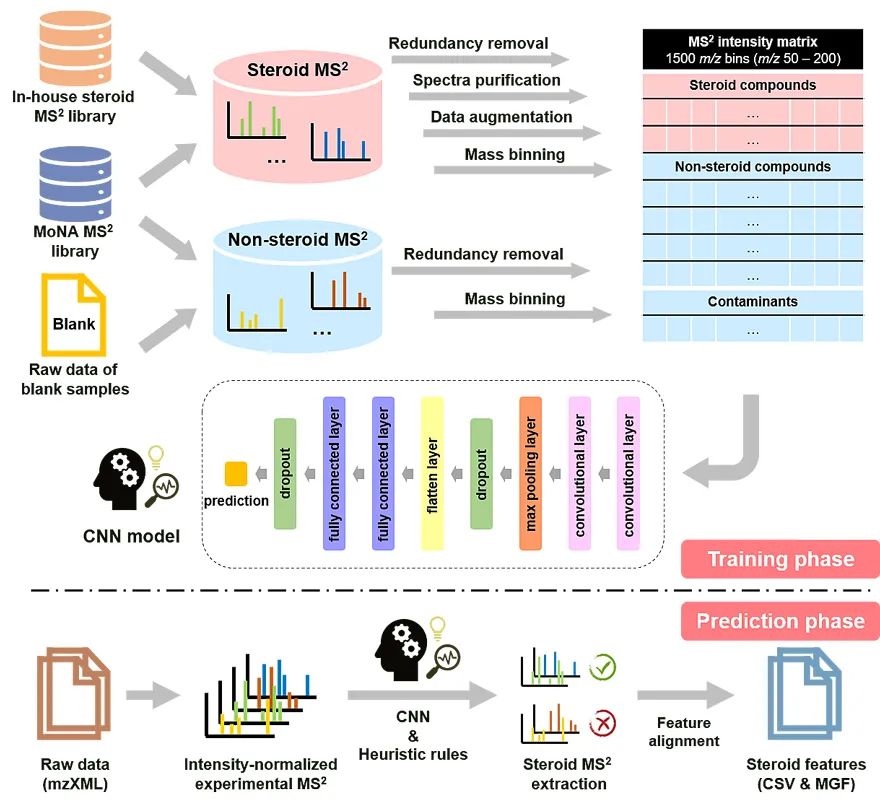
图1. SteroidXtract中深度学习模型的训练和应用。SteroidXtract是一种基于深度学习卷积神经网络(CNN)的生物信息学程序,可以根据其类固醇的二级质谱谱图特异性识别类固醇类代谢物(图1)。SteroidXtract的训练过程中,类固醇和非类固醇化合物的二级质谱谱图都首先经过冗杂去除、谱图纯化、数据增强和数据离散化处理。共有30,176个类固醇二级质谱谱图和106,650个非类固醇二级质谱谱图被采集,用于机器学习模型训练。卷积神经网络模型的搭建基于目前十分流行的Keras机器学习框架和Tensorflow深度学习平台。该深度学习模型可在1个小时内完成训练过程(英特尔i9-9900k CPU @ 3.60 GHz,8核,32 GB内存,Windows 10,64位操作系统)。应用SteroidXtract时,以mzXML格式存储的液相色谱串联质谱(LC-MS/MS)原始数据在Python中被自动读取。所有的实验二级质谱谱图先进行预处理以滤除低质量谱图。SteroidXtract接着对每个二级质谱谱图生成预测结果,存储在CSV表格文件中。从原始数据中提取的类固醇化合物的二级质谱谱图则存储在一个MGF格式文件中。SteroidXtract的处理效率很高——使用如上配置的个人计算机进行测试,处理一个包含10,000个二级质谱谱图的原始数据只需2分钟。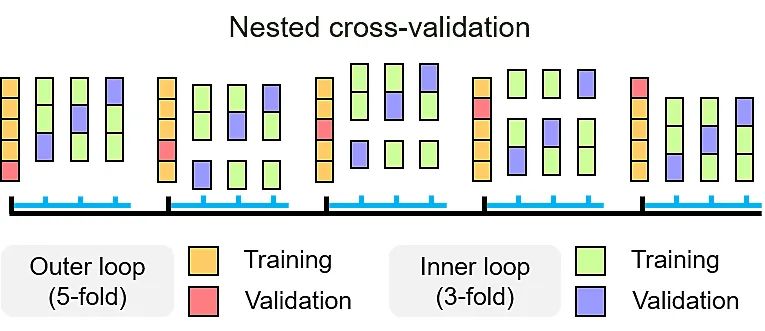
SteroidXtract的预测准确性通过嵌套交叉验证法(图2)进行评估,其中外循环用于算法评估,内循环用于超参数优化。验证结果由宏观平均F1值和宏观平均MCC值进行评估。对比于另外两种常见的机器学习算法随机森林(random forest)和XGBoost,卷积神经网络算法总体表现显著优异,达到了0.991的宏观平均F1值和0.989的宏观平均MCC值。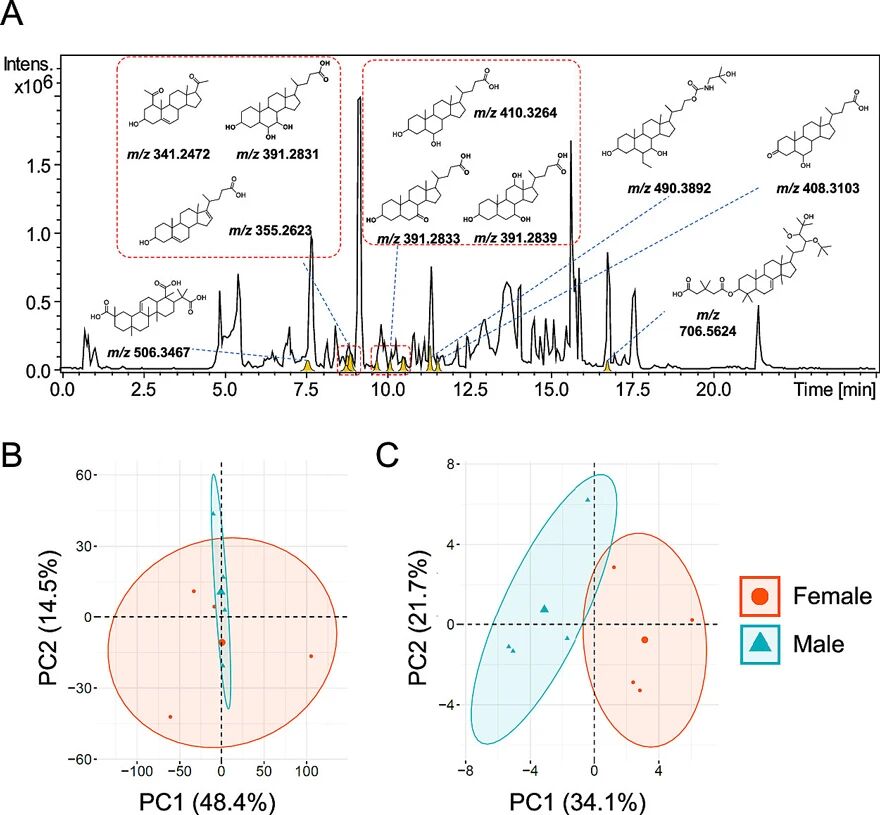
图3. 通过SteroidXtract在小鼠粪便样品中识别的部分类固醇化合物。此外,SteroidXtract还被应用于小鼠粪便样品数据中以测试其性能(图3)。结果显示,在样品数据中不仅检测了已知的类固醇化合物,还找到了一些从未被报道过的类固醇结构。在该工作中,研究人员首次提出了一种生物学驱动的方法来挖掘非靶向代谢组学数据。与传统的代谢组学不同,该方法绕过了统计分析,侧重于提取属于相同代谢类别或具有相似生物学功能的一类化合物。此外,基于深度学习开发的生物信息学软件SteroidXtract是一种可从代谢组学数据中以高灵敏度和特异性提取类固醇化合物的强大工具。生物样品实际应用更揭示了SteroidXtract在发现已知和未知类固醇化合物方面的高效性。可以预见,SteroidXtract为在非靶向代谢组学数据中基于某一类化合物的特征挖掘和识别铺平了道路,为生物学驱动代谢组学数据处理开启了新的篇章。该研究结果发表在国际分析化学权威杂志Analytical Chemistry 上。文章第一作者为英属哥伦比亚大学博士研究生邢世沛,文章的通讯作者是英属哥伦比亚大学化学系还涛教授。SteroidXtract: Deep Learning-Based Pattern Recognition Enables Comprehensive and Rapid Extraction of Steroid-Like Metabolic Features for Automated Biology-Driven MetabolomicsShipei Xing, Yibo Jiao, Melody Salehzadeh, Kiran K. Soma, and Tao Huan*Anal. Chem., 2021, DOI: 10.1021/acs.analchem.0c04834https://www.x-mol.com/groups/huan_tao

本文版权属于X-MOL(x-mol.com),未经许可谢绝转载!欢迎读者朋友们分享到朋友圈or微博!
长按下图识别图中二维码,轻松关注我们!
点击“阅读原文”,查看 化学 • 材料 领域所有收录期刊
