AI 科技评论报道
编辑 | 琰琰
5月11日,一篇题为“Neural network aided approximation andparameter inference of non-Markovian models of gene expression”(人工神经网络辅助的非马尔可夫基因表达模型近似与参数推断)的论文登上Nature子刊《Nature Communications》。

论文地址:https://www.nature.com/articles/s41467-021-22919-1
论文是中国工程院院士钱锋教授团队和英国爱丁堡大学合作撰写,工作由华东理工大学青年教师姜庆超、傅晓鸣等完成,通讯作者是华东理工大学曹志兴教授和英国爱丁堡大学Ramon Grima教授。
该工作的主要内容是:针对细胞内复杂的大规模生化反应,通过将大量中间反应中间步骤等效成一个时滞反应,并采用机理数据深度融合的思想和微分机器学习方法,对时滞随机动态进行高效精确求解,实现对于基因表达实验数据的高通量、高精度解析。
在论文中,作者展示了如何通过结合实验数据,采用神经网络将无法闭式求解的记忆性(非马尔可夫)方程转化成无记忆(马尔可夫)方程,从而实现闭式高效求解。
作者通过测试发现,相较于传统的蒙特卡洛模拟算法,该方法在不牺牲建模精度的前提下,提高计算效率6倍,降低数据依存度至1/30。
美国伊利诺伊大学厄巴纳-香槟分校(UIUC)IdoGolding教授评价说,该工作有望大幅提高研究人员对于基因调控精准建模的能力(”withgreat promise of advancing our ability to model gene regulation with betterrealism”)。
主要思想
在过去二十年里,单细胞实验技术的飞速发展,通过随机动态建模,为探究基因表达噪声如何影响细胞动态提供了崭新的视角。细胞内复杂的大规模生化反应导致难以对所有反应进行细致完备的随机动态表征,只能诉诸等效近似进行刻画。这意味着一个等效反应包含了一系列中间反应步骤,而蕴藏在这生化反应动力学背后的重要假设是无记忆假设,又称马尔可夫假设,即系统的未来动态仅依赖于当前状态。
虽然该假设大大简化了模型分析,但采用该类假设来分析细胞内生化反应动态是值得商榷的。
例如,对于一个基因的转录过程(如上图所示),基因上附着的RNA聚合酶(RNAP)数量是反应基因转录动态的重要变量。RNA聚合酶会首先与启动子结合,标志着转录开始,之后沿着基因转录新生RNA直至转录完成,脱离基因。从动力学角度看,转录的完成与过去的一个转录开始事件的发生相关,这意味着该系统具有记忆,而不是马尔可夫的。诚然,可以通过状态扩增对RNA聚合酶在基因上的运动进行微观表征,但这以大幅增加模型变量为代价,从而无法闭式求解该类马尔可夫模型。
在数学上,研究人员对于非马尔可夫模型的理解远远落后于马尔可夫模型,通常采用计算效率低下的蒙特卡洛模拟方法进行求解,因而亟需突破非马尔可夫模型的解析困难,从而深化对于基因转录过程的量化理解。鉴于神经网络在解决各类科学问题上大放异彩,作者提出了一种基于神经网络的新方法来研究基因转录为代表的非马尔可夫模型。
主要方法与实验结果
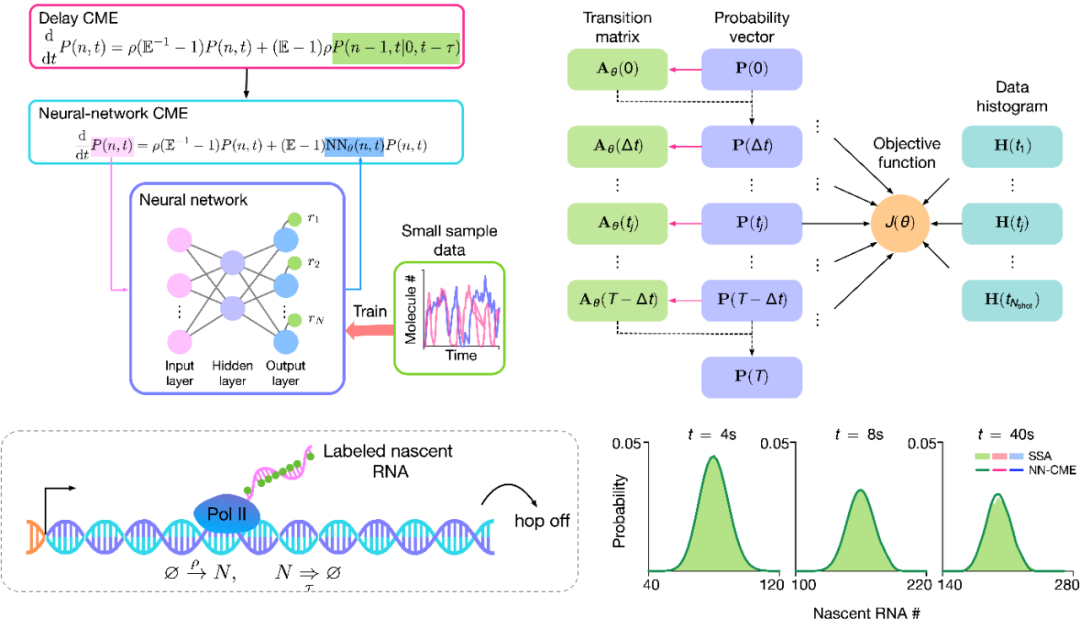
以基因转录过程为典型的非马尔可夫模型,其概率表述通常可以采用时滞化学主方程式(Delay Chemical Master Equation, Delay CME)进行表征,如图1所示。值得注意的是,该方程的左边是关于当前时刻的概率分布,而方程右边包含双时刻的概率分布,因此该方程是不闭合的,也就无法进行求解。
对此,作者提出将双时刻概率分布项用神经网络进行马尔可夫近似,使其仅依赖于当前时刻的概率状态,从而闭合方程。新获得的方程命名为神经网络主方程式(NN-CME),NN-CME在神经网络参数确定的情况下,可以直接通过有限状态映射方法(Finite State Projection, FSP)进行求解。
文中作者采用了具有单个隐藏层的多层感知器(MLP)的神经网络结构,神经网络的输入为当前时刻的概率状态,输出为状态转移概率。该神经网络的参数训练主要包含三个步骤(见图1):
1)对于Delay CME进行小规模蒙特卡洛随机模拟,在若干时刻点进行概率分布采样,并将这些概率分布记为H(t);
2)对于给定参数的神经网络进行数值离散化求解,并获得采样时刻的概率分布,并记为P(t)
;
3)基于H(t)和P(t)计算训练损失函数,判断是否大于阈值。如果损失函数仍大于阈值,通过反向传播计算梯度,并用ADAM算法更新神经网络参数,并重复步骤2,反之,则训练完成。
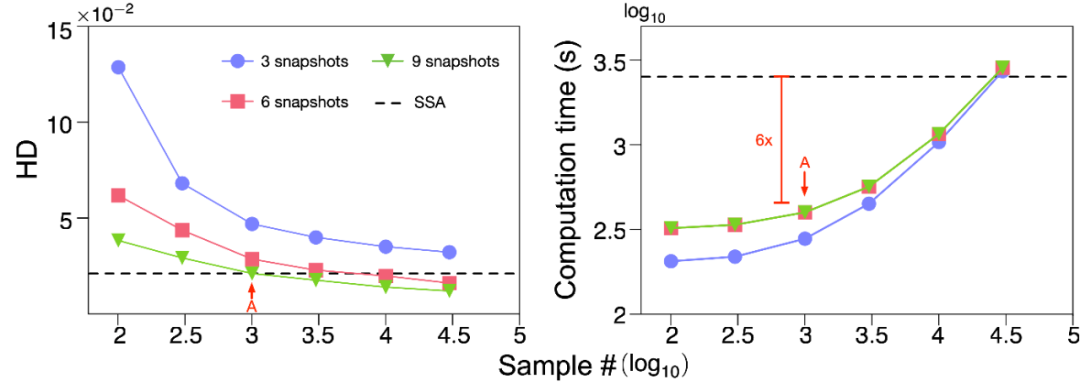
接下来,作者测试了NN-CME的求解方法相对于传统蒙特卡洛随机模拟求解的计算效率。图2中,作者采用上述两种方法求得的新生RNA数量的概率分布与其精确解之间的海林格距离(HD),来表征两种求解方法的精度。图2中显示了NN-CME精度随时间采样次数以及随机模拟次数的变化。值得注意的是,经通过1000次随机模拟得到的直方图来训练NN-CME,其精度与30000次随机模拟的直方图精度相仿,从而提高计算效率6倍,降低数据依存度至1/30。
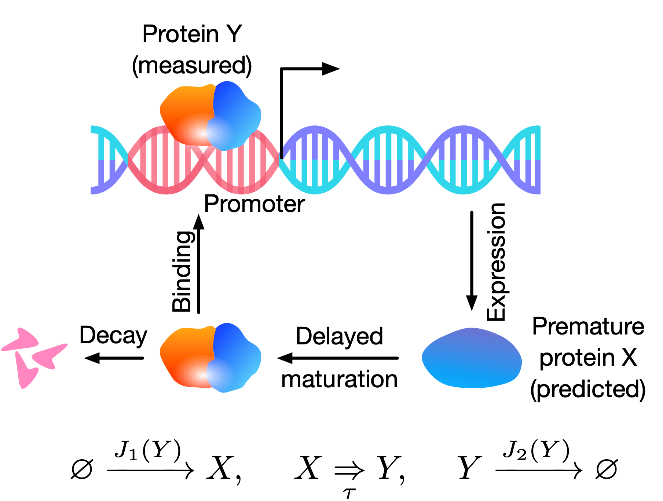
作者进一步采用NN-CME来探究基因震荡电路。如图3所示,基因表达生成中间蛋白X,经过固定时间τ的熟化后,转变成成熟蛋白Y,而蛋白Y可以调控该基因的表达量。
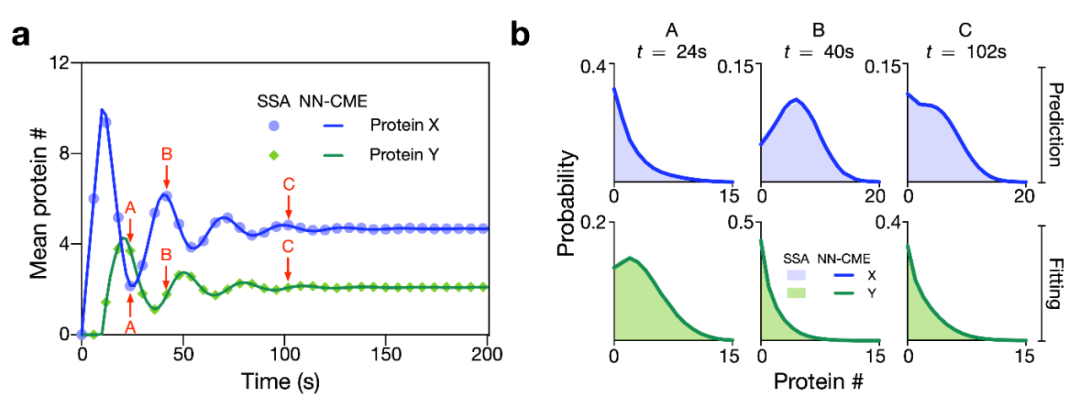
图4:推测隐含反应物数量的均值和概率分布
图4结果显示,当仅采用蛋白Y的数据训练NN-CME后,NN-CME很好地拟合了蛋白Y的均值以及各时间点的概率分布。值得一提的是,NN-CME也很好地预测了蛋白X的均值以及各时间点的概率分布,然而并没有任何关于蛋白X的数据进入训练。因此,说明NN-CME具有很强的发现生化反应动力学规律的能力。
作者还在多个哺乳动物基因的转录阵发式基因网络上进行动力学参数推断的测试。将需要推断的动力学参数并入NN-CME的神经网络参数,并一同进行优化训练。通过图5所示结果发现,该方法得出的阵发频率与阵发幅值与真值非常接近,说明了动力学参数推断的高精度。此外,通过结合轮廓似然法,可以得出推断参数的置信区间,进一步说明该参数推断方法具有很好的鲁棒性。
该NN-CME方法的提出,为通过机理数据融合来实现小样本机器学习提供了新的范式。
相关介绍
钱锋院士简介:

钱锋,自动控制和过程系统工程专家,中国工程院院士,上海市政协副主席,华东理工大学副校长,中国自动化学会会士、监事,中国人工智能学会会士,国务院学位委员会控制科学与工程学科评议组成员。他长期从事化工过程资源与能源高效利用的流程制造智能控制和系统集成优化方法与关键技术研究。创新研发了乙烯装置智能控制与优化运行技术、软件和系统,在国内乙烯行业全面推广应用,成效显著;突破了精对苯二甲酸装置全流程优化运行关键技术,实现工业装置大幅度节能降耗;发明的油品管道在线调合优化控制技术,实现了调合过程实时优化系统长周期高效运行。研究成果已在数十套大型石油化工装置上成功应用,取得了显著经济和社会效益。
曹志兴教授简介:

曹志兴,华东理工大学教授、博士生导师,国家高层次青年人才。2012年本科毕业于浙江大学控制科学与工程学系,2016年博士毕业于香港科技大学化学与生物分子工程学系,其先后于美国哈佛大学、英国爱丁堡大学担任博士后。担任中国自动化学会过程控制专委会委员和智能健康与生物信息专委会委员。他长期从事复杂生化反应智能建模和微分机器学习的前沿研究,多次以一作和通讯作者身份在Nature子刊、美国科学院院刊PNAS、Current Opinion in Biotechnology、IEEE Transactions on Automatic Control等著名期刊发表研究结果。

由于微信公众号试行乱序推送,您可能不再能准时收到AI科技评论的推送。为了第一时间收到AI科技评论的报道, 请将“AI科技评论”设为星标账号,以及常点文末右下角的“在看”。