人、动物和植物的共生微生物组中存在的编码多肽和小蛋白,被认为是微生物中数量巨大的一类“暗物质”,其蕴含的功能多样性有着非常大的想象空间。
例如抗菌肽就是这样一种“暗物质”。先前的研究得出,抗菌肽可以作为非常有潜力的治疗耐药菌的药物或者前体分子,并且不容易产生极强的耐药性,有助于应对当下愈演愈烈的耐药菌感染问题。
因此,挖掘和研究共生微生物组中海量的多肽具有十分重要的意义。
近日,来自中国科学院微生物研究所的团队结合 LSTM、Attention 和 BERT 等多种自然语言处理神经网络模型,建立了一个用于从人类肠道微生物组数据中识别候选腺苷-磷酸(AMP)的统一管道。在被确定为候选 AMP 的 2349 个多肽序列中,有 216 个是化学合成的,其中显示出抗菌活性的有 181 个;并且,在这些多肽中,大多数与训练集中 AMP 的序列同源性低于 40%。
相关论文以《利用深度学习法从人体肠道微生物群中鉴定抗菌肽》(Identification of antimicrobial peptides from the human gut microbiome using deep learning)为题发表在 Nature Biotechnology 上,中国科学院微生物研究所研究员、博士生导师王军担任最后通讯作者。
审稿人评价该研究道,“从计算预测到结果非常好的动物模型,这项研究总结了一系列令人印象深刻的工作,包括一些用于进一步研究的候选肽。使用机器学习发现新的 AMP 后,再对其功效进行详细的微生物学验证,非常有趣,这也许会对该领域产生积极影响。”

图 | 相关论文(来源:Nature Biotechnology)
在微生物以及其他生物体内发挥功能的分子,不仅包括各种代谢途径和通路所产生的小分子,还有一系列的生物大分子。这些大分子有的是生化反应的产物,如细菌细胞壁的肽聚糖和表面的脂多糖等;有的则是直接编码在基因组中的,包括多肽和小 RNA 等。
还是以抗菌肽为例,现在天然界已知的抗菌肽约有几千条,来源非常广泛,从最原始的细菌到高等生物中都有。这些肽在人类和两栖类动物中是天然免疫的组成成分,可用于在细菌中相互竞争和维持群落结构,且具有抗癌、调节免疫和改善代谢等功能。
然而,针对这些多种多样、序列相似性低、功能类型复杂的生物大分子,目前还未有能够将其序列和功能直接联系到一起的方法。
由于大分子序列相对来讲比较短,整体上相似性非常低,传统方法基于序列相似性进行挖掘存在较大的困难。
王军表示,“针对这些特别短、相似性又不高的多肽序列,进行更加准确高效地判别是我们此次研究的核心出发点。”
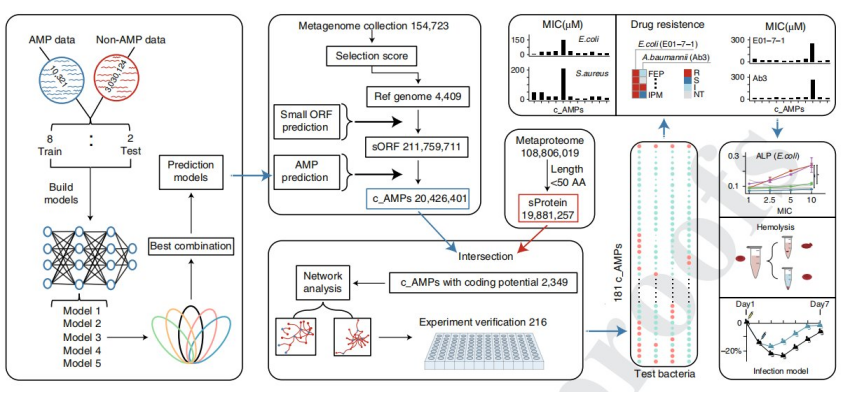
图 | 该团队研究工作流程的示意图(来源:Nature Biotechnology)
据了解,王军团队应用了 AI 领域中自然语言分析(NLP)的最新方法,来对基因组序列进行研究,尤其是其中编码的小蛋白的功能预判。在现有的几千个已知抗菌肽的基础上,构建了多个神经网络模型整合的分析流程,并实现 90% 以上的判定准确率。
接下来,他们运用了现在已积累的大量健康人体微生物组数据,其巨大的编码潜力意味着,存在多种类型的抗菌肽和其他肽类,并且这些肽可能在相互竞争以及与宿主的互作中起着非常重要的作用。
该团队认为,在肠道中表达的多肽应该具有比较好的真核细胞的安全性。为此,他们在 1 万多个微生物组中进行层层数据筛选,逐步降低其假阳性,最终得出,在合成的 200 多条多肽中,有 180 多条肽具有非常明确的抗菌能力,从而验证了其方法的可靠性。
此外,该研究还显示,在大规模的基因组和宏基因组数据中,借助 AI 可以进行特定类群功能分子的直接挖掘和判定,利用高通量筛选验证后,可再进行后续的机理和有效性以及体内研究。
这种研究方法被王军称为“从硬盘到药物”,该方法可以极大提高有治疗前景药物的研究速度和产出率。
王军表示,该研究最初的想法来自与临床的合作。通过之前的多个临床合作,王军团队逐渐意识到,在肠道菌群中与疾病和健康相关的分子不局限于经常研究的小分子,有一系列的蛋白类物质也能够与宿主互作并起到调节免疫、代谢等作用。
例如细菌的多肽,其能够模拟人自身蛋白的序列,从而形成一个“模拟表位”抗原,能够诱导显著的炎症反应并与自身免疫抗体结合。也就是说,微生物基因组直接编码的多肽等大分子,也可以作为功能分子发挥致病或者治病的作用。
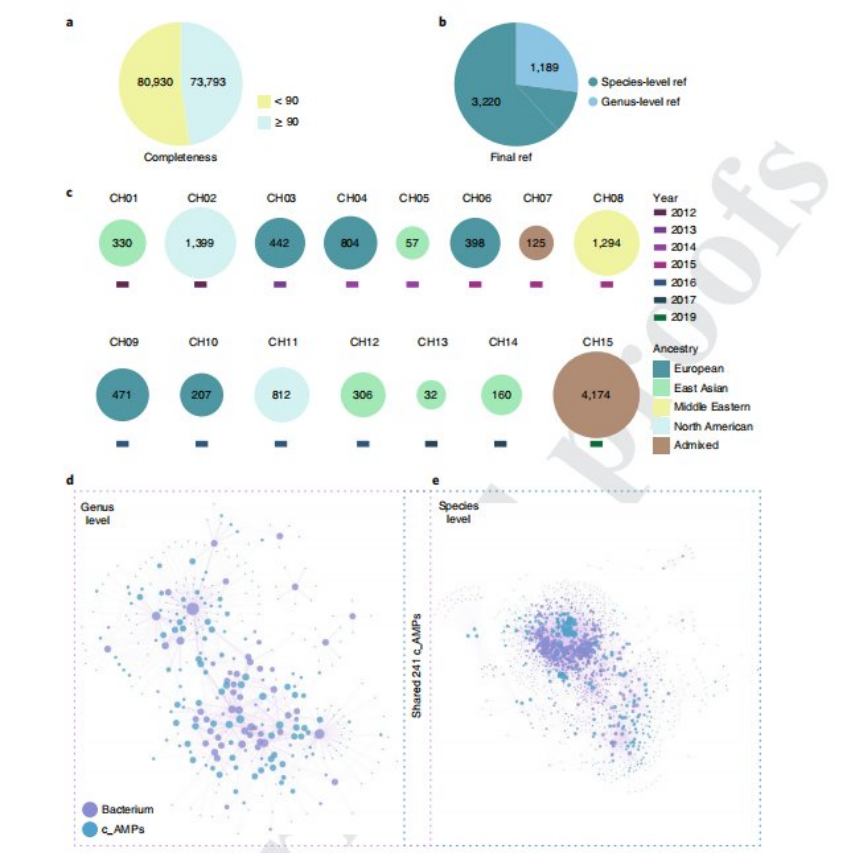
图 | 从宏基因组数据中挖掘候选 AMP(来源:Nature Biotechnology)
该团队认为,虽然现阶段还无法从大量宏基因组数据中有效推断出哪些是代谢所产生的小分子,但其实很多 ORF(Open Reading Frame,可读框)所编码的特定功能蛋白是可以直接挖掘的。问题在于,用什么样的方法能够完成这种短序列的挖掘。
对此,他们利用对计算机领域的深入理解和掌握,建立了以 AI 为基础的预测模型,并将自然语言分析的很多方法转化应用到基因组的挖掘中来。
在一段时间的训练以后,模型的准确度已经达到了一个比较可信的数值,然后该团队用真核数据中的预测对抗菌肽的十个短肽进行了验证,结果发现其中有 8 个具有活性。
接着,他们开始利用现已公开的大量宏基因组数据,进行多肽的挖掘及逻辑推导,并将更多信息整合在一起,以达到更加有效的挖掘。
最后,该团队开始研究合成多肽的机理、安全性与动物实验等,并得出,对真核细胞没有明显毒性的肽能够在动物体内降低感染菌的载量,并有效治疗肺炎克雷伯菌所导致的感染。
王军表示,此次研究还要感谢中国科学院微生物研究所陈义华研究组的大力支持。据了解,两个研究组一起合作解析了多个有潜力的多肽结构及其作用机制,并证实这些肽在结构和机理上均具有较高的多样性。
该研究表明,他们的方法不仅能够发现比较新的肽,而且在机理和结构上没有特定偏好或局限。
值得一提的是,该研究的应用前景极为广泛。一方面,其扩大了微生物组及其他基因组数据的转化出口,将其中编码的很多大分子直接呈现在研究人员眼前,有利于进行多肽类和 RNA 类药物的挖掘;另一方面,伴随测序方法的革新和快速增长的数据,或将出现更多能治疗自身免疫病、代谢类疾病、肿瘤等的多肽。
此外,在现有多肽的基础上,研究人员可以对其进行化学改性(chemical modification),有助于后续稳定性、延长半衰期及安全性的提高,这也是进入临床前不可或缺的一步。
王军称,“我们发现的多肽有望快速进入临床使用,以协助解决现在所面临的耐药菌感染问题和更多重大非传染性慢病等。”

图 | 王军(来源:王军)
目前,王军主要进行生物数据的深度挖掘和分析工作。他利用统计学和生物信息学结合的方法,来分析肠道菌群对于人和动物中的基因组及疾病所起的作用。
截至现在,他已在 Science、Nature Genetics 等刊物上发表了 60 余篇 SCI 论文,并承担了 5 项重大基金项目,申请专利 5 项。
对于该研究,王军称,后续他们将持续扩大所挖掘大分子的应用范畴,将微生物功能大分子从抗感染逐渐拓展到代谢类疾病、免疫性疾病等治疗中。
他表示,“我们还计划对现在的多肽进行临床前的优化,逐步提高成药性和抗菌的范畴,将其进一步优化到革兰氏阳性菌和真菌等的治疗上”
此外,借助 AI 的进步和以往知识的积累,该团队或能实现从头设计出一系列现在天然界中不存在的大分子。
-End-

参考:
1.Yue Ma et al. Identification of antimicrobial peptides from the human gut microbiome using deep learning. Nature Biotechnology, DOI: s41587-022-01226-0 (2022)