hi,大家好,我是晨曦
这期的内容我们来解析一篇非肿瘤机器学习的文献,并且根据这篇文献整合出来一份机器学习的分析模板,相信各位小伙伴只要找到自己领域的数据集并且有了相应的idea,就可以快速根据分析模板产出自己的机器学习文章啦~
那么,我们开始吧

本期文献的中文题目:通过RNA测序分析确定早期诊断精神分裂症潜在的血液生物标志物
首先,我们从题目就可以知道这篇文献切入的主要疾病为精神分裂症,要做的其实就是寻找诊断标志物,那么可以运用到的机器学习算法我们预先可以有一个大概的预想,可能会有SVM、随机森林以及神经网络,当然Logistic回归也是一个不错的选择
1.Schizophrenia (SCZ) :精神分裂症
2.healthy controls (CTL):健康对照
3.Bipolar disorder (BPD):双向情感障碍
4.Major depressive disorder(MDD):重度抑郁症
精神分裂症(SCZ)是一种高度遗传性、多基因的复杂精神障碍,诊断边界不精确。寻找敏感性、特异性的新生物标志物,提高SCZ诊断的生物学同质性仍是研究热点之一
晨曦解读
告诉我们SCZ是一个比较棘手的临床问题,需要特异性的诊断标志物来帮助临床医生
为了鉴定SCZ的血液特异性诊断生物标志物,我们对来自15例首次用药SCZ患者和15例健康对照(CTL)的30份外周血样本进行了RNA测序(RNA-seq)
晨曦解读
这里选择首次用药的患者可能也是考虑,如果患者没有来进行治疗,可能也不会发现这个患者有精神分裂症
通过基于RNA-seq数据和微阵列数据集的多种生物信息学分析算法,包括差异表达基因(DEGs)分析、WGCNA和CIBERSORT,我们首先在SCZ中鉴定了6个特定的关键基因(TOMM7、SNRPG、KRT1、AQP10、TMEM14B和CLEC12A)
晨曦解读
WGCNA分析,本质上来讲就是把基因进行聚类,这样我们就会得到一些表达相似或者协同的基因,然后如果有表型信息,还可以把模块与表型进行关联,这个时候我们就会得到表型和哪些基因相关,当然,解读方向的不同,你把模块内的基因认为是更重要的基因也是可以的,但是需要注意,WGCNA需要的输入数据为表达矩阵(未经筛选的),差异分析后的表达矩阵显然是不行的,因为WGCNA纳入的数据越多,其稳定性是越高的(涉及到构建无尺度网络,当然其中的算法细节,不建议深究,会用即可)

此外,我们发现CTL样本与SCZ样本在淋巴细胞、单核细胞和中性粒细胞的比例有显著差异。因此,我们结合年龄、性别和新的血液生物标志物等各种特征,通过重复的k倍交叉验证,构建了三个分类器(RF:随机森林;SVM:支持向量机;DT:决策树)的风险预测模型,确保了更好的通用性。RF分类器的受试者工作特征面积评分为0.91,在外部验证数据集中ROC为0.77
晨曦解读
和我们预先想的是大致符合的,基本上分类问题需要考虑的就是这几个机器学习的算法
总之,该研究确定了3个外周血核心免疫细胞和6个与SCZ发生相关的关键基因,需要进一步的研究来测试和验证这些新的生物标志物,用于SCZ的早期诊断和治疗
GSE38485——精神分裂症
GSE124326——双向情感障碍
GSE32280——重度抑郁症
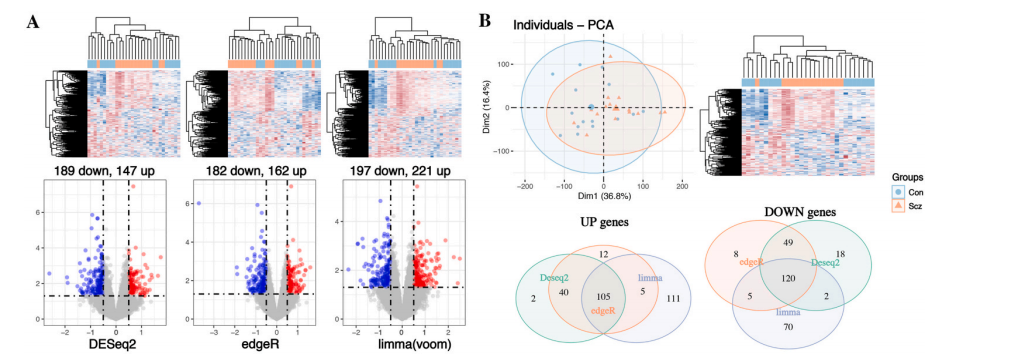
Figure1:展示SCZ与正常对照血液样本之间的差异表达基因
晨曦解读
文章的主要结论并不是我们关心的内容,之所以叫分析模板,是因为我们重点关注作者的分析思路,这里的Figure1A图作者展示了三大差异分析的结果,因为算法的不同,这三种方式肯定会存在差异,所以作者对这三种结果取交集,也就是说,通过Figure1我们获得了SCA与正常对照组之间的差异基因
PCA属于机器学习中无监督学习中聚类的算法,其实就是展示疾病组和正常组是否分割清晰,如果完全重合其实就没有往下分析的必要了,这个图其实应该和差异分析的顺序调换一下,因为这一步是属于数据质控的步骤
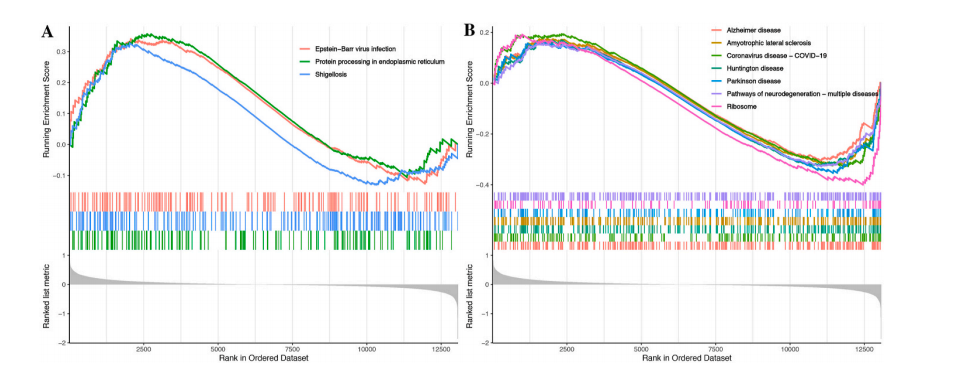
Figure2:明确差异基因的功能——GSEA富集分析
晨曦解读
这一步骤其实是存在问题的,因为小伙伴们都知道,GSEA富集分析需要纳入全部基因,如果单纯取差异基因则会丢失部分信息,因为GSEA提出的一部分原因就在于摒弃掉传统富集分析存在阈值或者之前有logFC筛选的情况,如果我们把经过筛选的基因集纳入到GSEA富集分析中,其实从某种程度上来说是丢掉了GSEA富集分析的优势
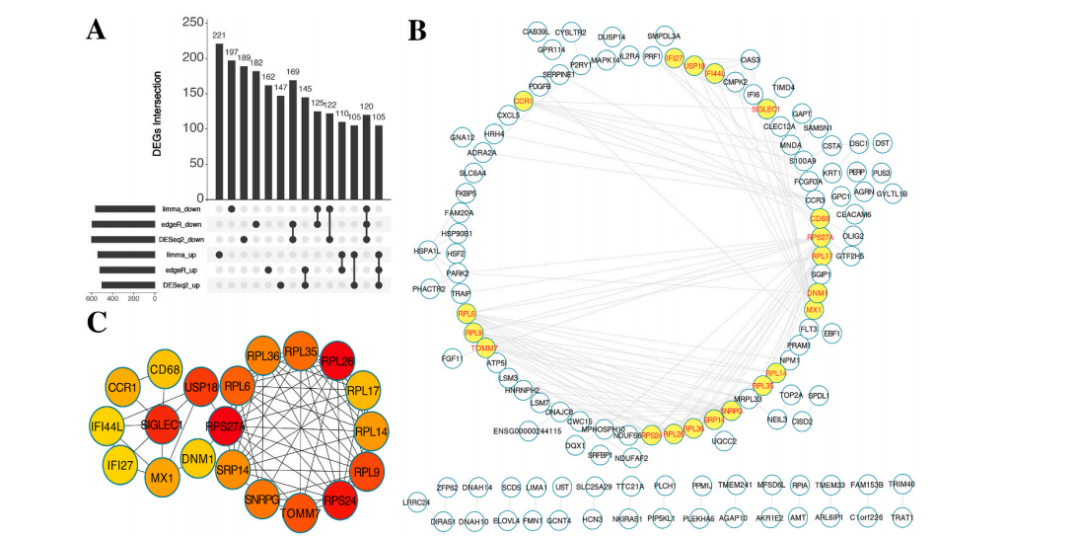
FIgure3:构建差异基因的交互网络
晨曦解读
这一个步骤其实就是我们常说的挑圈联靠中的联的过程,有的同学可能会疑惑FigureA是什么意思,其实很简单,Upset 图和韦恩图类似都是展示差异情况的,如果遇到多分组用韦恩图无法展示,可以考虑使用Upset 图
Figure4:通过WGCNA识别共表达模块以及模块与疾病状态的关系
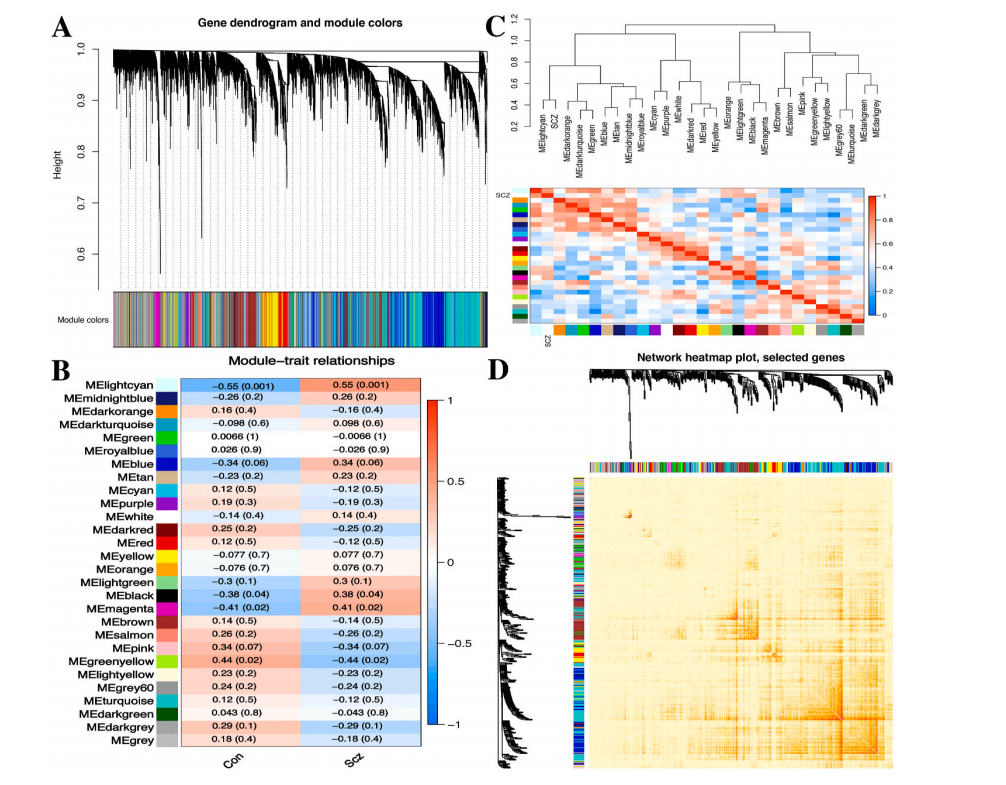
晨曦解读
这一步骤其实就没有过多的修改空间了,就是进行一个WGCNA分析,相关的很多内容在挑圈联靠公众号上都已经连载过很多期了,而且可视化的方式,运用我们的扒图技能也没有太大的一个难度,所以当我们有表型信息的时候,不妨可以考虑进行WGCNA分析来探索一下基因表达与表型之间的相关性(当然,数据挖掘很多的时候你可以都做一遍,然后挑结果好的进行展示)
FIgure5:模块与基因表达情况
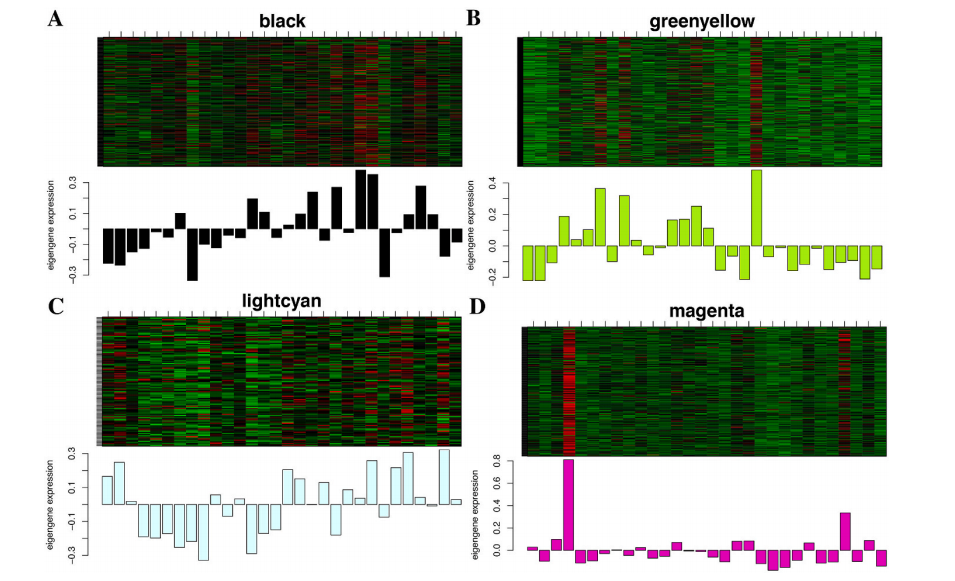
晨曦解读
在这里,作者展示了关键模块中所有基因的表达水平,以及关键模块对于相同测序样本对应的模块基因表达值。红色代表上调的基因,绿色代表下调的基因,这一步骤,其实在别的文献中展示的情况不是很多,在R中运用ggplot2和patchwork即可以完成
Figure6:富集分析
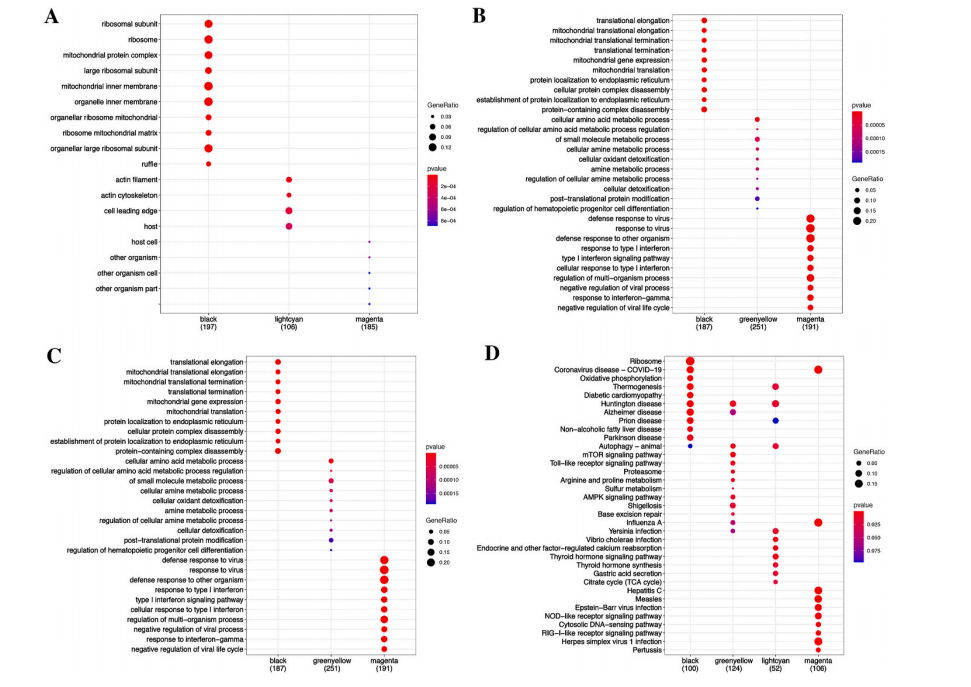
Figure7:进一步筛选关键基因
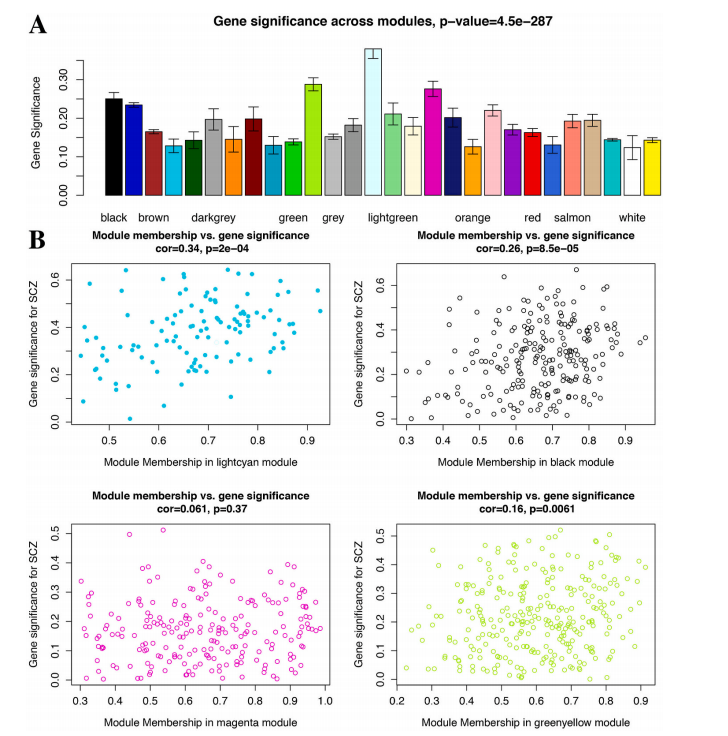
FIgure8:富集分析
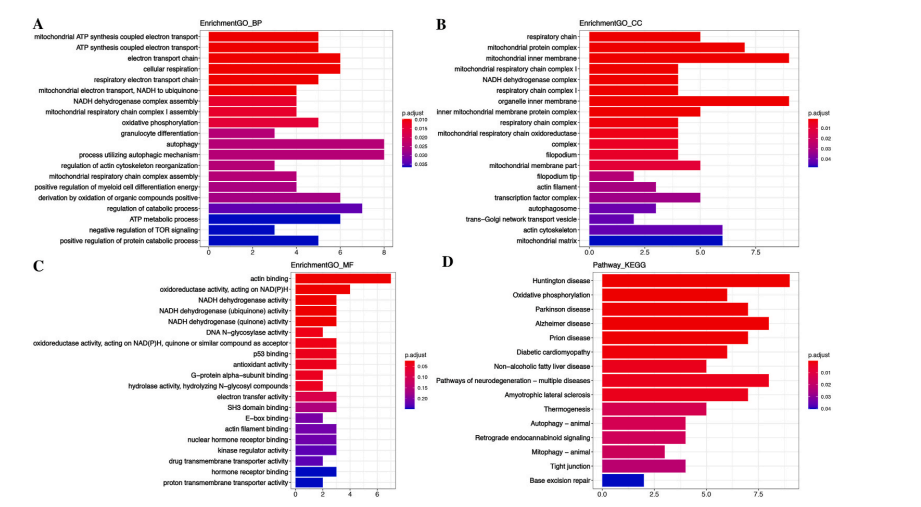
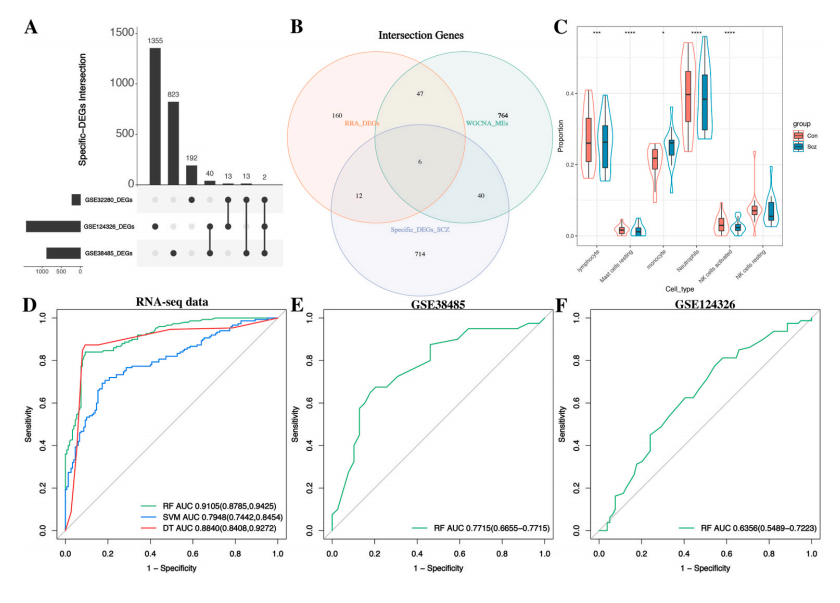
FIgure9
晨曦解读
好,到这里,我们来重点阐述一下Figure9整体的逻辑
这里其实我们就需要与临床预测模型的文献区分开来,临床预测模型最后得到的是一个预测模型,落在结果上其实是得到一个列线图或者网页评分器,但是这个不是,这个是获得一个临床诊断标志物,所以说并不涉及到上面的内容,然后我们来看一下图表的内容
第一步:三个数据集的差异分析,然后通过一种比较少见的可视化方式展示,本质上来说也就是获取三个数据集差异分析的交集(为什么把重度抑郁症还有双向情感障碍纳入到分析流程中,因为晨曦并不是相关专业的,从文章中可知,作者想要探索的标志物是在SCZ中特异性表达,但是在BDP与MDD中不表达的)
第二步:筛选真正的差异基因,寻找在RRA合并三大数据集后与WGCNA得到的hub基因还有SCZ特有的差异基因取交集后作为真正的差异基因(作者认为单纯的SCZ差异基因可能会有不阳性的可能,这个时候通过WGCNA缩小一遍范围,然后再通过RRA合并三大数据集后得到的差异基因再筛选一遍)
如果把三类基因做一个总计可以简单理解为,RRA合并的三大基因集的差异分析相当于土壤,这里的基因是在SCZ或者BDP或者MDD中有差异表达的,然后通过WGCNA也获得了一些模块内的hub基因,最后则是SCZ单独进行差异分析的基因,三者的交集才是最终可能的诊断标志物
第三步:然后把交集后的6个Gene symbol,做一个箱线图来看一下在样本中的表达
第四步:通过随机森林、支持向量机、决策树来评估标志物的诊断效力并在另外两个测试集上进行验证
这里晨曦读到的时候,其实是在想一个问题,展示模型的第一张图是在哪个数据集上进行验证的,结果看到方法学部分是自测数据,但是作者在全文中并没有提供自测数据的编号,所以,这部分自测数据我们是拿不到的(痛失我爱TUT)
好,到这里,整篇文献就给大家介绍到了这里
其实我们可以看出,这篇文献并没有使用太过高深的分析思路,所以如果是咱们挑圈联靠的老粉丝,花上一段时间来复现也是没有问题的,其实重点就是结合了机器学习的最后一张图,我们可以用机器学习来评估我们诊断标志物的诊断效力
那么本期推文到这里就结束啦~
下期推文估计晨曦会带大家来练习一下空间转录组的数据,也算是晨曦最近学习的笔记吧,敬请期待~
我是晨曦,我们下期再见
晨曦单细胞文献阅读系列
晨曦单细胞笔记系列传送门
1. 首次揭秘!不做实验也能发10+SCI,CNS级别空间转录组套路全解析(附超详细代码!)
2. 过关神助!99%审稿人必问,多数据集联合分析,你注意到这点了吗?
3. 太猛了!万字长文单细胞分析全流程讲解,看完就能发文章!建议收藏!(附代码)
4. 秀儿!10+生信分析最大的难点在这里!30多种方法怎么选?今天帮你解决!
5. 图好看易上手!没有比它更适合小白入手的单细胞分析了!老实讲,这操作很sao!
6. 毕业救星!这个R包在高分文章常见,实用!好学!
7. 我就不信了,生信分析你能绕开这个问题!今天一次性帮你解决!
晨曦单细胞数据库系列传送门
1. 宝儿,5min掌握一个单细胞数据库,今年国自然就靠它了!(附视频)
2. 审稿人返修让我补单细胞数据咋办?这个神器帮大忙了!
3. 想白嫖、想高大上、想有高大上的SCI?这个单细胞数据库,你肯定用得上!(配视频)
4. What? 扎克伯格投资了这个数据库?炒概念?跨界生信?
5. 不同物种也能合并做生信?给你支个妙招,让数据起死回生!
6. 零成本装逼指南!单细胞时代,教你用单细胞数据库巧筛基因,做科研!
7. 大佬研发的单细胞数据库有多强? 别眼馋 CNS美图了!零基础的小白也能10分钟学会!
8. 纯生信发14分NC的单细胞测序文章,这个北大的发文套路,你可以试下!实在不行,拿来挖挖数据也行!
9. 如何最短时间极简白嫖单细胞分析?不只是肿瘤方向!十分钟教你学会!
10. 生信数据挖掘新风口!这个单细胞免疫数据库帮你一网打尽了!SCI的发文源头!

欢迎大家关注解螺旋生信频道-挑圈联靠公号~