
“实际上,我们人类所处的世界是一个由分子组成的世界。要了解这个客观物质世界,就要先弄清楚分子的性质,比如分子组成、结构和运动特征等。”近日,在未来论坛主办的 2022《理解未来》科学讲座 02 期——“AI + 分子模拟与药物研发”活动期间,北京大学化学与分子工程学院教授、北京大学理学部副主任高毅勤如是说。
 ▲图 | 高毅勤(来源:资料图)
▲图 | 高毅勤(来源:资料图)
在研究与分子结构及运动相关的各种物理、化学性质时,一般需要用到分子模拟。其中,蒙特卡洛方法和分子动力学是两种主要的模拟方法,它们可用来模拟分子本身发生的结构和化学变化、以及探索分子的热力学与动力学性质等。目前,分子模拟已在多个学科领域得到广泛应用。
比如,在生物医药领域,研究者可以借助分子模拟来研究药物分子和蛋白质的相互作用,这有利于设计更好的小分子,通过提高它的靶向性和结合能力,来进一步优化药物分子。在材料领域,通过分子模拟,则可让人认识到电池的分子结构与分子运动情况,加速完成电池材料或高分子材料的改造和设计。
近年来,随着人工智能的深入发展,人们发现将人工智能算法与科学计算协同起来,即可形成闭环的研究模式,从而加快推进相关领域的革新。

SPONGE:高效融合分子模拟与深度学习,提升模拟效率和准确度
为了实现深度学习与分子模拟的高效融合,高毅勤课题组开发了新一代具有自主知识产权的开源分子模拟软件 SPONGE(Simulation Package tOward Next GEneration molecular modeling)。在此基础上,还与华为昇思 MindSpore 团队合作,将 SPONGE 植入华为自研的开源 AI 框架 MindSopre 中,让该框架得以同时具备 AI 能力和传统分子模拟能力。
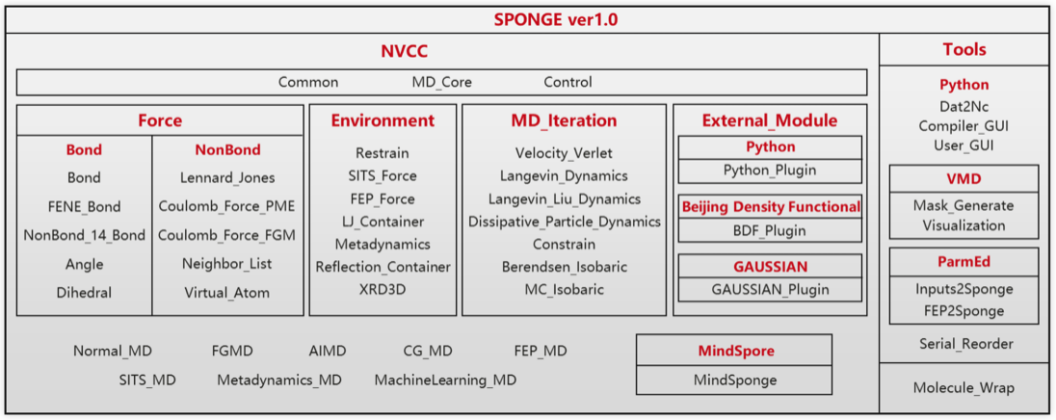 ▲图|SPONGE 软件架构图(来源:北京大学官网)
▲图|SPONGE 软件架构图(来源:北京大学官网)
在 SPONGE 的研发中,该团队总结和参考了大量前人经验,因此包含了传统分子模拟软件的大多数常用功能,可以方便地完成初始结构和输入文件的准备和处理、进行分子结构优化、支持各种系综下的分子动力学模拟、以及计算分子体系的热力学和动力学性质等工作任务。
不过,SPONGE 采用新型计算模型,目前有两个相对独立的版本,一个接近于传统模型,另一个则与深度学习结合得较为紧密。同时,它基于模块化思想写就,将分子模拟中诸如运动方程演化、优化结构、控制温度/压强和增强取样等功能都做成了模块,这能帮助开发者根据需求组装出多种具有不同功能的模块。
高毅勤解释道:“它是一个组合相对自由的软件。比如,如果你感兴趣的是优化问题,可以直接调用优化模块;如果感兴趣的是复杂构象变化问题,可以调用增强取样模块。根据不同的计算要求,进行相对自由的组合,从而加速提高计算效率。”
 ▲图|SPONGE 模拟体系示例(来源:北京大学官网)
▲图|SPONGE 模拟体系示例(来源:北京大学官网)
SPONGE 软件的优点主要体现在以下三方面。
第一,力场多样化。它既可以使用经典力场,又可以使用开发者自己开发的深度学习力场,还可以混合使用两种方法。
第二,SPONGE 采用高度融合的深度学习方法,既可以直接使用其中的张量算法来实现高通量计算、使用自动微分完成力的计算,也可以充分利用深度学习框架中的自动并行等先进的特性和技术。
第三,具有独特的处理静电相互作用的算法,能够准确地计算静电力。
 ▲图|SPONGE 区别于其它分子模拟软件的特性(来源:北京大学官网)
▲图|SPONGE 区别于其它分子模拟软件的特性(来源:北京大学官网)
此外,在提高分析模拟的效率和准确度方面,融合进 SPONGE 的 AI 能力和深度学习能力功不可没。
首先,将深度学习里的强化学习和生成对抗学习这些方法和原理,应用到构象搜寻或化学反应途径搜寻当中,可以大大加速取样效率。
其次,运用深度学习方法,能够优化分子模型或力场,使其更加符合实验观察;也可以把那些差别较大、关联度较弱的性质组合到一起,推动计算的优化。
最后,借助深度学习,可利用量子化学计算的结果拟合出深度学习的力场,从而实现以较低计算代价和更高精度完成分子模拟的目标。
随着算力的逐渐强大,研究者需要使用大量实验数据来验证模型。在此过程中,用来训练和校准模型的数据库,可能会面临数据量不充足的问题。并且,由于分子模拟的应用场景较为广泛,针对不同的场景往往需要不同的数据,所以收集数据就变得十分困难。那么,该如何克服这一难题?
高毅勤提供了几个方法。他认为,科学计算本身就是为大规模的深度学习模型提供数据的。因此,若是生物医药应用领域、或电池材料应用领域缺乏标准化的可靠实验数据,可以借助量子化学计算或分子模拟等科学计算,来得到质量较高、同时也较为可靠的数据。这在有效补充实验数据的同时,也能促进深度模型自身的优化与应用。
不过,由于药物分子的构象空间非常大,目前已知的数据比较少,且很多数据没有出自同一个标准,因此会产生不同的表征形式。那么,当设计药物分子时,就会面临较多问题。在此情况下,如果借助相对高精度、且能快速完成高通量计算的分子模拟技术,能得到更多小分子和蛋白质结合的数据。那么,就可以给筛选药物分子、完成分子的生成式模型计算以及设计新的药物分子提供很大的帮助。

团队成员学科背景多样,研究计划丰富且颇具挑战性
据此,高毅勤目前就职于北京大学化学与分子工程学院。他和团队主要专注基础理论方面的研究,并在 DNA 和蛋白质结构形成的分子机理、分子模拟方法发展等方向取得了诸多前沿成果。
对于跨学科的研究,研究人员所具备的学科背景也是推动课题发展的一大助力。课题组的成员多数来自理论化学方向,也有不少来自生命科学、物理学、计算机和软件等方向。
未来,该团队将继续深化对 SPONGE 软件的开发和应用推广。从大方向看,不但会进一步发展完善适合的深度学习模型,实现分子模拟与深度学习更为深入的融合,还将更好地实现张量计算,希望在节约计算资源的同时,大大加速计算效率。
从具体的研究方向上看,他们将着重关注生物大分子结构预测,包括蛋白质复合物结构、蛋白质与 DNA 相互作用的结构以及蛋白质与小分子相互作用的结构等,并尝试设计具有特殊功能的蛋白质,有效搜寻蛋白质构象等。
高毅勤认为:“我们想努力做到每个阶段都比前一阶段有更高的精度、更快的速度、更低的算力要求和更广的使用范围。更重要的是,希望大家可以帮助我们一同完善做出来的工具,并期待更多人使用它来推进自己的工作。”
目前,除了完成 SPONGE 这个代表作以外,该团队还在其他领域开展了不少研究。比如,团队会利用 SPONGE 做一些化学反应方面的科学研究,也与其他课题组合作,不断释放 SPONGE 的潜力并推动其发展。
另外,他们还开展了一项与染色质结构相关的研究,尝试从染色质的三维结构出发,整合包括 DNA 甲基化与组蛋白修饰在内的表观遗传信息,试图构建一个从 DNA 到 RNA 再到蛋白质的多层调控网络,以便更好地理解包括发育、分化、疾病发生在内的各种生命过程。

