
今天给大家介绍的是西北农林科技大学李富义教授团队和莫纳什大学宋江宁副教授团队2022年联合发表在Bioinformatics上的文章“DeepGenGrep: a general deep learning-based predictor for multiple genomic signals and regions”。现有的大多数方法都是针对单个物种中的特定类型的基因组信号和区域(genomic signals and regions,GSRs)设计的,本文提出了一种新的通用深度学习框架DeepGenGrep,可以预测基因组序列中的多个GSRs。DeepGenGrep利用三层卷积神经网络和两层长短时记忆网络组成混合神经网络,预测多种GSRs。实验表明,DeepGenGrep的预测性能优于现有的方法。
Motivation:从 DNA 序列中准确注释不同基因组信号和区域 (GSRs) 对于理解基因结构、调控和功能至关重要。已经做出了许多努力来开发基于机器学习的预测器,用于GSRs的计算机识别。然而,识别 GSRs仍然是一个巨大的挑战,因为大多数现有方法的性能并不令人满意。因此,非常需要开发更准确的 GSRs 预测计算方法。Results:在这项研究中,本文提出了一个称为 DeepGenGrep 的通用深度学习框架,它是从基因组 DNA 序列中系统识别多个不同 GSRs 的通用预测器。DeepGenGrep 利用由三层卷积神经网络和两层长短期记忆组成的混合神经网络,有效地从序列中学习有用的特征表示。实验表明,DeepGenGrep 在识别四种真核物种(包括Homo sapiens, Mus musculus, Bos taurus, Drosophila melanogaster)的多腺苷酸化信号(PAS)、翻译起始位点(TIS)和剪接位点方面优于几种最先进的方法。 总体而言,DeepGenGrep 代表了一种有用的工具,可用于高通量和经济高效地识别真核基因组中的潜在 GSRs。真核生物具有多个基因组信号和区域(GSRs),包括多聚腺苷酸化信号(polyadenylation signal, PAS)、剪接位点和翻译起始位点(TIS)。GSRs在真核细胞中调节基因表达发挥着重要作用。在这种背景下,准确识别这些GSRs可以更好地理解基因结构。目前已经有很多基于机器学习的方法来识别特定类型的GSRs,但是这些方法仍然存在以下几个缺点:1.性能主要取决于手工制作的特征,但是这类特征可能包含噪声、无关或冗余特征,对模型的训练和性能评估产生负面影响;
2.大多数方法都是针对单个物种中的特定类型的GSRs设计和评估的。只有一种方法DeepGSR可以同时识别各种GSRs;
3.尽管DeepGSR比其他方法实现了更好的性能,但DeepGSR仅针对两个信号设计,仍有进一步的改进空间。
本文提出了一个名为DeepGenGrep的通用深度学习框架,用于预测基因组序列中的多个GSRs。它在识别四种真核物种(Homo sapiens, Mus musculus, Bos taurus, Drosophila melanogaster)的PAS、TIS和剪接位点方面优于目前几种最先进的方法,与DeepGSR相比,DeepGenGrep在迁移学习能力上也有了很大的提高。3.1数据集
在本研究中,有三种类型的GSRs用于模型训练和评估,包括PAS、TIS和剪接位点。四个物种(Homo sapiens, Mus musculus, Bos taurus, Drosophila melanogaster)的PAS和TIS数据集取自DeepGSR数据集。剪接位点数据集来自人类全基因组数据集(GWH)。
3.2DeepGenGrep的框架
DeepGenGrep由一个输入层、三层卷积神经网络(CNNs)、两层长短时记忆网络(LSTMs)、一个完全连接层和一个输出层组成。
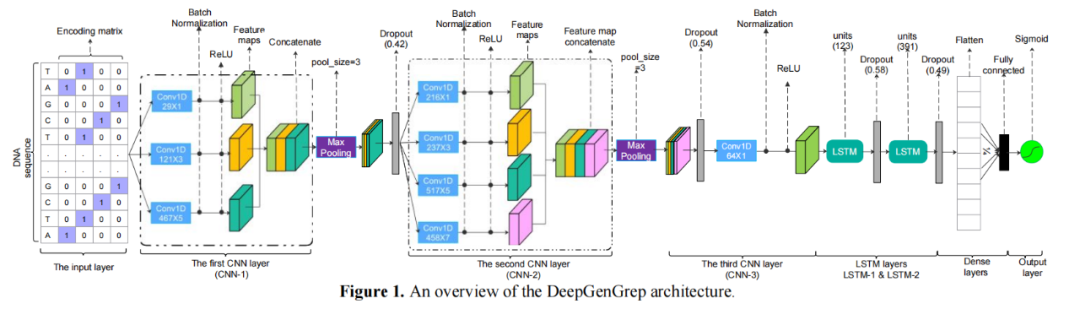
本文采用one-hot编码的方式来表示输入层中的核苷酸序列。
首先将DNA序列S编码到l×4矩阵中,l是S的长度。编码矩阵输入到具有不同尺度核的三层CNN块(即conv1、conv2和conv3)中,以学习局部特征。为了防止过度拟合,使用最大池化(max pooling)对三个CNN块的特征图进行下采样。随后,使用两个LSTM层来关注与顺序相关的输入序列。然后,使用具有一个单元的密集层和sigmoid激活来输出序列S包含特定GSRs的概率。
4.1 性能评估
在本节中,本文在三个数据集上进行比较和评估,分别为四种真核物种(Homo sapiens, Mus musculus, Bos taurus, Drosophila melanogaster)的PAS、TIS和剪接位点。
以TIS在Homo sapiens数据集上的结果为例,训练损失从1到60不断减少,验证损失在第7阶段取得最小值,验证准确性和训练准确性都是逐渐增加,到30取得最佳值,30到60逐渐平稳没有进一步提高,所以在60中断训练。可以看出,DeepGenGrep模型在预测TIS方面取得了优异的性能。

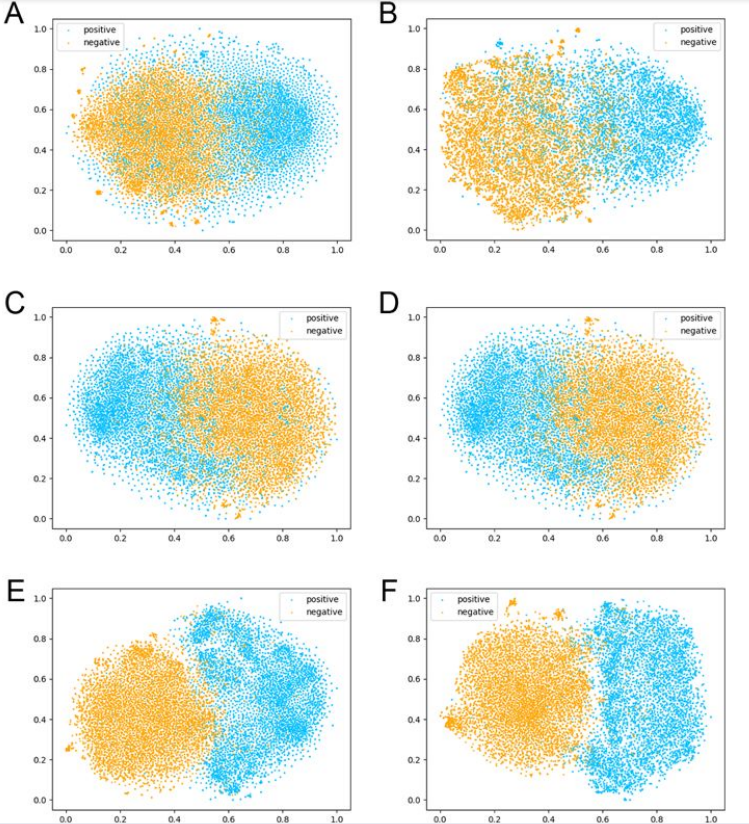
4.2 特征可视化结果
将自动编码器与t-SNE相结合,其中,自动编码器用来降维,t-SNE算法用来可视化。
可以看出,经过三层CNN和两层LSTM之后数据点几乎被划分成了两组(TIS信号的数据点逐渐与非TIS信号的数据点分离)。
4.3 消融分析
本文从完整的框架中删除五个模块中的一个来构建五个比较框架,用这五个比较框架与完整的模型进行比较,通过消融分析可以看出完整的模型效果比删除某一模块的效果好,所以这个模型中的每一层都是必不可少的。

4.4 与先进方法的性能比较
图A显示了DeepGenGrep和DeepGSR在四种物种中根据Acc识别TIS的独立测试结果。可以看到,DeepGenGrep在所有四个测试数据集上都明显优于DeepGSR,Homo sapiens的Acc分别为97.35%和94.32%。

在本研究中,作者开发了一个通用的深度学习框架,称为DeepGenGrep,用于从DNA序列中识别多种不同类型的GSRs。DeepGenGrep配备了由多个具有不同内核大小的1D-CNN组成的CNN块,以捕获不同的特征。通过堆叠三个CNN块和两层LSTM,增强了DeepGenGrep的特征表示能力。消融研究表明,DeepGenGrep框架为GSRs的识别提供了合理的性能。特征可视化表明,DeepGenGrep可以有效地学习特征表示来识别GSRs。基准测试和比较分析表明,DeepGenGrep在预测PAS、TIS和剪接位点方面比几种最先进的预测因子表现更好。此外,从Homo sapiens到其他三个物种的跨物种预测表明,DeepGenGrep比DeepGSR具有更好的转移学习性能和能力,DeepGSR是多个GSRs的最先进的通用预测因子。DeepGenGrep有望被更广泛的研究群体作为高通量和经济高效地鉴定真核基因组中假定的遗传物质受体的有价值的工具加以探索。
 文章地址
文章地址
https://doi.org/10.1093/bioinformatics/btac454
 代码地址
代码地址
https://github.com/wx-cie/DeepGenGrep/