合成生物学技术——机器学习
Nat Commun. 2022 Dec 15;13(1):7755. doi: 10.1038/s41467-022-34902-5.
蛋白质表达深度学习模型的准确性和数据效率。Accuracy and data efficiency in deep learning models of protein expression.
Nikolados EM(1), Wongprommoon A(1), Aodha OM(2)(3), Cambray G(4)(5), Oyarzún DA(6)(7)(8).
Author information: (1)School of Biological Sciences, University of Edinburgh, Edinburgh, EH9 3JH, UK. (2)School of Informatics, University of Edinburgh, Edinburgh, EH8 9AB, UK. (3)The Alan Turing Institute, London, NW1 2DB, UK. (4)Diversité des Génomes et Interactions Microorganismes Insectes, University of Montpellier, INRAE UMR 1333, Montpellier, France. (5)Centre de Biologie Structurale, University of Montpellier, INSERM U1054, CNRS UMR5048, Montpellier, France. (6)School of Biological Sciences, University of Edinburgh, Edinburgh, EH9 3JH, UK. d.oyarzun@ed.ac.uk. (7)School of Informatics, University of Edinburgh, Edinburgh, EH8 9AB, UK. d.oyarzun@ed.ac.uk. (8)The Alan Turing Institute, London, NW1 2DB, UK. d.oyarzun@ed.ac.uk.
合成生物学通常涉及改造微生物菌株来表达高价值蛋白质。由于快速DNA合成和测序的进展,深度学习已经成为一种有前途的方法来建立序列到表达模型,以进行菌株优化。但这样的模型需要大量昂贵的训练数据,这为许多实验室设置了陡峭的进入壁垒。在这里,我们研究了在不同大小和序列多样性的数据集上训练的机器学习模型集的准确性和数据效率之间的关系。我们表明,深度学习可以用比以前想象的小得多的数据集实现良好的预测准确性。我们证明了受控的序列多样性可以显著提高数据效率,并使用可解释的AI来证明卷积神经网络可以很好地区分输入的DNA序列。我们的研究结果为设计基因型-表型筛选提供了指导方针,以平衡训练数据的成本和质量,从而有助于促进深度学习在生物技术领域的广泛采用。
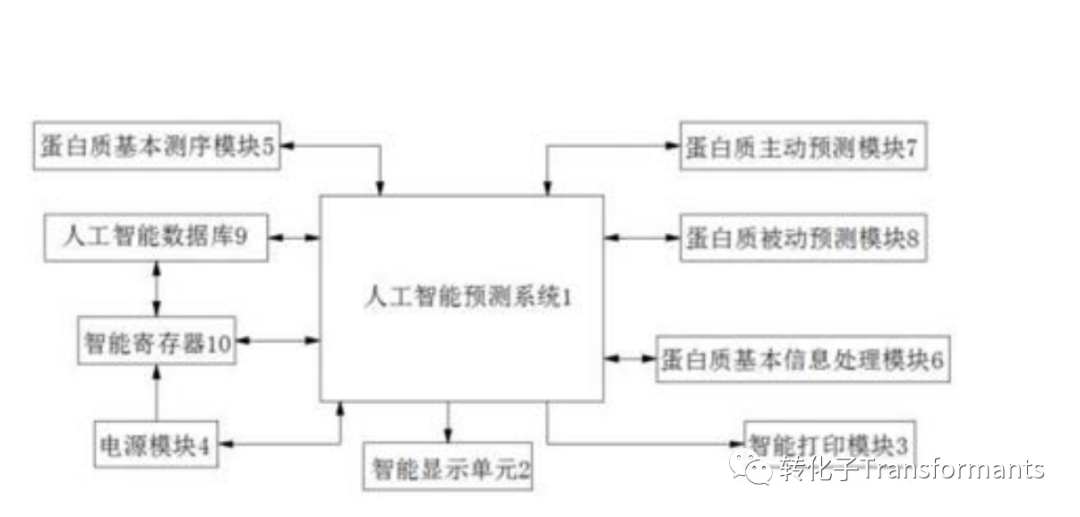
CN110931078A 一种基于人工智能的蛋白质互作组预测服务系统
该发明公开了一种基于人工智能的蛋白质相互作用预测服务系统,主要包括以人工智能为核心的高效预测系统,所述人工智能预测系统的输出端分别与智能显示单元和智能打印模块的输入端电性连接,所述人工智能预测系统的输入端与电源模块的输出端电性连接,所述电源模块的输出端与智能寄存器的输入端电性连接,该发明涉及生物大分子预测服务技术领域。该基于人工智能的蛋白质互作组预测服务系统,通过蛋白质基本测序模块中蛋白质理化处理模块和蛋白质基本测序模块,利用蛋白质基本信息处理模块中蛋白质基本信息填充模块和蛋白质信息自动比对模块对蛋白质基本信息的处理,从而有效的提高了蛋白质的已知相关数据,该发明加入了蛋白质的主动和被动的预测模块,进一步提高了后续的预测精度。