合成生物学技术——机器学习
Sci Adv. 2023 Jan 13;9(2):eadc9130. doi: 10.1126/sciadv.adc9130. Epub 2023 Jan 11.
机器学习能够预测细菌代谢系统的进化。Machine learning enables prediction of metabolic system evolution in bacteria.
Konno N(1), Iwasaki W(1)(2)(3)(4)(5)(6).
Author information: (1)Department of Biological Sciences, Graduate School of Science, The University of Tokyo, Bunkyo-ku, Tokyo 113-0032, Japan. (2)Department of Integrated Biosciences, Graduate School of Frontier Sciences, The University of Tokyo, Kashiwa, Chiba 277-0882, Japan. (3)Department of Computational Biology and Medical Sciences, Graduate School of Frontier Sciences, The University of Tokyo, Kashiwa, Chiba 277-0882, Japan. (4)Atmosphere and Ocean Research Institute, The University of Tokyo, Kashiwa, Chiba 277-0882, Japan. (5)Institute for Quantitative Biosciences, The University of Tokyo, Bunkyo-ku, Tokyo 113-0032, Japan. (6)Collaborative Research Institute for Innovative Microbiology, The University of Tokyo, Bunkyo-ku, Tokyo 113-0032, Japan.
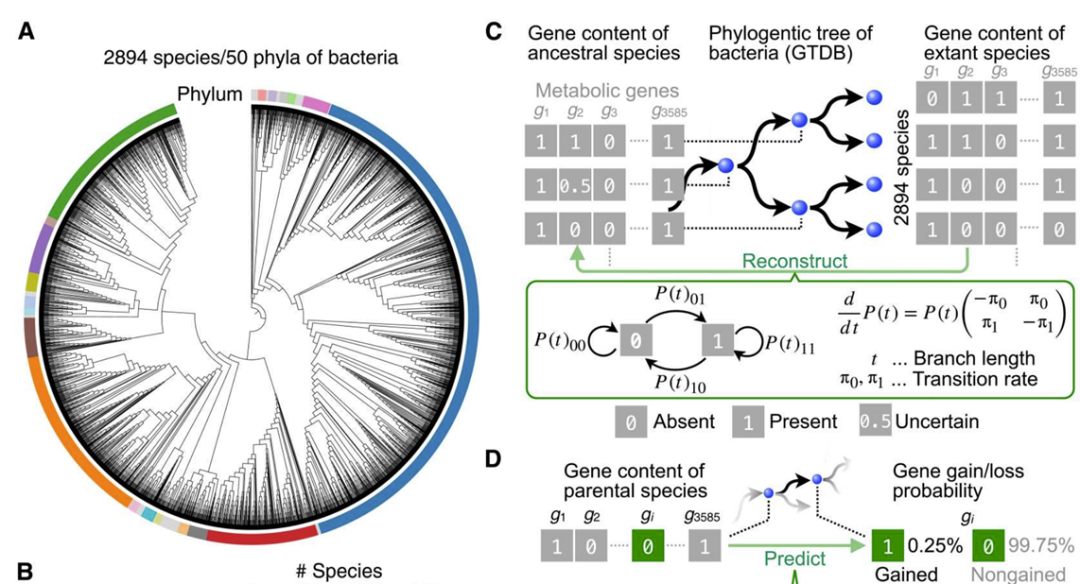
进化预测是进化生物学的一个长期目标,对病原体控制、基因组工程和合成生物学具有潜在的影响。虽然实验室进化研究已经表明了短期和序列级进化的可预测性,但长期和系统级进化的可预测性还没有得到系统的研究。在这里,我们表明,代谢系统的基因含量进化通常是可预测的,通过将祖先基因含量重建和机器学习技术应用于约3000个细菌基因组。我们的框架Evodictor成功地预测了参考系统发生树分支的基因增益和损失进化,这表明代谢系统的进化压力和限制是普遍共享的。途径结构的研究和宏基因组数据集的分析证实,这些进化模式具有代谢反应和细菌栖息地变化之间的功能依赖的生理和生态基础。最后,对种内基因含量变化的泛基因组分析证明,即使是现有细菌物种的“正在进行的”进化,在我们的框架中也是可预测的。
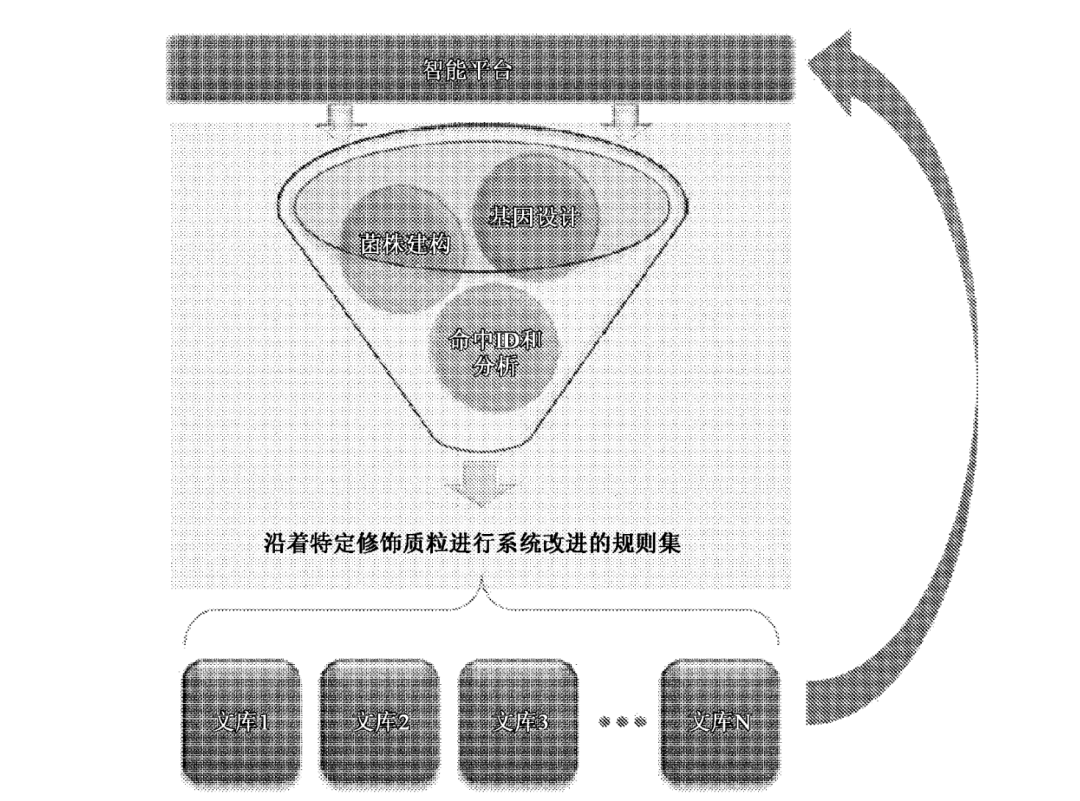
CN110945125A 用于改进大肠杆菌的HTP基因工程改造平台
一种使大肠杆菌微生物进化以获得所期望表型的高通量(HTP)基因工程改造方法,其包括: a.扰动多种具有相同基因组菌株背景的初始大肠杆菌微生物的基因组,借此创建包括具有独特基因变异的个别大肠杆菌菌株的初始HTP基因设计大肠杆菌菌株文库; b.针对所述所期望表型来筛选和选择所述初始HTP基因设计大肠杆菌菌株文库的个别菌株; c.提供各自包括基因变异独特组合的后续多种大肠杆菌微生物,所述基因变异选自所述前一步骤中筛选的至少两种个别大肠杆菌菌株中所存在的基因变异,借此创建后续HTP基因设计大肠杆菌菌株文库; d.针对所述所期望表型来筛选和选择所述后续HTP基因设计大肠杆菌菌株文库的个别大肠杆菌菌株;和e.以线性或非线性方式重复步骤c)-d)一或多次,直到大肠杆菌微生物已经获得所述所期望表型为止,其中每次后续迭代创建新的HTP基因设计大肠杆菌菌株文库,所述新的HTP基因设计大肠杆菌菌株文库包括具有独特基因变异的个别大肠杆菌菌株,所述独特基因变异是选自前一个HTP基因设计大肠杆菌菌株文库的至少两种个别大肠杆菌菌株的基因变异的组合。