1.机器学习基本概念介绍
2.常用机器学习模型介绍
3.混淆矩阵
4.ROC曲线
5.主成分分析(PCA)
6.蛋白组学基本概念
R语言简介及实操
1.R语言概述
2.R软件及R包安装
3.R语言语法及数据类型
4.条件语句
5.循环
6.函数
7.常用的机器学习相关R包介绍
1.利用机器学习鉴定疾病相关蛋白标志物
2.利用机器学习基于蛋白组学数据预测表型
3.利用机器学习基于蛋白组学数据进行分类
4.利用机器学习基于蛋白组学数据构建预后模型
蛋白组学相关数据库介绍
1.Uniport
2.HPA
3.TCPA
4.CPTAC
利用PLOS Computational Biology(IF:5分)发表零代码工具,轻松完成差异表达分析,常见统计分析,常见可视化,内置7种机器学习方法,轻松调用。
1.数据导入(两套数据,二分类,多分类)
2.数据可视化(散点图,热图,柱形图,相关性热图,火山图,层次聚类图)
3.缺失值填充
4.数据归一化
5.离群值检测/清理
6.常见统计方法应用(t-test, limma, Kruskal-Wallis ,ANOVA, PCA, k-means, 相关性分析)
7.机器学习方法应用(RF, lasso, SVM等)
利用机器学习基于蛋白组学数据预测表型,基于蛋白组学数据复现cell中机器学习分析结果
实操内容
1.蛋白组学数据处理,差异表达分析
2.火山图,多分组热图,多组箱型图展示差异表达分析结果
3.构建Random Forest模型
4.重要蛋白筛选
5.绘制ROC曲线
6.独立测试集检测模型表现
利用机器学习鉴定疾病相关蛋白标志物,基于Olink数据,复现影响因子17分文章中,蛋白数据常规分析+时序蛋白聚类分析+机器学习分析结果
实操内容
1.读取蛋白表达数据
2.差异蛋白挑选,火山图绘制,箱型图绘制
3.时序蛋白表达数据聚类分析
4.构建随机森林模型
5.挑选重要特征
6.独立测试集进行验证
利用机器学习基于质谱的蛋白质组学数据,构建肝病相关分类和预后模型,复现Nature Medicine文章中的机器学习,生存分析,预后模型相关的结果。
实操内容
1.鉴定与不同肝病显著相关的蛋白
2.比较22种不同的机器学习分类器,挑选最优算法构建不同肝病的分类模型
3.独立队列验证模型准确性
4.构建预后模型
5.绘制生存曲线和时间依赖的ROC曲线
案例图片:
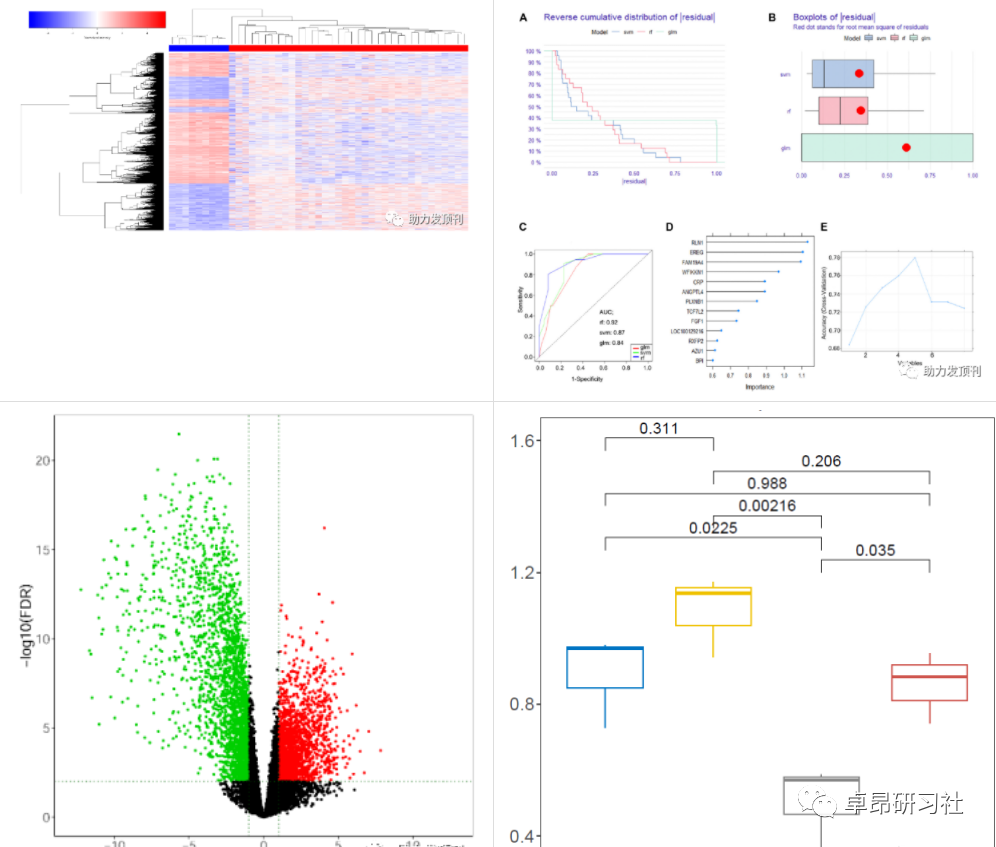
1.机器学习基本概念介绍
2. 常用机器学习模型介绍(GLM,BF,SVM,lasso,KNN等等)
3. 混淆矩阵
4. ROC曲线
5. 主成分分析(PCA)
6. 微生物学基本概念
7. 微生物学常用分析介绍
1.R语言概述
2.R软件及R包安装
3.R语言语法及数据类型
4.条件语句
5.循环
1.利用机器学习基于微生物组学数据预测宿主表
2.利用机器学习基于微生物组学数据预测疾病状态
3.利用机器学习预测微生物风险
4.机器学习研究饮食对肠道微生物的影响
微生物学常用分析(实操)
1. 微生物丰度分析
2. α-diversity,β-diversity分析
3. 进化树构建
4. 降维分析
5. 基于OTU的差异表达分析,热图,箱型图绘制微生物biomarker鉴定
1. 加载数据及数据归一化
2. 构建训练模型(GLM, RF, SVM)
3. 模型参数优化
4. 模型错误率曲线绘制
5. 混淆矩阵计算
6. 重要特征筛选
7. 模型验证,ROC曲线绘制利用模型进行预测
利用机器学习基于微生物组学数据预测宿主表型(二分类变量以及连续变量)
1. 加载数据(三套数据)
2. 数据归一化
3. OUT特征处理
4. 机器学习模型构建(RF, KNN, SVM, Lasso等9种机器学习方法)
5. 5倍交叉验证
6. 绘制ROC 曲线,比较不同机器学习模型模型性能评估
1. 加载数据
2. 机器学习模型构建(RF, gbm, SVM, LogitBoost等等)
3. 10倍交叉验证
4. 模型性能评估
利用机器学习预测刺激前后肠道菌群变化
1. 数据加载及预处理
2. α-diversity,β-diversity分析
3. RF模型构建(比较分别基于OUT,KO,phylum的模型效果)
4. 10倍交叉验证, 留一法验证
5. 特征筛选及重要特征可视化外部数据测试模型
案例图片:
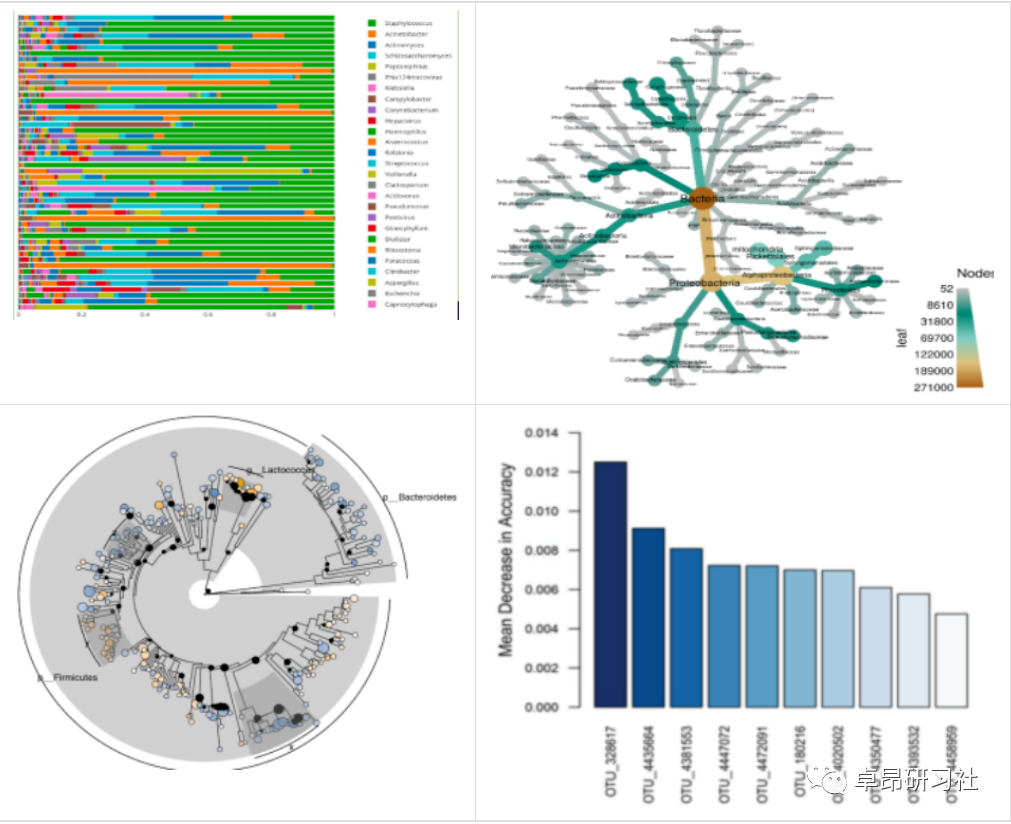
(1) 代谢生理功能;
(2) 代谢疾病;
(3) 非靶向与靶向代谢组学;
(4) 空间代谢组学与质谱成像(MSI);
(5) 代谢流与机制研究;
(6) 代谢组学与药物和生物标志物。
A2 代谢组学实验流程简介
A3 色谱、质谱硬件原理
(1) 色谱分析原理;
(2) 色谱的气相、液相和固相;
(3) 色谱仪和色谱柱的选择;
(4) 质谱分析原理及动画演示;
(5) 正、负离子电离模式;
(6) 色谱质谱联用技术;
(7) LC-MS 的液相系统
A4 代谢通路及代谢数据库
(1) 几种经典代谢通路简介;
(2) 能量代谢通路;
(3) 三大常见代谢物库:HMDB、METLIN 和 KEGG;
(4) 代谢组学原始数据库:Metabolomics Workbench 和Metabolights.
(1) 组织、血液和体液样本的提取流程与注意事项;
(2) 用 ACN 抽提代谢物的流程与注意事项;
(3) 样本及代谢物的运输与保存问题;
B2 LC-MS 数据质控与搜库
(1) LC-MS 实验过程中 QC 样本的设置方法;
(2) LC-MS 上机过程的数据质控监测和分析;
(3) XCMS 软件数据转换与提峰;
B3 R 软件基础
(1) R 和 Rstudio 的安装;
(2) Rstudio 的界面配置;
(3) R 的基本数据结构和语法;
(4) 下载与加载包;
(5) 函数调用和 debug;
B4 ggplot2
(1) 安装并使用 ggplot2
(2) ggplot2 的画图哲学;
(3) ggplot2 的配色系统;
(4) ggplot2 画组合图和火山图;
机器学习
C1 无监督式机器学习在代谢组学数据处理中的应用
(1) 大数据处理中的降维;
(2) PCA 分析作图;
(3) 三种常见的聚类分析:K-means、层次分析与 SOM
(4) 热图和 hcluster 图的 R 语言实现;
C2 一组代谢组学数据的降维与聚类分析的 R 演练
(1) 数据解析;
(2) 演练与操作;
C3 有监督式机器学习在代谢组学数据处理中的应用
(1) 数据用 PCA 降维处理后仍然无法找到差异怎么办?
(2) PLS-DA 找出最可能影响差异的代谢物;
(3) VIP score 和 coef 的意义及选择;
(4) 分类算法:支持向量机,随机森林
C4 一组代谢组学数据的分类算法实现的 R 演练
(1) 数据解读;
(2) 演练与操作;
第四天
(1) 代谢组学中的 t、fold-change 和响应值;
(2) 数据清洗流程;
(3) R 语言 tidyverse
(4) R 语言正则表达式;
(5) 代谢组学数据过滤;
(6) 代谢组学数据 Scaling 原理与 R 实现;
(7) 代谢组学数据的 Normalization;
(8) 代谢组学数据清洗演练;
D2 在线代谢组分析网页 Metaboanalyst 操作
(1) 用 R 将数据清洗成网页需要的格式;
(2) 独立组、配对组和多组的数据格式问题;
(3) Metaboanalyst 的 pipeline 和注意事项;
(4) Metaboanalyst 的结果查看和导出;
(5) Metaboanalyst 的数据编辑;
(6) 全流程演练与操作
(1) Nature Communication 一篇代谢组学小鼠脑组织样本 database 类型的文献;
(2) Cell 一篇代谢组学患者血液样本的机器学习与疾病判断的文献;
(3) 1-2 篇代谢组学与转录组学和蛋白组学结合的文献。
E2 文献数据分析部分复现(1 篇)
(1) 文献深度解读;
(2)实操:从原始数据下载到图片复现;
(3) 学员实操。
案例图片:
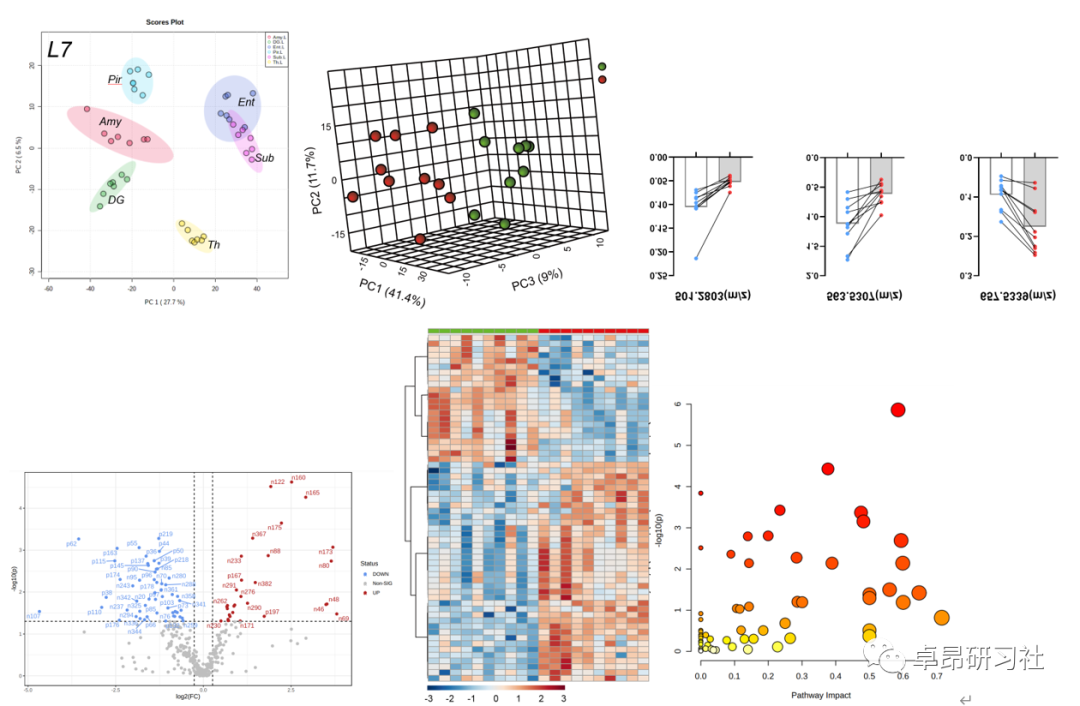
第一天上午
背景与理论知识以及工具准备
1.PDB数据库的介绍和使用
1.1数据库简介
1.2靶点蛋白的结构查询与选取
1.3靶点蛋白的结构序列下载
1.4靶点蛋白的下载与预处理
1.5批量下载蛋白晶体结构
2.Pymol的介绍与使用
2.1软件基本操作及基本知识介绍
2.2蛋白质-配体相互作用图解
2.3蛋白-配体小分子表面图、静电势表示
2.4蛋白-配体结构叠加与比对
2.5绘制相互作用力
3.notepad的介绍和使用
3.1 优势及主要功能介绍
3.2 界面和基本操作介绍
3.3插件安装使用
下午
一般的蛋白
-配体分子对接讲解
1.对接的相关理论介绍
1.1分子对接的概念及基本原理
1.2分子对接的基本方法
1.3分子对接的常用软件
1.4分子对接的一般流程
2.常规的蛋白-配体对接
2.1收集受体与配体分子
2.2复合体预构象的处理
2.3准备受体、配体分子
2.4蛋白-配体对接
2.5对接结果的分析
以新冠病毒蛋白主蛋白酶靶点及相关抑制剂为例
第二天
虚拟筛选
1.小分子数据库的介绍与下载
2.相关程序的介绍
2.1 openbabel的介绍和使用
2.2 chemdraw的介绍与使用
3.虚拟筛选的前处理
4.虚拟筛选的流程及实战演示
案例:筛选新冠病毒主蛋白酶抑制剂
5.结果分析与作图
6.药物ADME预测
6.1ADME概念介绍
6.2预测相关网站及软件介绍
6.3预测结果的分析
第三天
拓展对接的使用方法
1.蛋白-蛋白对接
1.1蛋白-蛋白对接的应用场景
1.2相关程序的介绍
1.3目标蛋白的收集以及预处理
1.4使用算例进行运算
1.5关键残基的预设
1.6结果的获取与文件类型
1.7结果的分析
以目前火热的靶点
PD-1/PD-L1等为例。
2.涉及金属酶蛋白的对接
2.1 金属酶蛋白-配体的背景介绍
2.2蛋白与配体分子的收集与预处理
2.3金属离子的处理
2.4金属辅酶蛋白-配体的对接
2.5结果分析
以人类法尼基转移酶及其抑制剂为例
3.蛋白-多糖分子对接
4.1蛋白-多糖相互作用
4.2对接处理的要点
4.3蛋白-多糖分子对接的流程
4.4蛋白-多糖分子对接
4.5相关结果分析
以
α-糖苷转移酶和多糖分子对接为例
5.核酸-小分子对接
5.1核酸-小分子的应用现状
5.2相关的程序介绍
5.3核酸-小分子的结合种类
5.4核酸-小分子对接
5.5相关结果的分析
以人端粒
g -四链和配体分子对接为例。
操作流程介绍及实战演示
第四天
拓展对接的使用方法
1.柔性对接
1.1柔性对接的使用场景介绍
1.2柔性对接的优势
1.3蛋白-配体的柔性对接
重点:柔性残基的设置方法
1.4相关结果的分析
以周期蛋白依赖性激酶
2(CDK2)与配体1CK为例
2.共价对接
2.1两种共价对接方法的介绍
2.1.1柔性侧链法
2.1.2两点吸引子法
2.2蛋白和配体的收集以及预处理
2.3共价药物分子与靶蛋白的共价对接
2.4结果的对比
以目前火热的新冠共价药物为例。
3.蛋白-水合对接
3.1水合作用在蛋白-配体相互作用中的意义及方法介绍
3.2蛋白和配体的收集以及预处理
3.3对接相关参数的准备
重点:水分子的加入和处理
3.4蛋白-水分子-配体对接
3.5结果分析
以乙酰胆碱结合蛋白
(AChBP)与尼古丁复合物为例
第五天
分子动力学模拟(linux与gromacs使用安装)
1. linux系统的介绍和简单使用
1.1 linux常用命令行
1.2 linux上的常用程序安装
1.3 体验:如何在linux上进行虚拟筛选
2.分子动力学的理论介绍
2.1分子动力学模拟的原理
2.2分子动力学模拟的方法及相关程序
2.3相关力场的介绍
3.gromacs使用及介绍
重点:主要命令及参数的介绍
4.origin介绍及使用
第六天
溶剂化分子动力学模拟的执行
1.一般的溶剂化蛋白的处理流程
2.蛋白晶体的准备
3.结构的能量最小化
4.对体系的预平衡
5.无限制的分子动力学模拟
6.分子动力学结果展示与解读
以水中的溶菌酶为例
第七天
蛋白-配体分子动力学模拟的执行
1.蛋白-配体在分子动力学模拟的处理流程
2.蛋白晶体的准备
3.蛋白-配体模拟初始构象的准备
4.配体分子力场拓扑文件的准备
4.1 高斯的简要介绍
4.2 ambertool的简要介绍
4.3生成小分子的力场参数文件
5.对复合物体系温度和压力分别限制的预平衡
6.无限制的分子动力学模拟
7.分子动力学结果展示与解读
8.轨迹后处理及分析
以新冠病毒蛋白主蛋白酶靶点及相关抑制剂为例
部分模型案例图片
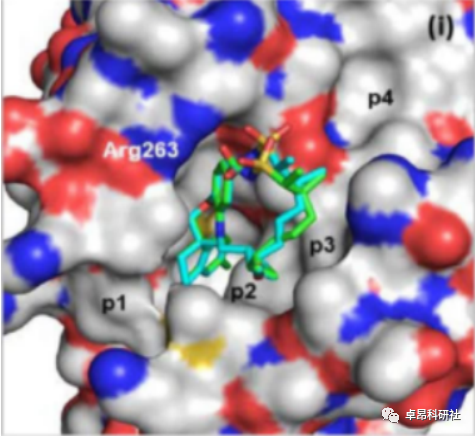
课程五、AIDD人工智能药物发现与设计专题(第一天)人工智能药物发现(AIDD)简介
机器学习和深度学习在药物发现领域的应用
工具的介绍与安装
1.人工智能药物发现(AIDD)简介2.机器学习和深度学习在药物发现领域的应用
1.2环境搭建
python
anaconda
工具包
RDKit
scikit-learn
pandas
numpy
(第二天)
机器学习
机器学习与药物发现
2.1 机器学习
2.1.1 随机森林Random Forest (RF)
2.1.2 支持向量机Support Vector Machines (SVMs)
2.1.3卷积神经网络
梯度下降
反向传播
随机梯度下降
学习率和激活函数
卷积神经网络CNN
常用框架介绍
Pytorch
TensorFlow
2.1.4机器学习任务
分类任务:classification
回归任务:regression
聚类任务:clustering
2.1.5机器学习验证和评估指标
验证:K折交叉验证K-fold cross validation
性能评估指标:
Sensitivity
Specificity
Accuracy
ROC-curve
AUC
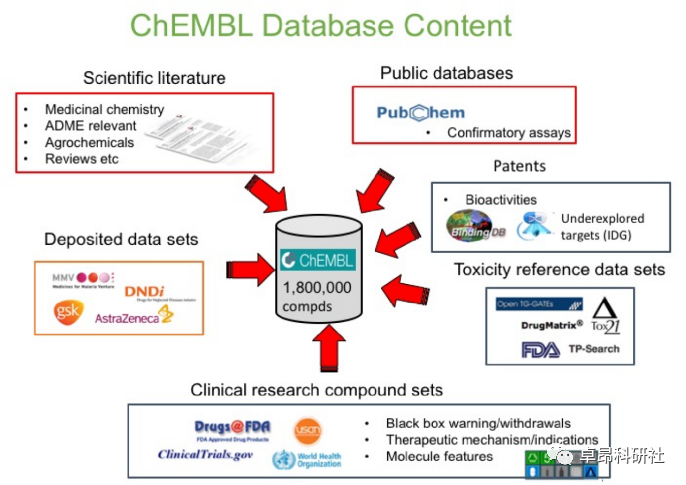
2.2 ChEMBL数据库介绍和使用
compound activity measures
IC50
pIC50
2.3 化合物的编码方式及化学相似性
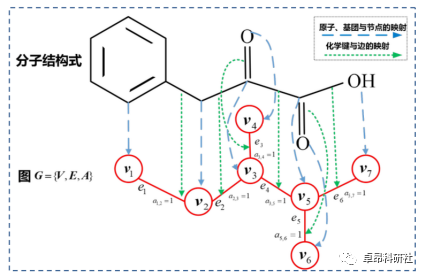
2.3.1 化合物编码方式
SMILES
InChI
Chebi
分子指纹
MACCS:Molecular ACCess System fingerprints (MACCS Keys)
Morgan Fingerprints:Extended-Connectivity Fingerprints (ECFPs)
2.3.2 化合物的化学相似性
Tanimoto 系数
Dice 系数
2.4 项目实战
2.4.1 Classification:基于分子指纹的化合物活性预测
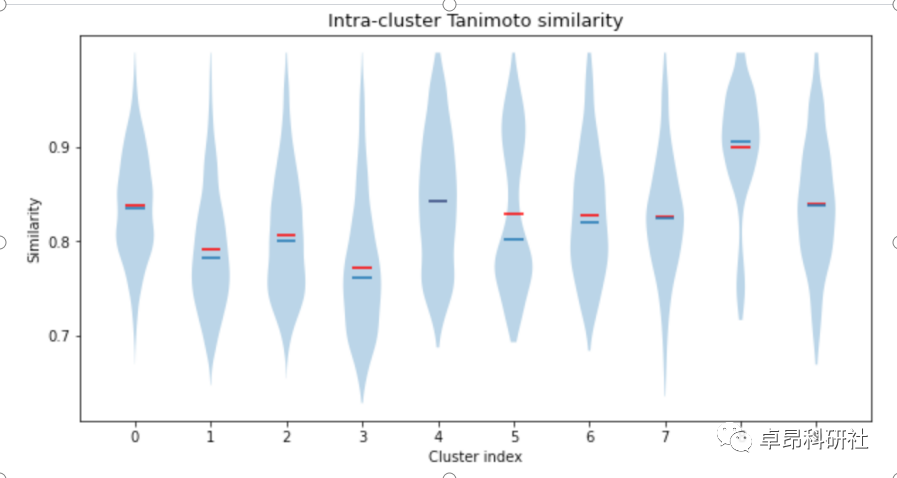
2.4.2 Clustering:基于Butina算法的分子聚类方法研究
(第三天)
图神经网络与药物发现
3.1 图神经网络
图卷积网络 GCN
图注意力网络 GAN
图同构网络 GIN
常用框架介绍
Pytorch_Geometric
DGL
3.2 分子毒性简介与相关数据集介绍
Tox21
ToxCast
ClinTox
3.3 项目实战:基于图神经网络的分子毒性预测
3.4 经典论文讲解:DeepTox: Toxicity Prediction using Deep Learning
(第四天)
自然语言处理与药物发现
4.1 自然语言处理
循环神经网络 RNN
LSTM
Seq2seq
Transformer
常用框架介绍:
Pytorch
TensorFlow
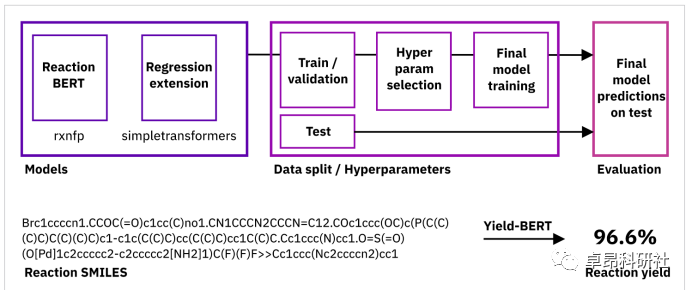
4.2 有机反应产量简介及相关数据集
4.2.1 有机反应的表示方法
4.2.2 有机反应的产量
4.2.3 有机反应相关数据集 USPTO
4.3 项目实战:基于Transformer的有机化学反应产量预测
4.4 经典论文解读:Extraction of organic chemistry grammar from unsupervised learning of chemical reactions
(第五天)
生化代谢路径设计与药物发现
5.1 生化数据集介绍与使用
KEGG
BiGG
BioCyc
PubChem
Chebi
5.2 搜索方法
基于化学计量矩阵的搜索方法
基于逆合成的搜索方法
基于图结构的搜索方法
基于进化算法的搜索方法
5.3 评估方法
通量平衡分析FBA
理论产量计算
热力学可行性分析
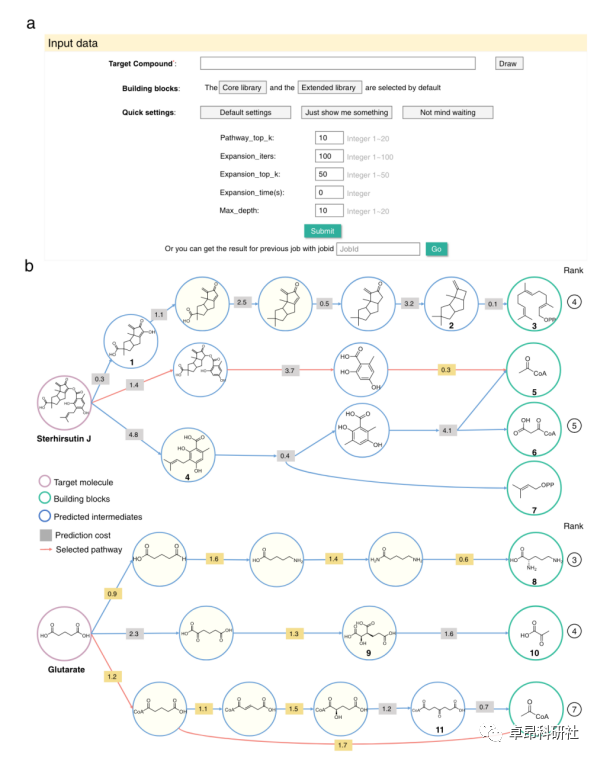
5.4 项目实战:基于逆合成的生物代谢路
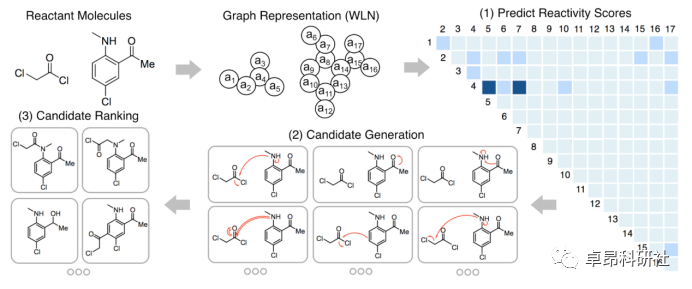
5.5 经典论文讲解:Predicting Organic Reaction Outcomes with Weisfeiler-Lehman Network
赠送视频:深度学习AiphaFold2蛋白质结构预测实例讲解
(以下内容为赠送的视频教程)
蛋白质结构与功能的概述。
蛋白质的组成
蛋白质的结构
蛋白质的功能
常见蛋白质结构预测的网站及方法。
常用蛋白质结构预测的相关网站及软件
常用网站及软件的使用方法及说明
机器学习在蛋白质结构预测的应用。
蛋白质结构与小分子药物库获取
机器学习加速预测小分子药物
AlphaFold2机器学习模型对蛋白结构预测
实战蛋白结构预测目前最好的人工智能模型AlphaFold2。
AlphaFold2模型的获取及安装
AlphaFold2相关数据的获取
AlphaFold2模型的实战操作
案例图片:


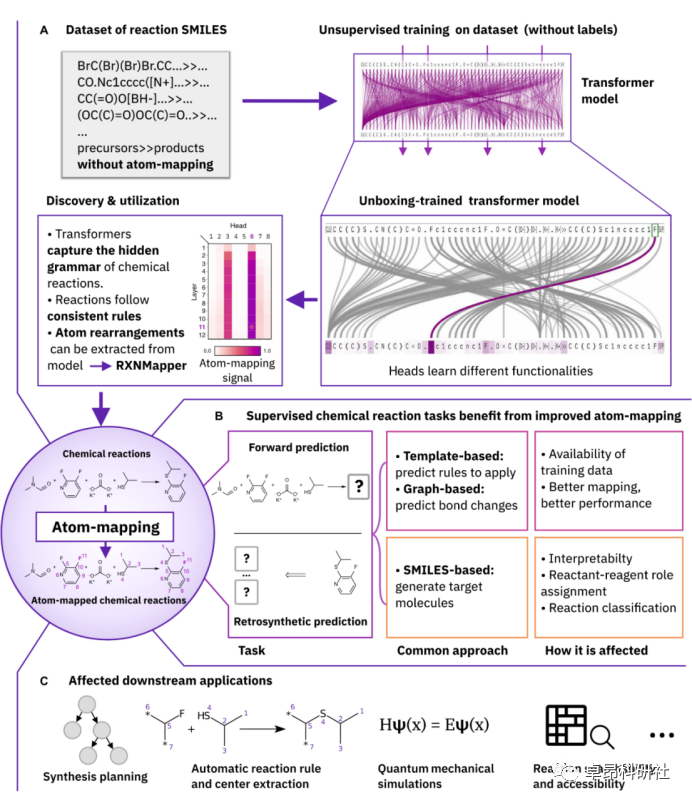
第一天
理论部分
深度学习算法介绍
1.有监督学习的神经网络算法
1.1全连接深度神经网络DNN在基因组学中的应用举例
1.2卷积神经网络CNN在基因组学中的应用举例
1.3循环神经网络RNN在基因组学中的应用举例
1.4图卷积神经网络GCN在基因组学中的应用举例
2.无监督的神经网络算法
2.1自动编码器AE在基因组学中的应用举例
2.2生成对抗网络GAN在基因组学中的应用举例
实操内容
1.Linux操作系统
1.1常用的Linux命令
1.2 Vim编辑器
1.3基因组数据文件管理, 修改文件权限
1.4查看探索基因组区域
2.Python语言基础
2.1.Python包安装和环境搭建
2.2.常见的数据结构和数据类型
第二天
理论部分
基因组学基础
1.
基因组数据库
2. 表观基因组
3. 转录基因组
4. 蛋白质组
5. 功能基因组
实操内容
基因组常用深度学习框架
1. 安装并介绍深度学习工具包tensorflow, keras,pytorch
2. 在工具包中识别深度学习模型要素
2.1.数据表示
2.2.张量运算
2.3.神经网络中的“层”
2.4.由层构成的模型
2.5.损失函数与优化器
2.6.数据集分割
2.7.过拟合与欠拟合
3.基因组数据处理
3.1安装并使用keras_dna处理各种基因序列数据如BED、 GFF、GTF、BIGWIG、BEDGRAPH、WIG等
3.2使用keras_dna设计深度学习模型
3.3使用keras_dna分割训练集、测试集
3.4使用keras_dna选取特定染色体的基因序列等
4.深度神经网络DNN在识别基序特征中应用
4.1实现单层单过滤器DNN识别基序
4.2实现多层单过滤器DNN识别基序
4.3实现多层多过滤器DNN识别基序
第三天
理论部分
卷积神经网络CNN在基因调控预测中的应用
1. Chip-Seq中识别基序特征G4,如DeepG4
2. Chip-Seq中预测DNA甲基化,DeepSEA
3. Chip-Seq中预测转录调控因子结合,DeepSEA
4. DNase-seq中预测染色体亲和性,Basset
5. DNase-seq中预测基因表达eQTL,Enformer
实操内容
复现卷积神经网络CNN识别基序特征DeepG4、非编码基因突变DeepSEA
,预测染色体亲和性Basset,基因表达eQTL
1. 复现DeepG4从Chip-Seq中识别G4特征
2. 安装selene_sdk,复现DeepSEA从Chip-Seq中预测DNA甲基化,非编码基因突变
3. 复现Basset,从Chip-Seq中预测染色体亲和性
4. 复现Enformer,从Chip-Seq中预测基因表达eQTL
第四天
理论部分
深度学习在识别拷贝数变异DeepCNV、调控因子DeepFactor上的应用
1. SNP微阵列中预测拷贝数变异CNV,DeepCNV
2. RNA-Seq中预测premiRNA,dnnMiRPre
3. 从蛋白序列中预测调控因子蛋白质,DeepFactor
实操内容
1. 复现DeepCNV利用SNP微阵列联合图像分析识别拷贝数变异
2. 复现循环神经网络RNN工具 dnnMiRPre,从RNA-Seq中预测premiRNA
3. 复现DeepFactor,从蛋白序列中识别转录调控因子蛋白质
第五天
理论部分
深度学习在识别及疾病表型及生物标志物上的应用
1. 从基因表达数据中识别乳腺癌分型的深度学习工具DeepType
-
2. 从高维多组学数据中识别疾病表型,XOmiVAE
3. 基因序列及蛋白质相互作用网络中识别关键基因的深度学习工具DeepHE
实操内容
1. 复现DeepType,从METABRIC乳腺癌数据中区分乳腺癌亚型
2. 复现XOmiVAE,从TCGA多维数据库中识别乳腺癌亚型
3. 复现DeepHE利用基因序列及蛋白质相互作用网络识别关键基因
第六天
理论部分
深度学习在预测药物反应机制上的应用
1. 联合肿瘤基因标记及药物分子结构预测药物反应机制的深度学习工具SWnet
实操内容
1. 预处理药物分子结构信息
2. 计算药物相似性
3. 在不同数据集上构建self-attention SWnet
4. 评估self-attention SWnet
5. 构建多任务的SWnet
6. 构建单层SWnet
7. 构建带权值层的SWnet
案例图片:
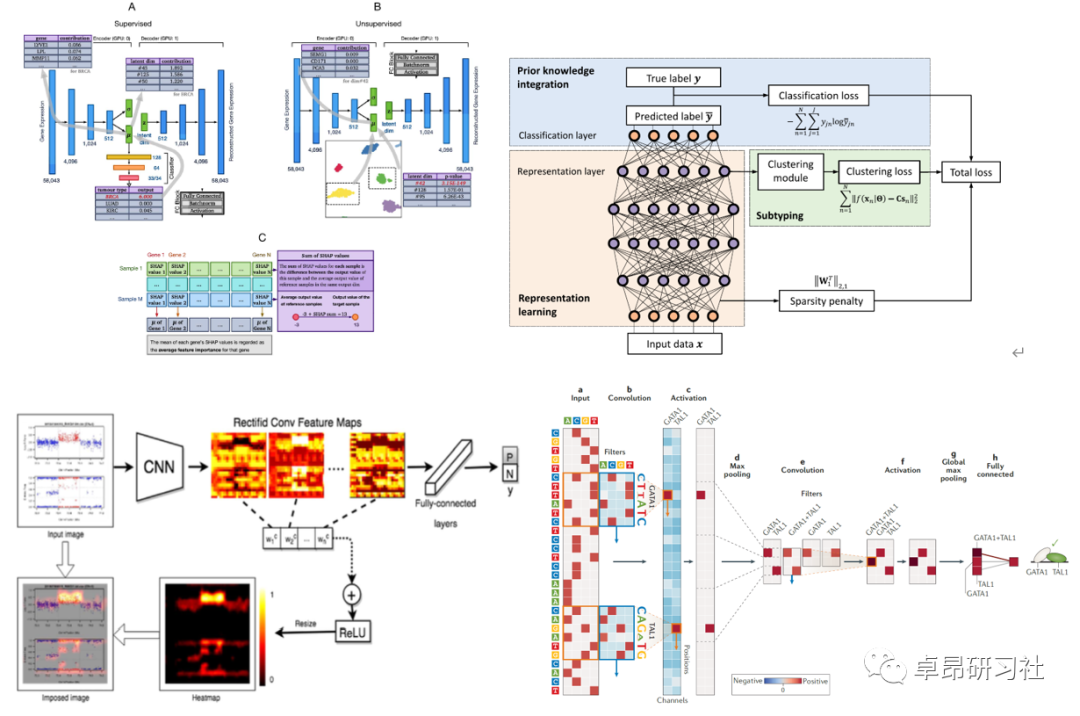
第一天
理论部分
1.单细胞测序原理
2.单细胞测序基础
3.单细胞测序方法及数据
4.单细胞数据分析流程
实操内容
1.R语言基础
2.R(4.1.3)和Rstudio的安装
3.R包安装和环境搭建
4.数据结构和数据类型
5.R语言基本函数
6.数据下载
7.数据读入与输出
第二天
理论部分
1.机器学习概述
2.线性模型
3.决策树
4.支持向量机
5.集成学习
6.模型选择与性能优化
实操内容
1.决策树算法实现
2.随机森林算法实现
3.支持向量机(SVM)算法实现
4.朴素贝叶斯算法实现
5.Xgboost算法实现
6.主成分分析PCA算法实现
7.聚类算法实现
8.DBSCAN算法实现
9.层次聚类算法实现
第三天
理论部分
1.多组学基础
2.常用生物组学实验与分析方法
3.常用组学数据库介绍
4.批量处理组学数据
5.生物功能分析
6.基于转录组学的差异基因筛选,疾病预测
7.基于差异基因联合多组学分析疾病发生机制
8.组学数据可视化
实操内容
1.Linux操作系统
2.Linux操作系统的安装与设置
3.网络配置与服务进程管理
4.Linux的远程登录管理
5.常用的Linux命令
6.在Linux下获取基因数据
7.利用Linux 探索基因组区域
8.Shell script与Vim编辑器
9.基因组文件下载与上传
10.Linux权限管理
11.文件的身份
12.修改文件的所有者和所属组
13.修改文件权限
第四天
理论部分
1.单细胞分析中的常见机器学习方法
2.降维聚类的机器学习算法
3.分群注释的机器学习算法
4.单细胞分析中常见的深度学习方法
5.降维聚类的深度学习算法
6.分群注释的深度学习算法
实操内容
1.Python语言基础
2.python安装与开发环境的搭建
3.基本数据类型组合数据类型
4.分析环境搭建
5.Jupyter notebook的使用
6.函数、列表、元组、字典、集合
7.控制结构、循环结构
8.Numpy模块——矩阵的科学计算
9.Matplotlib模块——数据处理与绘图
10.Pandas模块——csv数据处理与分析
11.Sklearn模块——
机器学习模型基础软件包调用
第五天
理论部分
1.机器学习在单细胞分析中的应用
2.收集数据
3.数据准备
4.选择一个模型
5.模型训练
6.模型评估
7.参数调整
8.模型预测
实操内容
1.创建Seurat对象
2.数据质控
3.测序深度差异及标准化
4.单细胞数据降维
5.批次效应去除
6.数据整合
7.亚群注释
8.GSVA通路活性分析
9.单细胞富集分析
第六天
理论部分
1.深度学习在单细胞分析中的应用
2卷积神经网络基本构成
3.卷积核
4.循环神经网络基本构成
5.循环核
6.图神经网络基本构成
实操内容
1.卷积神经网络在单细胞分析的算法实现
2.循环神经网络在单细胞分析的算法实现
3.图神经网络在单细胞中分析的算法实现
4.单细胞数据的自动注释
5.单细胞数据分析聚类与批量效应
6.单细胞测序数据进行聚类
7.加权图神经网络的细胞类型注释方法
8.单细胞表达数据中深度学习基因关系
案例图片:
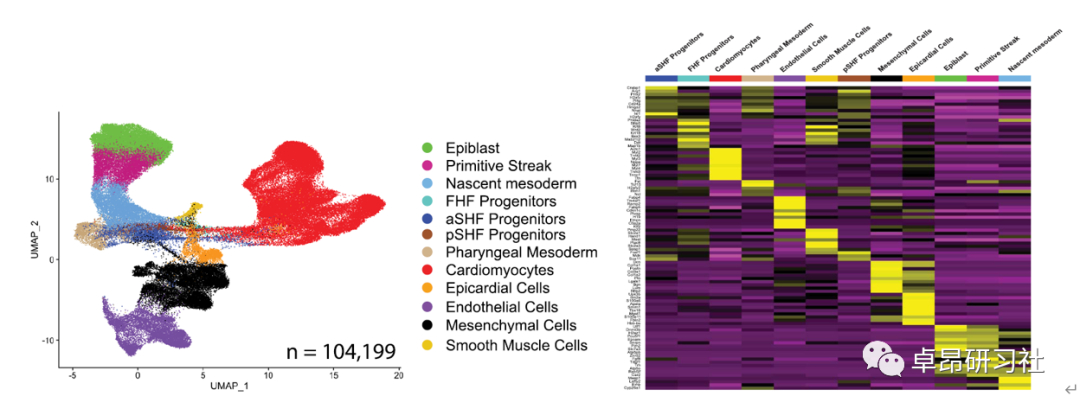
理论内容:
1.单细胞组学研究简介
2.单细胞转录组测序技术进展及其原理:1992\2009-至今
3.单细胞多组学和空间转录组技术;
4.单细胞转录组测序技术的常见应用和重要生物学发现;
5.单细胞重大项目及数据库介绍。
实操内容:
1. Linux 命令入门讲解及实操训练。
2. R 语言简介及安装。
3. R 语言简单语法及常见命令。
4. 数据挖掘及其统计应用。
5. R 语言实操画图 ggplot2 为主。
理论内容:
1. 单细胞实验介绍,常见建库结构。
2. 单细胞转录组 Pipeline 软件和代码介绍。
3. 单细胞转录组转录因子及其细胞通讯介绍。
4. 单细胞组学在肿瘤、发育、免疫及其它等领域的研究思路。
实操内容:
1. 10X 官方单细胞软件 Cellranger 讲解及实操。
2.质控基因和细胞。
3.选取高可变基因。
4.降维与分群。
5.Biomarker 定义细胞类型。
6.寻找差异基因
7. 通过 Seurat 合并多样本及消除样本异质性。
单细胞转录组轨迹、通路、转 录因子等分析及绘图实操
实操内容;
1. 通过 Monocle 软件对单细胞转录组进行拟时序的分析。
2. 对单细胞各个簇进行通路的功能富集分析。
3. 通过 GSVA 给细胞通路打分等。
4. 利用 cellphonedb 软件对细胞互作进行分析。
5. 通过典型的生信软件 infercnv 对染色体拷贝数变异进行推断。
6. 全面解析 SCENIC 软件进行转录因子预测分析。
理论内容:
1. 空间转录组技术的介绍。
2. 空间转录组技术的应用。
3. 空间转录组文章图表的解读。
4. 空间转录组技术在癌症、发育、神经科学等领域的研究思路。
空间转录组数据比对、降维以 及聚类等分析 空间转录组多样本及与但单细 胞数据关联分析实操内容:
1. 10x Visium 组织优化及文库制备。
2. 10x Visium 官方分析软件 Space Ranger 讲解及实操。
3. Space Ranger 输出结果解读。
4. Loupe Browser 软件安装及使用。
5. 通过 Seurat 软件进行降维、聚类和可视化。
6. 通过 Seurat 进行基因表达可视化。
理论+实操内容
1. 通过 Seurat 进行空间变量特征的识别。
2. 与单细胞数据关联分析(空间细胞类型定义)
3. 通过 Seurat 处理多个切片。
4. 单细胞及空间转录组数据分析总结。
机器学习蛋白组学与机器学习微生物组学主讲老师来自国内高校李老师授课,有十余年的微生物组数据分析经验。研究领域涉及机器学习,芯片数据分析,微生物组数据分析,DNA,RNA,甲基化测序数据分析,单细胞测序数据分析,miRNA及靶基因分析等,发表SCI论文30余篇,其中一作及并列一作15篇。
机器学习代谢组学 985高校神经科学博士,主要利用代谢组学、转录组学和分子生物学等技术研究神经内科慢性病的发病机制和生物标志物。擅长高效液相色谱-质谱联用(LC-MS)技术进行非靶向和靶向代谢组学从样本制备到数据分析的全流程研究,以及多组学大数据的生物信息学整合分析。5年内在J Clin Invest, EBioMedicine, Cell Death Dis, Cell Death Discov, Nanotoxicology等杂志发表SCI论文10篇。
CADD主讲老师来自国内高校、中科院等单位,老师主要擅长深度学习、机器学习、药物虚拟筛选、计算机辅助药物设计、人工智能药物发现、分子对接、分子动力学等方面的研究,在我们单位长期进行授课,讲课内容和授课方式以及敬业精神受到参会学员 的一致认可和高度评价
AIDD授课老师余老师,有十余年的计算机算法研究和程序设计经验。研究方向涉及生物信息学,深度学习,药物靶标识别,药物不良反应等。参与了国自然基金2项,主持了省厅级科研项目3项。一作身份发表SCI论文数篇,包括BMC Bioinformatics, Journal of Biomedical Informatics, International Journal of Molecular Sciences等知名期刊。
深度学习基因组学主讲老师刘老师,生物信息学博士,有十余年的测序数据分析经验。研究领域涉及人工智能、自然语言处理、功能基因组学、转录组学、miRNA及靶基因网络分析,单细胞测序数据分析,基因调控网络时序分析,蛋白质互作网络分析,多组学联合分析等。主持省自然科学基金等项目4项,出版医学实用教材《Python医学实战分析》,发表SCI论文22篇,其中一作及并列一作9篇。
机器学习单细胞主讲老师来自国内高校陈老师和张老师授课。在国内外学术刊物发表论文数篇,包括Nature Communication, Cell Regeneration等知名期刊,研究方向为生物信息学,发育生物学和遗传学等。利用多组学数据,通过深度学习算法进行数据分析和挖掘,包括ChIP-seq,ATAC-seq,RNA-seq,CNV等。
全国各大高校、企业、科研院所从事人工智能、生命科学、代谢工程、有机合成、天然产物、药物、生物信息学、植物学,动物学、化学化工,医学、基因组学、农业科学、植物学、动物学,临床医学、食品科学与工程、肿瘤免疫与靶向治疗、 全基因组泛癌分析、人黏连蛋白折叠基因组机、病毒检测、功能基因组、遗传图谱、基因挖掘变异、代谢组学、蛋白质组学、转录组学、生物医学、癌症、核酸、毒物学等研究科研人员及爱好者
1、课程特色--全面的课程技术应用、原理流程、实例联系全贯穿
2、学习模式--理论知识与上机操作相结合,让零基础学员快速熟练掌握
3、课程服务答疑--主讲老师将为您实际工作中遇到的问题提供专业解答
福利:报名缴费成功赠送报名班型全套预习视频,课后学习完毕提供全程录像视频回放,针对与培训课程内容进行长期答疑,微信解疑群永不解散,参加本次课程的学员可免费再参加一次本单位后期组织的相同的专题培训班(任意一期都可以)
授课方式:通过腾讯会议线上直播,理论+实操的授课模式,老师手把手带着操作,从零基础开始讲解,电子PPT和教程开课前一周提前发送给学员,所有培训使用软件都会发送给学员,有什么疑问采取开麦共享屏幕和微信群解疑,学员和老师交流、学员与学员交流,培训完毕后老师长期解疑,培训群不解散,往期培训学员对于培训质量和授课方式一致评价极高!
学员对培训非常认可,我们也保证二次学习是免费的
机器学习微生物组学培训班
2023.4.22 ----- 全天授课(上午09.00-11.30 下午13.30-17.00)
2023.4.24 -----4.27晚上授课 (晚上19.00-22.00)
2023.4.29-----2023.4.30 全天授课(上午09.00-11.30 下午13.30-17.00)
机器学习蛋白质组学培训班
2023.5.7全天授课(上午09.00-11.30 下午13.30-17.00)
20235.8 -----2023.5.11晚上授课(晚上19.00-22.00)
2023.5.13----2023.5.14 全天授课(上午 09.00-11.30 下午13.30-17.00)
机器学习代谢组学培训班
2023.4.22 -----2023.4.23 全天授课(上午09.00-11.30 下午13.30-17.00)
2023.5.6-----2023.5.7全天授课(上午09.00-11.30 下午13.30-17.00)
2023.5.10 -----2023.5.11晚上授课 (晚上19.00-22.00)
CADD计算机辅助药物设计专题培训班
2023.4.22 -----2023.4.23 全天授课(上午09.00-11.30 下午13.30-17.00)
2023.4.25-----2023.4.28晚上授课(晚上19.00-22.00)
2023.5.6-----2023.5.7全天授课(上午09.00-11.30 下午13.30-17.00)
2023.5.8 -----2023.5.9晚上授课 (晚上19.00-22.00)
AIDD人工智能药物发现与设计专题培训班
2023.5.6-----2023.5.7 全天授课(上午 09.00-11.30 下午 13.30-17.00)
2023.5.9----2023.5.10 晚上授课(晚上 1 9.00-22.00)
2023.5.13----2023.5.14 全天授课(上午 09.00-11.30 下午 13.30-17.00)
深度学习在基因组学培训时间
2023.5.6-----2023.5.7全天授课(上午09.00-11.30 下午13.30-17.00)
2023.5.8 -----2023.5.9
晚上授课 (晚上19.00-22.00)
2023.5.11——2023.5.12晚上授课 (晚上19.00-22.00)
2023.5.13----2023.5.14 全天授课(上午 09.00-11.30 下午 13.30-17.00)
机器学习单细胞培训时间
2023.4.22 -----2023.4.23 全天授课(上午09.00-11.30 下午13.30-17.00)
2023.5.6-----2023.5.7 全天授课(上午09.00-11.30 下午13.30-17.00)
2023.5.13----2023.5.14 全天授课(上午 09.00-11.30 下午 13.30-17.00)
单细胞空间转录组培训时间
2023.4.22 -----2023.4.23 全天授课(上午09.00-11.30 下午13.30-17.00)
2023.5.7 全天授课(上午09.00-11.30 下午13.30-17.00)
2023.5.8-----2023.5.9 晚上授课 (晚上19.00-22.00)
CADD计算机辅助药物设计;AIDD人工智能药物发现;深度学习基因组学;机器学习单细胞
公费价:每人每班¥5880元 (含报名费、培训费、资料费)
自费价:每人每班¥5480元 (含报名费、培训费、资料费)
单细胞空间转录组;机器学习蛋白质组学;机器学习微生物组学;机器学习代谢组学
公费价:每人每班¥4680元 (含报名费、培训费、资料费)
自费价:每人每班¥4280元 (含报名费、培训费、资料费)
优惠
优惠1:两班同报:9880元 三班同报:13880元 四班同报:17880元 五班同报:20000元
优惠2:提前报名缴费学员+转发到朋友圈或者到学术交流群可享受每人300元优惠(仅限15名)
优惠3:同时报名两个班免费赠送一个学习名额(赠送班任选)
优惠4: 报名五个培训班以上,免费赠送三个培训名额(赠送班任选)
证书:参加培训并通过考试的学员,可以申请获得工业和信息化部工业文化发展中心颁发的“工业强国建设素质素养提升尚工行动”岗位能力适应评测证书。该证书可在中心官网查询,可作为能力评价,考核和任职的重要依据。评测证书查询网址:www.miit-icdc.org(自愿申请,须另行缴纳考试费500元/人)

联系人:范老师
电话:15036025979(微信同号)
发现真的是脚踏实地的同时 需要偶尔仰望星空非常感谢各位对我们培训的认可!祝愿各位心想事成!