
目前,许多深度学习方法已经被提出来预测表观基因组、染色质结构组织和基因转录。虽然这些方法在使用一种模态预测另一种模态方面取得了令人满意的性能,但学习到的表征不能在预测任务或细胞类型之间推广或运用。2023年5月24日,美国密歇根大学医学院Jie Liu教授在Nucleic Acids Research上发表了题为A generalizable framework to comprehensively predict epigenome, chromatin organization, and transcriptome 的研究论文。
在该研究中,作者开发了一个名为EPCOT 的深度学习模型并且使用了预训练 (pre-training) 和微调 (fine-tuning) 的框架全面的预测了表观遗传学特征 (epigenomic feature)、基因表达 (gene expression)、染色质结构组织 (chromatin organization) 和增强子活性 (enhancer activity)。该模型可以扩展到其他细胞或组织类型,并且只需要染色质可及性 (chromatin accessibility) 数据。作者运用了包括ENCODE,GTEx 和 4DN数据库,并且和之前的预测模型和方法做比较来证明了EPCOT在跨细胞类型预测上的准确性。另外,作者还开发了网页 (https://liu-bioinfo-lab.github.io/EPCOT_APP.github.io/) 来帮助研究人员使用训练好的模型去预测多种基因组模态,该工具只需要用户上传一个ATAC-seq文件。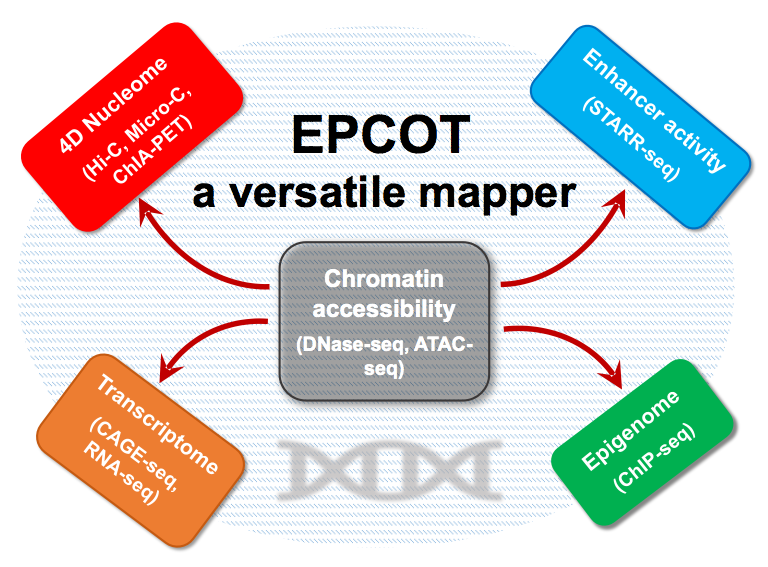
在该研究中,作者还通过模型学习到了一个通用的序列表征 (sequence representations),可以在所有的预测任务中得到较好的预测表现,揭示了计算机可以使用一种通用的语言来编码DNA序列。密歇根大学Liu Lab博士生张振豪为第一作者,Jie Liu教授为论文的通讯作者。原文链接:
https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad436/7177889?searchresult=1
制版人:十一

【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。