我们直接开门见山好了,第一件事:什么是t-SNE?t-SNE的全称叫做t分布式随机邻居嵌入(t-SNE)。该算法是一种非监督的非线性技术,主要用于数据探索和可视化高维数据。简而言之,t-SNE为我们提供了数据如何在高维空间中排列的感觉或直觉。它由Laurens van der Maatens和Geoffrey Hinton于2008年开发。一提到降维,我们会想到大名鼎鼎的PCA,PCA是线性降维的技术,那么较之于我们今天要介绍的t-SNE,它们有什么不同或者联系吗。如果您熟悉主成分分析(PCA),那么像我一样,你可能想知道PCA和t-SNE之间的区别。首先要注意的是,PCA是在1933年开发的,而t-SNE是在2008年开发的。自1933年以来,数据科学领域发生了很大变化,主要是在计算和数据大小领域。其次,PCA是一种线性降维技术,旨在最大化方差并保持较大的成对距离。PCA可能导致可视化效果不佳,特别是在处理非线性结构时。这里非线性结构可以视为任何几何形状,如:圆柱体、球、曲线等。t-SNE与PCA的不同之处在于只保留小的成对距离或局部相似性,而PCA则关注的是保持大成对距离以最大化方差。
图1-“瑞士卷“数据集,保持与t-SNE(实线)的小距离vs最大化方差PCALaurens很好地利用上图中的“瑞士卷”数据集很好地说明了PCA和t-SNE方法(实线为t-SNE,虚线为PCA)。你可以看到,由于这个“瑞士卷”数据集(流形)的非线性并保持了大距离,PCA会错误地保留数据的结构。
现在我们知道为什么有时候我们不用pca而用t-SNE,让我们来看看t-SNE是如何工作的,其背后有怎样的算法原理。其实,概括来说:t-SNE算法计算高维空间和低维空间中一对实例之间的相似性度量。然后,该算法用代价函数来优化这两个相似度指标。这个算法将其分为3个基本步骤。
步骤1:测量高维空间中点之间的相似性
我们以分散在二维空间上的一堆数据点(图2)说明。对于每个数据点(),我们将在该点上以高斯分布为中心。然后,我们测量高斯分布下所有点()的密度。然后对所有点进行重整化。我们根据高斯得出所有点的概率()。这些概率与相似之处成正比。这意味着,如果数据点()和()在这个高斯圆下具有相等的值,那么它们的比例和相似性是相等的,因此在这个高维空间的结构中具有局部相似性。高斯分布或圆可以使用所谓的复杂度来操作,这影响分布(圆大小)的方差,本质上影响最近邻居的数量。复杂度的正常范围在5到50之间。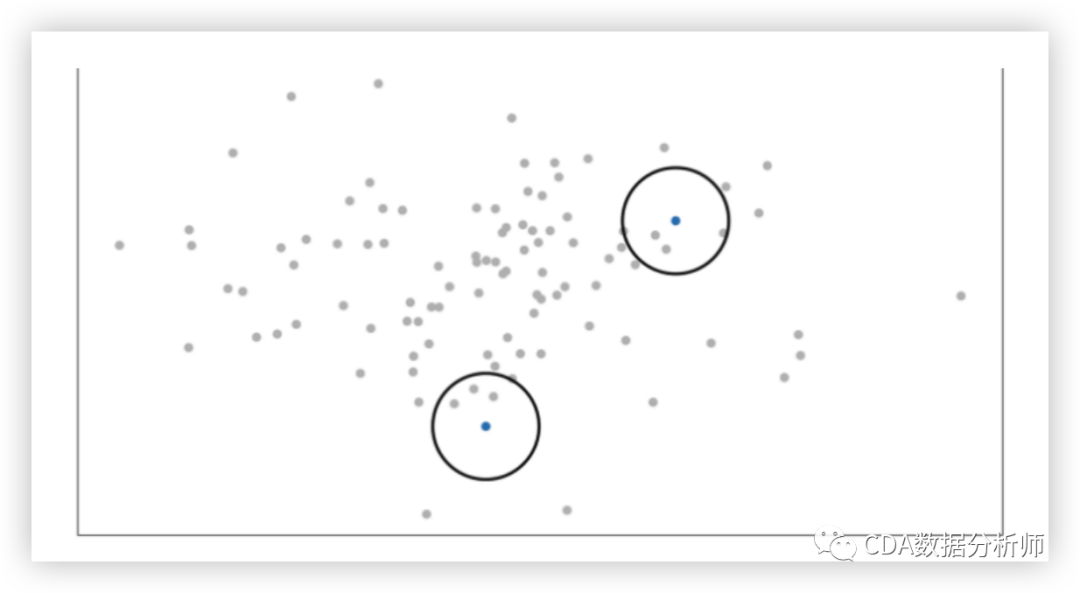
图2-围绕数据点的高斯分布在高维空间中测量成对的相似度
步骤2:第2步类似于第1步,不用高斯分布而是使用具有一定自由度的学生t分布T分布这也被称为柯西分布(图3)。这给了我们在低维空间中的第二组概率。如您所见,学生t分布的尾部比正态分布更“重”。沉重的尾部可以更好地模拟相距很远的距离。
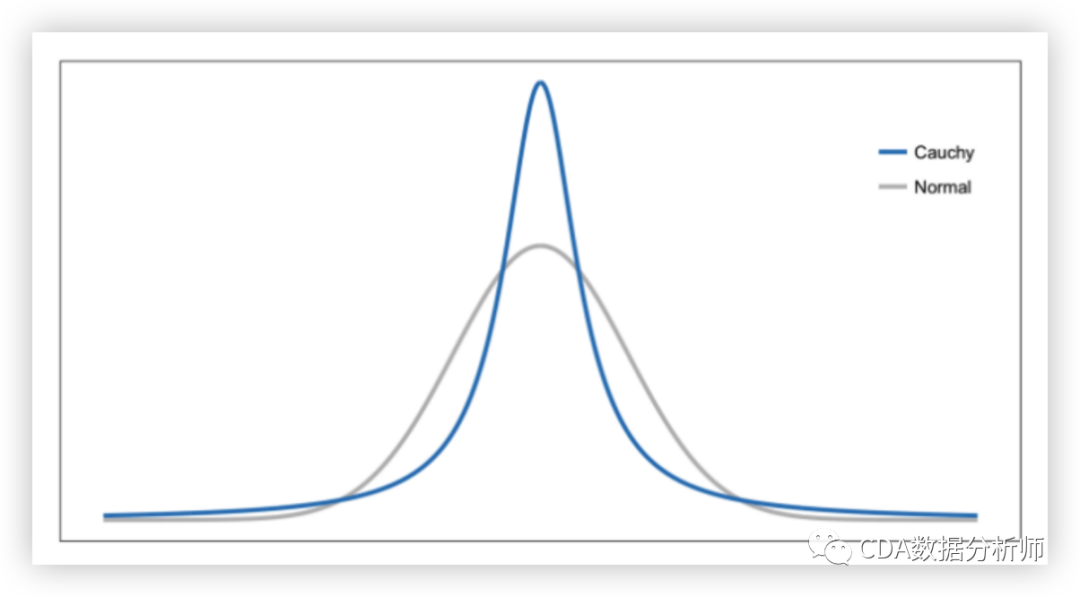
图3-柯西分布和正态分布概率密度图的比较
步骤3:最后一步是,我们希望这些来自低维空间()的概率集尽可能地反映高维空间()的概率我们希望这两个地图结构相似。我们使用库尔巴克-利布勒发散(KL)测量二维空间的概率分布之间的差异。可以有效地比较和值。最后,我们使用梯度下降来最小化我们的KL损失函数。现在我们已经了解了t-SNE的工作原理,让我们快速谈谈它在哪里使用。Laurens van der Maaten在他的视频演示中展示了许多例子,他提到了t-SNE在气候研究、计算机安全、生物信息学、癌症研究等领域的使用。也就是说t-SNE可用于高维数据(主要用于可视化),然后这些维度的输出成为其他分类模型的输入。然而,t-SNE不是聚类方法,因为它不保留PCA等输入,并且值可能经常在运行之间发生变化,因此纯粹是为了探索、可视化等工作。本次案例的目标是通过蘑菇的特征(比如形状、气味等)来区分其是否可以食用,同时会在二维空间上呈现基于PCA和t-SNE的不同的降维分类效果,以此来认识二者的不同。import matplotlib.pyplot as plt data = pd.read_csv('mushrooms.csv')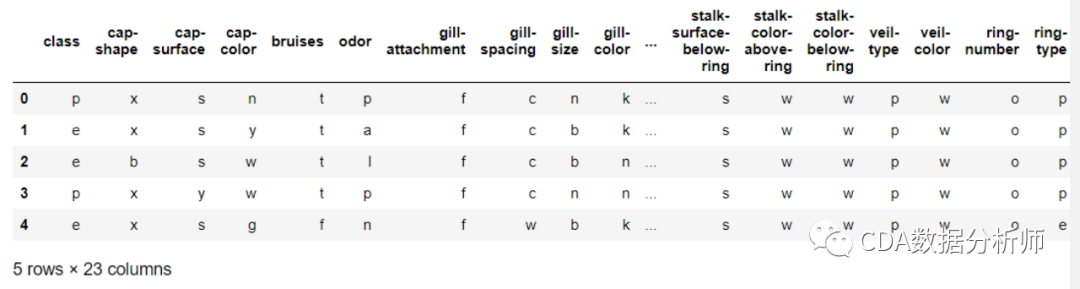
第2步-区分解释变量与被解释变量,查看数据的基本信息

cols = X.columns.tolist() print(col, X[col].unique())
X[col] = X[col].astype('category') X[col] = X[col].cat.codes
X.drop('veil-type', axis=1, inplace=True)from sklearn import preprocessingfrom sklearn.decomposition import PCAX_std = preprocessing.scale(X)X_pca=pd.DataFrame(pca.fit_transform(X_std)).rename(columns={0:'1st_component',1:'2nd_component'})data_pca = pd.concat([X_pca, Y], axis = 1)

plt.figure(figsize=(8, 8)) sns.scatterplot(data=data_pca, hue='class', x='1st_component', y='2nd_component') 
from sklearn.manifold import TSNEtsne = TSNE(n_components=2)X_tsne = pd.DataFrame(tsne.fit_transform(X_std)).rename(columns={0:'dim1', 1:'dim2'})data_tsne = pd.concat([X_tsne, Y], axis = 1)plt.figure(figsize=(8, 8)) sns.scatterplot(data=data_tsne, hue='class', x='dim1', y='dim2') 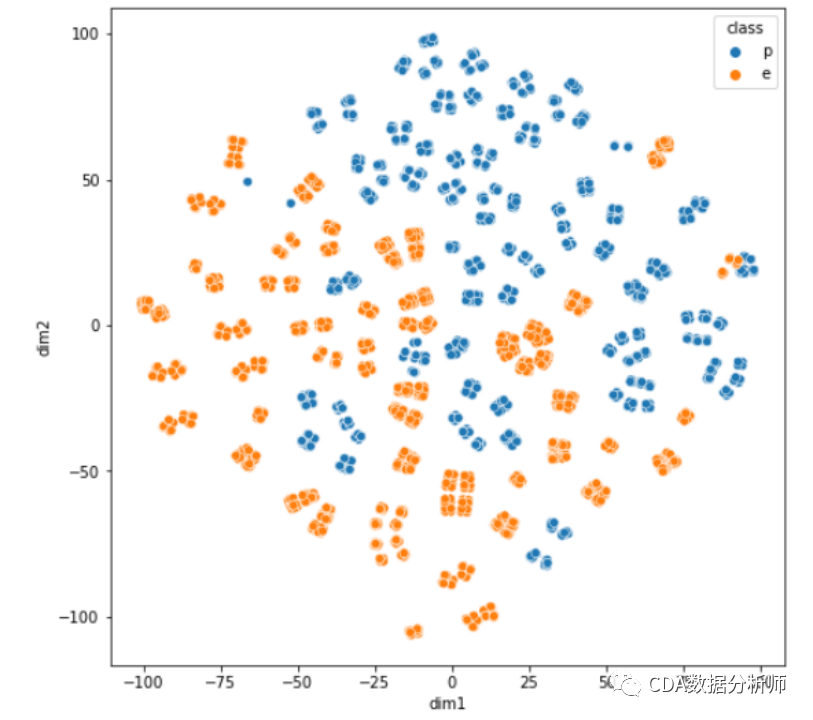
从可视化的结果可以看出,基于PCA降维的结果会产生重叠,这是因为主成分降维无法维护数据的局部结构而导致的,而基于t-SNE降维的结果分类更加清晰,基本没有类别之间的重叠,这就是t-SNE算法在降维过程中很好的保留了数据局部特征而产生的结果,所以,t-SNE算法可以很好的用来进行数据降维和可视化展示。