
这里是python3学习笔记的第七篇。小编也从蒙圈的状态开始写一些手推车脚本去完成一些文件的处理。今天的推送就贴上一个最近写的脚本,一定存在可以优化的点,也希望大家不吝赐教。
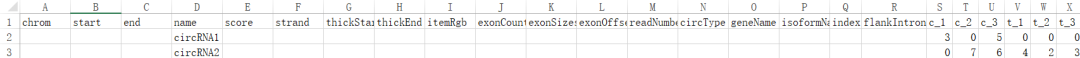
circRNA和lncRNA的分析依旧火热,生信预测软件也相对成熟,而且好用。伴随着研究的透彻,你也极有可能获得如上的表格,非常多的信息(为了展示本脚本想获取的有效信息,因此就简单的写了两行,右边展示为该分子在各个样本中比对到的reads数)。
在转录组分析中,我们会对不同样本(即生物学意义不同的组)进行分别测序,拟找到不同样本间差异表达的基因,也就是可能在该过程中发挥潜在功能的分子。表格信息太多就需要正确的姿势去获取。
贴代码:
import os
import re
os.chdir('C:\\Users\\user\\desktop')
f = open('expression.txt', 'rt')
f = f.readlines()
f3 = open('circRNA_sampleinfo.fa','w+')
dict_sample = {}
from itertools import islice
for line in islice(f, 1, None):
line = line.split('\t')
ids = line[3]
dict_sample[ids] = []
t_sample = int(line[-1]) + int(line[-2]) + int(line[-3])
c_sample = int(line[-4]) + int(line[-5]) + int(line[-6])
if t_sample >0:
dict_sample[ids] = ['T']
if m_sample > 0 :
dict_sample[ids].append('C')
f2 = open('circ.fa','rt')
for orf_line in f2:
if orf_line.startswith('>'):
orf_id = orf_line.split(':')[0]
circ_id = re.findall(r'_(.*)', orf_id)[0]
sample_info = ':'+str(dict_sample[circ_id])
orf_id1 = re.sub(r'$',sample_info,orf_line,1)
f3.write(orf_id1)
else:
f3.write(orf_line)f3.close()
代码分两段,放在pycharm上跑的,为了方便就将工作路径放在了桌面。区分样本主要依靠其中的判断,也就是上一期讲的条件判断。在此认为,在不同的生物重复中检测到我们将报这条circRNA在其中表达。
第二段是为了在fa文件中将样本特异信息写入其中的ID,如果单独只是获取组织特异表达的circRNA,只要参照第一段去修改,并不用引入字典,当然lncRNA等也类似。
如果你想整一个更有效的列表,在读行过程中,把其他列的信息一同写入。最后的最后,使用Venn图简单可视化。
这些是很简单的脚本啦,如果你想系统的学习、加入我们整理和分享的团队,或者是湿实验做多了想来看看生信的世界,亦或是出国深造打下基础、转行做分析,或者和团长面个基…
你现在有一个极好的机会。无论现在的水平如何,在你认定做这一件事的时候,将注定有所收获。不想了解一下么💁
生信技能树超级VIP入场券 (戳)
最后,专题的内容也整理放送,希望你会有所收获。
三剑客 PyCharm使用 | 编程基础与规范代码 | 列表使用一文就够了 | 元组拆包是个啥?| 字典与FASTA文件序列抽提 | 如何判断序列是否跨过剪切位点