师弟 Chuhao Li 用 Python 开发了第一个TBtools插件。插件的实用和制作的精细出乎意料。感慨之余,我邀请他分享一下制作经验(因为我完全不懂 Python,而我知道 Python 是现在绝大多数朋友最喜欢的编程语言)。收到推文,发现非常详细,且内容实用。尤为难得,还直接拓展了 TBtools 的一个功能痛点(没有批量化反向互补功能!)。Chuhao 开发的这个插件,直接填补了这个缺憾,同时还做了多线程加速,我用了下,非常方便,非常快!现在插件已经上传到商店,感兴趣的朋友可下载使用。另,欢迎大伙学习这篇博文后,也一起来开发插件,贡献力量,一起减除更多人的数据分析负担。- 陈程杰
前言
相信很多学习生物信息学的人都会学习python。你可能遇到过这样的情况:
在一个风和日丽的早上,你耗尽洪荒之力,写好了一个“反向互补”命令行工具,然后很激动地把代码发到群里面,分享给实验室其他人用。
小伙伴就会跟你说:这个脚本要怎么用呀?然后,你就会解释道:首先,安装一个python,然后这样打开终端,然后这样输入参数……。
小伙伴说:“嗯,我先试一下”。然后一顿操作,在不知名网站上下载了个“python”安装包,把python安装好,还付送了一堆4XX9小游戏之类的软件。
如无意外,过一会,小伙伴就会跑过来问你,“我输入命令,怎么报错了”?
你仔细地检查,发现如下问题:
安装了python2而不是python3
那行命令没有在特定工作目录
命令的某个单词少了一个字母
某两个参数之间没有空格
一顿操作过后,问题解决了,你还很详细地给小伙伴解释了其中的原理。能帮助到实验室的小伙伴,你很开心,回到工位上,准备迎接一天的工作。
然后,噩梦来了。另外一个小伙伴又来问你同样的问题……
这时候,我会告诉你:用pyinstaller打包你的工具,然后用TBtools的CLI Program Wrapper Creator来快速构建一个图形界面插件。
5分钟把插件打包好,直接把插件扔给小伙伴们,在群里说:“我项目需求,顺手写了个工具,有需要的拿去。” 然后潇洒离去。
下面我将用实际例子给大家介绍,如何操作。
python反向互补脚本
下面的脚本实现了单条序列的反向互补。为了减少代码量,我使用了biopython来进行fasta序列的读写和操作。接下来,我要把这个工具打包成TBtools插件。
import sysfrom Bio import SeqIO
infile = sys.argv[1]outfile = sys.argv[2]
seq = SeqIO.read(infile, 'fasta')seq.seq = seq.seq.reverse_complement()
with open(outfile, "w") as f: SeqIO.write(seq, f, "fasta")
安装pyinstaller
假设你已经安装好了python,进入cmd或者powershell,输入以下命令,即可安装pyinstaller以及脚本所依赖的biopython:
注意:如果你的python环境中已经安装了很多别的包,那么pyinstaller打包的时候会把一些不必要的包打包进来。这种情况下,建议使用virtualenv来新建一个新的环境(如果你是刚刚才安装的python,那就没必要做这一步了):
py.exe -m pip install virtualenv
py.exe -m virtualenv my_env
.\my_env\Scripts\activate
环境激活之后,安装所需的软件。这里除了pyinstaller,还安装了biopython,这是这个脚本中用到的。
py.exe -m
pip install pyinstaller biopython
一般来说,安装完后,在cmd或者powershell下运行下面的命令可以打印出帮助信息。
如果不行,那就试试下面的(注意大小写):
编写脚本需要注意的问题
参数传递
可以使用位置参数(参数前不带“-”的)和长参数(参数前带“--”的)。如果只有位置参数,就直接用sys.argv;如果要用到长参数,推荐使用argparse。
值得注意的是,当argparse和multiprocessing一起使用的时候,需要使用parser.parse_know_args(),而不能直接使用parser.parse_args(),否则传递参数的时候会出错。
多线程
使用到多线程时,主程序需要用if __name__ == '__main__':保护起来。下面一行紧接着freeze_support()。
调用其他工具
假如我的程序需要调用其他二进制文件,只需要把exe文件放在与脚本同一个目录下即可。TBtools运行脚本的时候,会自动把脚本所在目录添加到环境变量,于是在脚本中可以不需要指定二进制文件所在的路径。
调用外部命令,注意给输入、输入文件路径加引号,防止输入文件路径中有空格的问题。假设输入文件路径为infile = "C:\Use r\input.fasta",中间有个空格。那么,如果使用os.system(f'tool_name.exe {infile}')来调用就会有问题。需要在infile两边加上引号:os.system(f'tool_name.exe "{infile}"')。
值得注意的是,如果使用的是blastn.exe等TBtools已经有的二进制文件,则可以直接调用,不必打包到插件当中。
代码修改
下面的代码,在原来的基础上添加了多线程和使用argparse来解析参数。请注意注释部分。
import argparsefrom multiprocessing import Pool, freeze_supportfrom Bio import SeqIO
def rc(seq): seq.seq = seq.seq.reverse_complement() return seq
if __name__ == '__main__': freeze_support() parser = argparse.ArgumentParser() parser.description = "多线程反向互补" parser.add_argument("--threads", default=1, type=int, help="Number of threads") parser.add_argument("input", help="Input fasta file") parser.add_argument("output", help="Output fasta file") args, unparsed = parser.parse_known_args()
seqs = SeqIO.parse(args.input, 'fasta') with Pool(processes=args.threads) as pool: seqs_rc = pool.map(rc, seqs) pool.close() pool.join()
with open(args.output, "w") as f: for seq in seqs_rc: SeqIO.write(seq, f, "fasta")
打包
具体操作
在py脚本所在目录,按住“shift”键,然后右键点击文件浏览器的空白处,点击“在此处打开命令窗口”,就能进入终端,并自动切换工作目录为当前目录。
运行以下命令,即可打包:
pyinstaller -D .\multiprocess_rc.py
# 如果不行,就用下面的:# py.exe -m PyInstaller -D .\multiprocess_rc.py
打包完成,可以看到当前目录多了两个文件夹和一个文件:
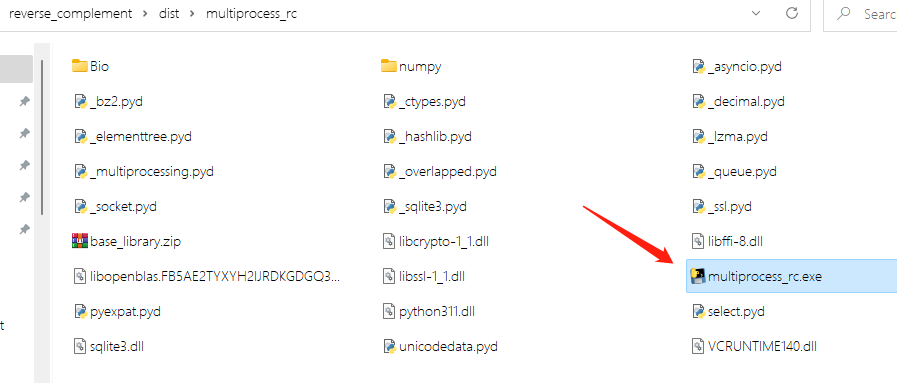
其中dist文件夹里面就是我们打包好的工具。里面长这样。里面的multiprocess_rc.exe就是程序的入口,其他文件是该程序依赖的资源,必须和主程序放在同一个目录。:
命令解析
在这个行命令中,我传递了-D和.\multiprocess_rc.py两个参数给pyinstaller。
-D 参数是指要打包成一个文件夹,把所有依赖的dll等文件放在同一个文件夹中。另外一种方式是-F,指定-F参数可以把全部文件打包成单个的exe文件。虽然-F参数打包出来的文件看上去很干净,其实在实际运行的时候还是会把exe文件里面的东西释放到一个临时文件夹中,然后再运行,而且会引入一些额外的bug。所以,我推荐使用-D参数。
.\multiprocess_rc.py 就是脚本的主程序。如果有多个模块,只需要输入主程序即可。打包工具会自动识别主程序中有import到的其他脚本,一起打包进去。
其他实用参数
如果你需要调用其他二进制文件,比如说tool.exe。你可以把tool.exe
放在和脚本同一目录,然后打包的时候增加以下参数:--add-data "tool.exe;tool.exe"。这样就可以把tool.exe一起放在打包好的文件夹中了。当然你也可以手动复制过去,不过如果涉及到多次打包,就会麻烦一丢丢。
输出文件
小技巧
常常会遇到需要打包、测试多次的时候。这时候,把打包和测试相关的命令写入到一个.bat文件中是比较方便的。这样直接双击.bat文件就可以重新打包,无需重新输入命令。
关于pyinstaller的更多信息,请参考:
pyinstaller官网
pyinstaller疑难问题解决
测试pyinstaller打包结果
进入dist/multiprocess_rc/目录,打开cmd,执行以下命令:
.\multiprocess_rc.exe --threads 4 .\input.fasta output.fasta
可以得到输出结果,代表运行成功。检查文件结果,也确实是反向互补了:
input.fasta:
>testATCGGGTTCCAA>test1ATCGGGTTCCAA>test2ATCGGGTTCCAA>test3ATCGGGTTCCAA>test4ATCGGGTTCCAA
output.fasta:
>testTTGGAACCCGAT>test1TTGGAACCCGAT>test2TTGGAACCCGAT>test3TTGGAACCCGAT>test4TTGGAACCCGAT
打包TBtools插件
在插件商店安装好CLI Program Wrapper Creator后,从Tbtools的菜单栏Others > Plugin中即可看到。点击进去,参照下方的设置:
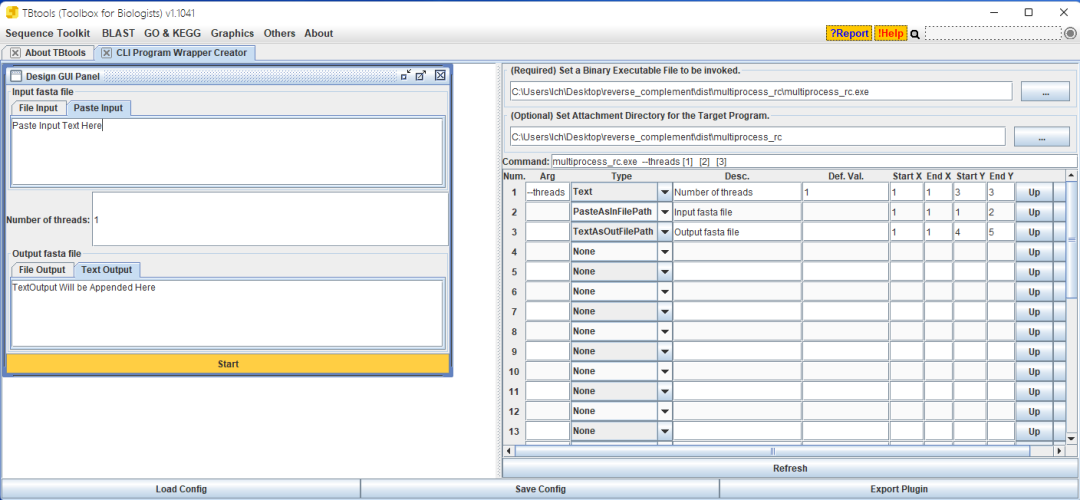
配置完成,点击“Refresh”就可以刷新界面。还可以直接在界面中运行程序,非常方便。如果配置到一半要去吃饭,可以按下方的“Save Config”暂时把界面设置保存为文件,下次可以直接用“Load Config”来加载,省去很多时间。
测试完成,点击右下角的“Export Plugin”,即可导出插件。导出的文件实际上是一个zip压缩文件。建议导出的时候不要加任何后缀,这样后面安装插件时,菜单栏上名字就会带有后缀。导出之后,再重新添加“.zip”后缀。
导出后,进入Others > Install Plugin 安装插件,然后就可以使用了。
把序列粘到Input输入框,点击开始,就可以看到下方的反向互补序列了!
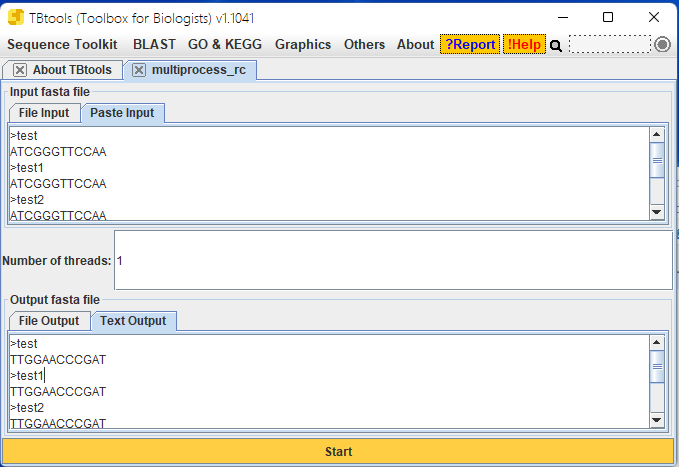
这里我忽略了很多UI设计界面使用的细节,详情可以回头看看公众号前几天的推文,有CJ的详细教程。
总结
把经常要做的、重复性的东西简单化、流程化、自动化,或许可以让大家把更多的时间投入到科学问题的研究中。