微生物的系统研究涉及从低通量的原位测序到高通量的质谱分析,而实验操作、样本来源和生物变异性会导致数据的差异性。机器学习为多组学数据的分析和预测提供了重要工具,特别在基因组规模代谢网络模型的构建中发挥了重要作用。然而,许多机器学习模型忽视了生物背景,限制了模型的可信度和解释性。
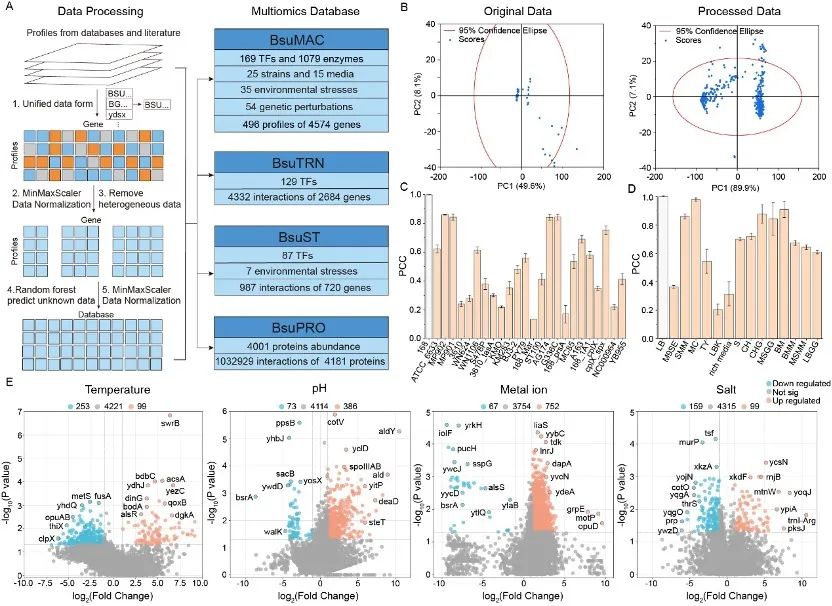
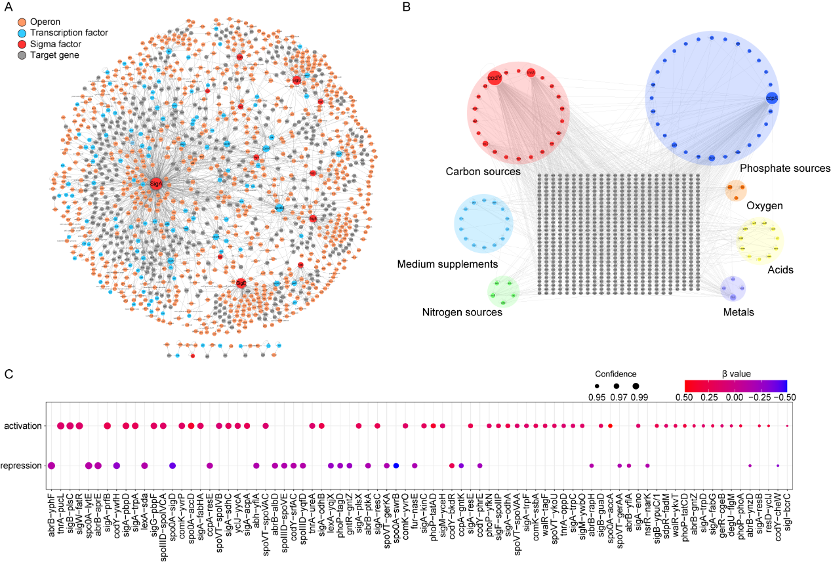
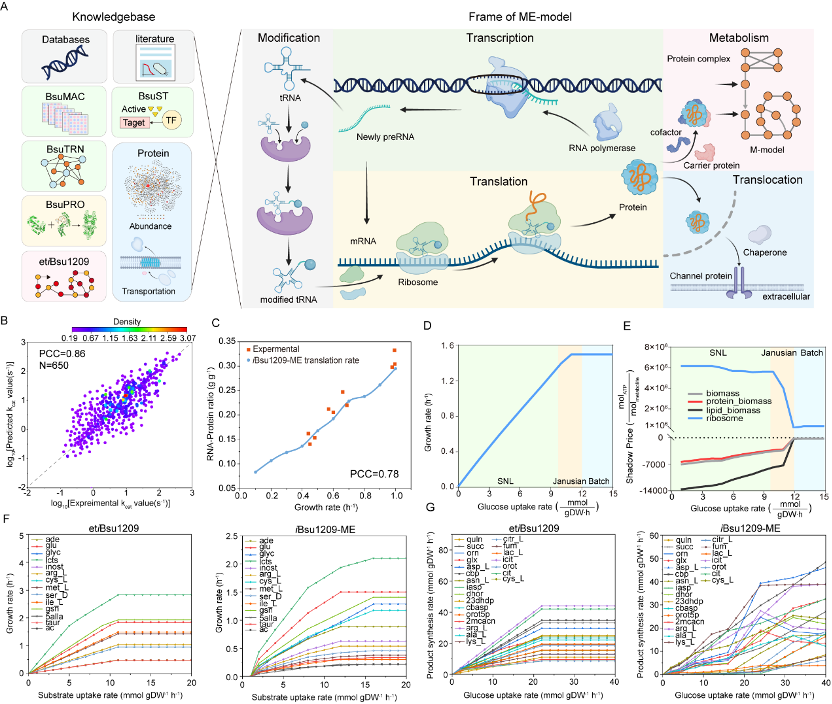
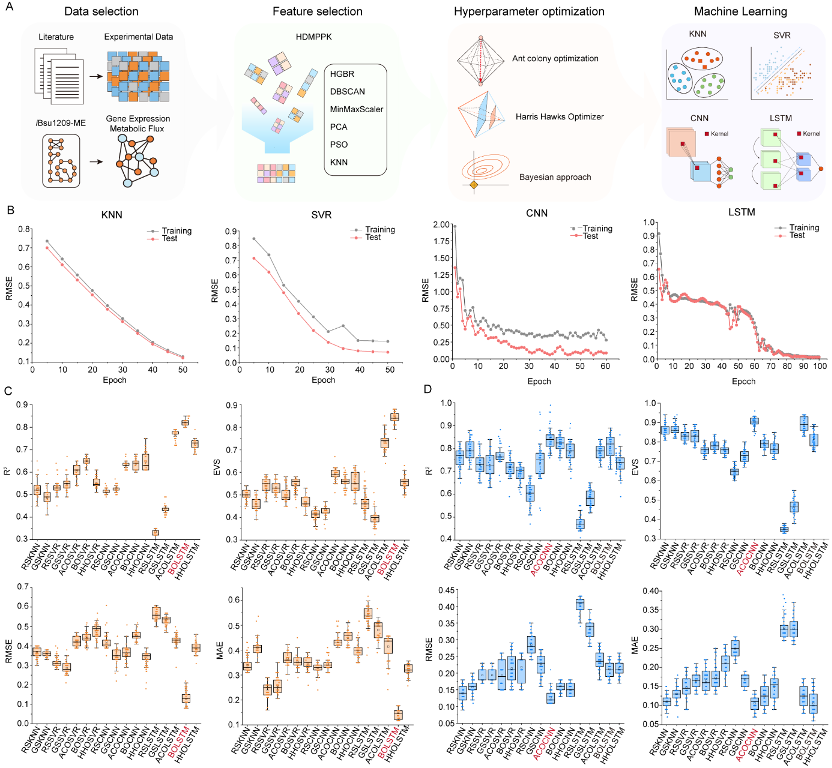
针对上述问题,该研究标准化建立了枯草芽孢杆菌高质量综合数据库,并搭建了枯草芽孢杆菌多组学综合代谢网络模型,设计了34个机器学习模型,将机器学习模型与多组学综合代谢网络模型相结合,实现了基因表达与细胞生长的精准预测。首先,手动收集了多个数据库和大量文献中的枯草芽孢杆菌组学数据,并建立了数据标准化策略,以消除实验条件和分析方法的系统性偏差,从而搭建了一个包括基因表达、转录调控、信号转导、蛋白翻译和细胞生长的标准化数据库(图1和图2)。其次,基于此构建了iBsu1209-ME模型,该模型涵盖了细胞所有主要代谢途径、基因转录、蛋白翻译、大分子修饰和转运反应,模型准确预测细胞生长速率和代谢途径的基因表达水平(图3)。随后,为完善iBsu1209-ME模型数据,选择五种超参数优化算法和四种机器学习框架,建立了34个机器学习模型,通过与湿实验数据对比,BOLSTM模型和ACOCNN模型分别实现了基因转录数据和蛋白翻译数据的精准预测(图4)。与此同时,提出了
HDMPPK特征工程框架,相比传统算法,HDMPPK框架在精简数据库的同时提取了原数据库的全部特征(图5)。
为全面理解基因表达对细胞生长的影响,在上述研究基础上构建了基因转录和蛋白翻译的集成回归模型。通过该模型成功预测了BsuMAC表达数据集中496个基因表达谱下的细胞生长速率,并优化iBsu1209-ME模型的转录和翻译模块,重建了EM_iBsu1209-ME。对比688个细胞生长相关基因的预测水平,EM_iBsu1209-ME的预测准确率达到87.9%,比iBsu1209-ME提高了46.7%
。此外,在12种不同培养条件下,EM_iBsu1209-ME的细胞生长速率预测值与实验数据高度相关(PCC=0.77)(图6)。
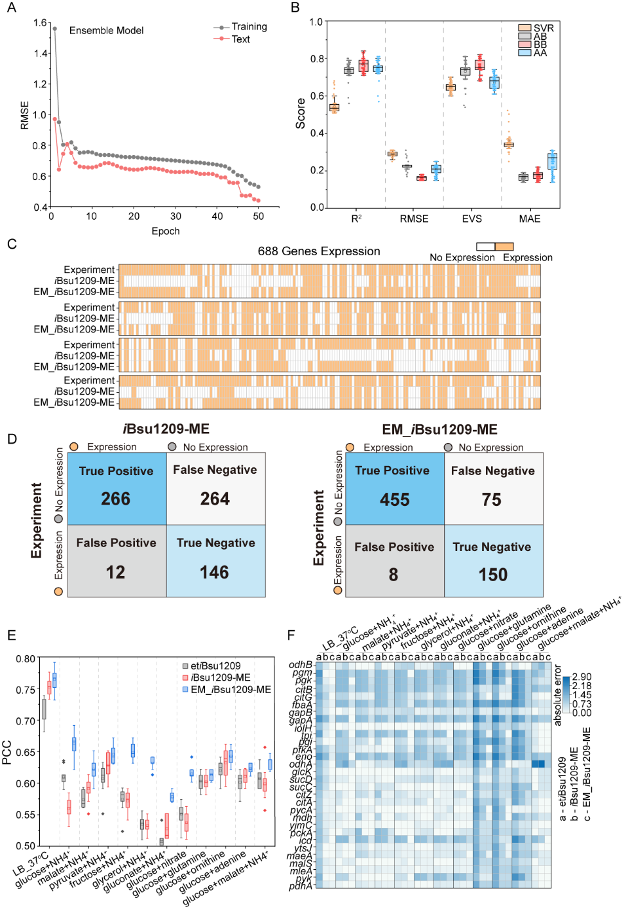
图6 EM_iBsu1209-ME集成模型构建及优化
总之,该研究为全面探索枯草芽孢杆菌的基因型-表型关系、指导细胞定向进化、探索未知的细胞过程提供了宝贵的数据支持。同时,这项工作为其他微生物的数据挖掘和模型开发提供了技术解决方案。
上述研究工作得到了国家重点研发计划(2020YFA0908300)、国家自然科学基金(32070085、32021005)等项目的资助。