最近有小伙伴反映收不到推送,因为公众号改了推送算法,现在需要加星标,多点赞、点在看,才能准时收到推送哦。
导语:转座因子 (TE) 对遗传多样性和基因调控至关重要。当前的单细胞定量方法通常将多重映射读取对齐到“最佳映射”或“随机映射”位置,并在亚家族级别对其进行分类,忽略了准确、位点特异性 TE 定量的生物学必要性。此外,这些现有方法主要针对转录组学数据而设计并侧重于转录组学数据,这限制了它们对其他模态单细胞数据的适应性。
作图丫不仅文章解读的好,课题做得也出色,已与国内多家知名医院的老师和名牌大学实验室达成合作。欢迎有生信分析需求的老师垂询,公共数据库数据挖掘或自测数据分析均可。

今天小编为大家带来的这篇文章,作者引入了 MATES,这是一种深度学习方法,可利用 TE 基因座两侧相邻读取对齐的上下文,将多重映射读取准确地分配给 TE 的特定基因座。文章发表在《nature communications》上,题目为:MATES: a deep learning-based model for locus-specific quantification of transposable elements in single cell。
本研究技术路线如图所示。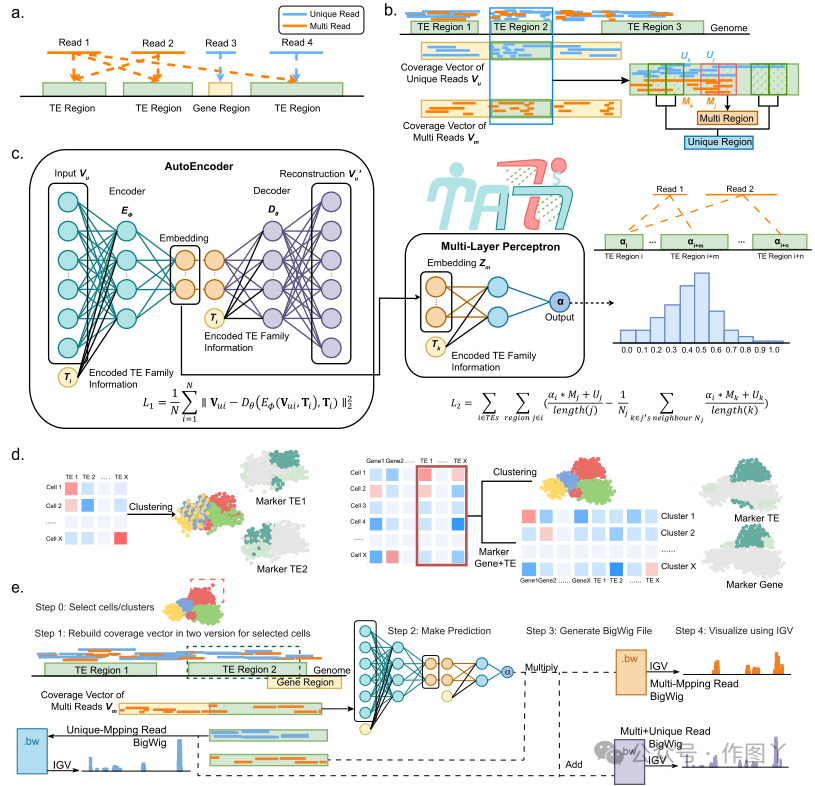 MATES 的 TE 量化和分析方法。a 将原始读取与参考基因组对齐,以解释 TE 基因座处的多重映射读取。b 构建 TE 覆盖向量,包括唯一读取覆盖向量 Vu 和多重读取覆盖向量 Vm,以捕获读取的分布信息。c AutoEncoder 模型从唯一读取覆盖向量中提取潜在嵌入。这些嵌入与 TE 家族数据 Ti 相结合,可预测多重映射读取与每个 TE 基因座对齐的可能性 α。d MATES 计算的多重映射概率 α 对于创建 TE 计数矩阵至关重要。该矩阵对于细胞分析至关重要,可以独立使用或与传统基因计数矩阵结合使用。这种组合使用增强了细胞聚类和生物标志物(基因和 TE)的发现,从而提供了对细胞特征的更全面了解。e MATES 在基因组浏览器中对全基因组读取覆盖率进行可视化。该方法可量化单个细胞中特定基因位点的 TE,生成 bigwig 文件,其中包含概率分配的多重映射读取的覆盖范围。这些文件包含唯一读取和多重映射读取,它们被合并以生成综合 bigwig 文件,以便使用诸如交互式基因组查看器 (IGV) 之类的工具对全基因组 TE 读取进行可视化。MATES 是一种专门用于对不同模态的单细胞数据集中的 TE 进行基因座级量化的工具。该方法涉及几个关键步骤。首先,将原始读取映射到参考基因组,识别唯一映射到 TE 基因座的读取(唯一读取)和映射到多个 TE 基因座的读取(多映射读取)(图 1a)。接下来,本研究计算每个 TE 基因座的覆盖向量,表示基因座周围唯一读取的分布(上下文)。然后将每个 TE 区域(基因座)细分为长度为 W 的较小箱体(例如,10 个碱基对)。根据箱体中唯一和多映射读取的百分比,将这些箱体中的每一个分类为唯一显性(U)或多显性区域(M)(图 1b)。有关选择这些超参数的详细信息,请参阅方法部分。第三,本研究采用自动编码器 (AE) 模型来学习潜在嵌入 (Vu),该嵌入表示 TE 基因座的高维唯一读取覆盖向量,这表明特定 TE 基因座两侧的映射上下文。独热编码的 TE 家族信息 (Ti) 也被作为模型的输入。第四,学习到的潜在嵌入 (Vu) 和 TE 家族嵌入 (Ti) 用于通过多层感知器回归器预测特定 TE 基因座的多重映射比率 (α)。学习模型的总损失由两个部分 (L1 和 L2) 组成。前者表示自动编码器的重建损失,而后者反映了 TE 上相邻小箱之间实际读取覆盖的连续性。本质上,由于它们的基因组接近,多重映射显性 (M) 箱上的最终读取覆盖应该接近其相邻的唯一显性 (U) 箱。最后,一旦本研究训练了预测每个 TE 基因座的多重映射比率的模型,本研究就可以利用它来计算落入特定 TE 基因座的总读取数,从而在基因座水平上呈现 TE 的概率量化(图 1c)。
MATES 的 TE 量化和分析方法。a 将原始读取与参考基因组对齐,以解释 TE 基因座处的多重映射读取。b 构建 TE 覆盖向量,包括唯一读取覆盖向量 Vu 和多重读取覆盖向量 Vm,以捕获读取的分布信息。c AutoEncoder 模型从唯一读取覆盖向量中提取潜在嵌入。这些嵌入与 TE 家族数据 Ti 相结合,可预测多重映射读取与每个 TE 基因座对齐的可能性 α。d MATES 计算的多重映射概率 α 对于创建 TE 计数矩阵至关重要。该矩阵对于细胞分析至关重要,可以独立使用或与传统基因计数矩阵结合使用。这种组合使用增强了细胞聚类和生物标志物(基因和 TE)的发现,从而提供了对细胞特征的更全面了解。e MATES 在基因组浏览器中对全基因组读取覆盖率进行可视化。该方法可量化单个细胞中特定基因位点的 TE,生成 bigwig 文件,其中包含概率分配的多重映射读取的覆盖范围。这些文件包含唯一读取和多重映射读取,它们被合并以生成综合 bigwig 文件,以便使用诸如交互式基因组查看器 (IGV) 之类的工具对全基因组 TE 读取进行可视化。MATES 是一种专门用于对不同模态的单细胞数据集中的 TE 进行基因座级量化的工具。该方法涉及几个关键步骤。首先,将原始读取映射到参考基因组,识别唯一映射到 TE 基因座的读取(唯一读取)和映射到多个 TE 基因座的读取(多映射读取)(图 1a)。接下来,本研究计算每个 TE 基因座的覆盖向量,表示基因座周围唯一读取的分布(上下文)。然后将每个 TE 区域(基因座)细分为长度为 W 的较小箱体(例如,10 个碱基对)。根据箱体中唯一和多映射读取的百分比,将这些箱体中的每一个分类为唯一显性(U)或多显性区域(M)(图 1b)。有关选择这些超参数的详细信息,请参阅方法部分。第三,本研究采用自动编码器 (AE) 模型来学习潜在嵌入 (Vu),该嵌入表示 TE 基因座的高维唯一读取覆盖向量,这表明特定 TE 基因座两侧的映射上下文。独热编码的 TE 家族信息 (Ti) 也被作为模型的输入。第四,学习到的潜在嵌入 (Vu) 和 TE 家族嵌入 (Ti) 用于通过多层感知器回归器预测特定 TE 基因座的多重映射比率 (α)。学习模型的总损失由两个部分 (L1 和 L2) 组成。前者表示自动编码器的重建损失,而后者反映了 TE 上相邻小箱之间实际读取覆盖的连续性。本质上,由于它们的基因组接近,多重映射显性 (M) 箱上的最终读取覆盖应该接近其相邻的唯一显性 (U) 箱。最后,一旦本研究训练了预测每个 TE 基因座的多重映射比率的模型,本研究就可以利用它来计算落入特定 TE 基因座的总读取数,从而在基因座水平上呈现 TE 的概率量化(图 1c)。
图 1
通过将 TE 量化与来自单细胞数据的传统基因量化(例如,基因表达或基因可及性)相结合,本研究在以下部分中将其称为“基因+TE 表达”,本研究可以更准确地对细胞进行聚类并识别综合生物标志物(基因和 TE)以表征获得的细胞簇(细胞亚群)。MATES 配备了先进的功能,可以有效处理各种单细胞数据模式。其应用提供了对 TE 在各种数据集中的作用的洞察,包括对细胞进行聚类以及识别潜在的生物标志物 TE(图 1d)。除了其分析能力之外,MATES 还提供了特定于基因座的 TE 可视化和解释。该工具有助于生成全面的 bigwig 文件和交互式基因组查看器 (IGV) 图,使研究人员能够直观地探索和解释整个基因组中 TE 基因座的读取分配(图 1e)。此功能解锁了对 TE 与位于 TE 基因座附近的基因之间潜在相互作用的研究,大大增强了本研究对 TE 动态及其对基因调控和细胞功能影响的理解。请注意,除非特别提到,否则本文中使用的术语“TE”代表从 RepeatMasker 中识别出的重复元素。这使本研究能够在研究中提供基因组重复的全面概述。在讨论 TE 的“更严格”定义时,本研究特别提到了包括哪些 TE 亚家族。为了证明 MATES 在单细胞 RNA 测序数据中 TE 定量的精确度,本研究将其应用于小鼠的 10x 单细胞化学重编程数据集。该分析确定了 2 细胞样细胞 (2CLC) 的标志性 TE。通过使用 MATES 量化 TE 表达,本研究将量化的 TE 计数矩阵与基因表达谱相结合,从而实现全面的聚类和可视化分析,如图 2a、b 所示。本研究揭示了 2CLC 的一个独特亚群(簇 17),位于重编程的 II 期和 III 期之间。值得注意的是,MATES 检测到了 2CLC 群体并在过渡阶段簇中区分了它们的标志性基因标记,尤其是 Zscan4d 和 Zscan4c。此外,MATES 还确定了在 2CLC 簇中富集的特定 TE 标记 MERVL-int 和 MT2_Mm,证实了先前的研究将这些 TE 视为 2CLC 的定义标记。这些发现凸显了 MATES 捕获细胞群及其重要生物标记(基因和 TE)的能力,为了解重编程的细胞动力学提供了见解。
接下来,本研究进行了以 TE 为中心的分析,以进一步验证 MATES 的 TE 表达量化在细胞聚类和生物标志物发现中的独特作用(图 2c、d)。在量化 TE 表达时,本研究注意排除 TE 与其相邻基因之间的重叠区域,以防止基因表达数据的潜在信息泄露。这种以 TE 为中心的分析专门关注 TE 表达,并确定了 2CLC 细胞群。此外,该分析不仅证实了先前与 2CLCs 群体相关的发现,而且还重申了其相关 TE 生物标志物(即 MERVLint 和 MT2_Mm)的相关性,如图 2c、d 所示。这表明本研究的细胞聚类和生物标志物发现不仅仅依赖于传统的基因表达分析。相反,MATES 独立进行的 TE 量化提供了一致的细胞聚类结果,并准确识别了已识别细胞群体的特征 TE。为了更清晰、定量地查看仅基于 TE 的聚类准确性,本研究纳入了混淆矩阵并计算了调整后的兰德指数 (ARI) 和归一化互信息 (NMI) 分数,以突出基于 TE 和传统基于基因的分析结果之间的相似性。将仅基于 TE 表达的聚类结果与基于基因表达的聚类结果进行了比较。主要簇(例如簇 1 和簇 12,分别代表 SIII_D12 和 2CLC)被仅 TE 簇有效捕获。这些 TE 簇与基因表达簇对应性良好,ARI(中位数 0.397,P < 1 × 10−6)和 NMI(中位数 0.496,P < 1 × 10−6)得分较高,表明高度一致。此外,通过关注由 TE 表达驱动的聚类,仅从多映射读取中进行量化,MATES 展示了其管理这些具有挑战性的读取并识别与其特定发育阶段精确一致的生物标志物的能力。
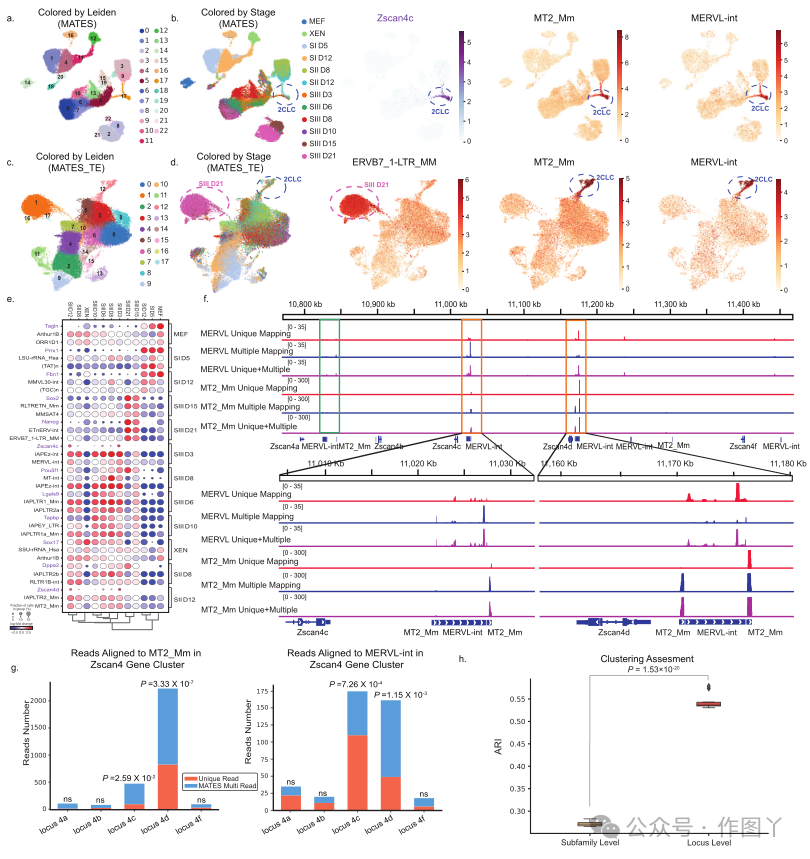
图 2
MATES 不仅可以识别 2CLC 和不同重编程阶段的细胞群的特征基因和 TE 标记(图 2e),而且还能有效地将多重映射读取对齐到特定基因座,而这一挑战阻碍了当前的方法。例如,scTE 仅限于将多重映射读取分配给元基因(同一亚家族的 TE),而没有对特定基因组位点进行明确分配。虽然 SoloTE 在基因座级别量化唯一映射到 TE 的读取,但它仅保留多重映射读取的最佳对齐,然后在亚家族级别对其进行量化。相比之下,通过利用每个 TE 基因座 (α) 的学习多重映射率,MATES 以概率方式将多重映射读取分配给整个基因组中的 TE 基因组位点。通过这种策略,本研究可以准确地量化基因座水平的 TE 表达,这在评估 2CLC 细胞的多重映射读取时很明显(图 2f、g)。观察到与 MT2_Mm 和 MERVL-int 链接的多重映射读取与基因 Zscan4c 和 Zscan4d 紧密对齐,并且与 MT2_Mm 和 MERVL-int 链接的与 Zscan4c 和 Zscan4d 基因座紧密对齐的总读取明显高于其他控制基因座(图 2g)。这种对齐与 Zhu 等人 31 的发现一致,其中 Zscan4c 的激活与内源性逆转录病毒 MT2_Mm/MERVL-int 的激活相关。请注意,基于唯一读取的基因座量化(在面板 g 中以橙色突出显示)代表 SoloTE 策略。该策略在基因座级别处理唯一读取,在亚家族级别处理多重映射读取。因此,在基因座级别,SoloTE 仅利用唯一读取,这可能导致映射到关键基因座(例如 4c 和 4d)的读取缺失,表明其存在潜在局限性。此外,与现有方法中通常使用的亚家族级别 TE 定量相比,基因座特异性 TE 定量提高了聚类准确性,这在图 2h 中清晰地得到了证明。这强调了精确的基因座级别 TE 定量的显著优势。
MATES量化人类胶质母细胞瘤中与疾病相关的TE表达为了证明 MATES 的跨平台适用性,本研究测试了该工具并将其应用于 Smart-Seq2 全长测序平台的另一个单细胞 RNA 测序数据集,重点关注人类胶质母细胞瘤数据集。MATES 的 TE 表达定量和常规基因表达分析的结合使用使本研究能够精确定位胶质母细胞瘤微环境中的不同细胞群,如 UMAP 图所示(图 3a、b)。本研究观察到某些 TE 的表达模式与关键的胶质瘤基因标记物(如 EGFR33,34)和 TE 标记物(包括 HUERS-P1-int35 和 HERVK-int36)以及免疫细胞基因标记物(如 CD7437,38 和 TE 标记物 LTR2B39,40)相关(图 3b)。这些相关性表明 TE 可能与肿瘤异质性和胶质母细胞瘤免疫反应相关的过程有关。需要进一步研究以探索任何因果关系和潜在机制。将基于 TE 的细胞分型与基因表达数据相结合,揭示了基因和 TE 之间的详细相互作用。这种整合展示了基于 TE 的聚类如何补充基因表达分析,从而提高细胞异质性研究的分辨率。 为了进一步证明 MATES 的精确度,本研究
还仅基于 MATES 量化的 TE 计数矩阵进行了细胞聚类。虽然仅 TE 分析可能无法比组合分析实现更好的聚类精度,但必须强调的是,TE 量化包含能够产生与传统基于基因的分析一致的结果的生物信息。具体而言,本研究系统地将仅 TE 的结果与基因表达聚类结果进行了比较,发现它们之间存在明显的相似性。莱顿簇 0 和 1 对应于免疫细胞,而簇 2、3 和 4 对应于肿瘤细胞。ARI(中位数为 0.105,P = 1.03 × 10−2)和 NMI(中位数为 0.161,P = 7.60 × 10−4)得分表明 TE 表达聚类和基因表达聚类之间存在弱但显著的一致性。混淆矩阵进一步将 TE 簇与基因簇和细胞类型进行比较,显示 TE 簇 0 与主要由免疫细胞组成的基因簇 0 和 1 显着重叠,而 TE 簇 2 与基因簇 4 和 5 一致,主要包含肿瘤细胞。这表明基于 TE 的聚类可以准确地重新捕获所有主要细胞群,并识别它们相关的 TE 标记(图 3c,d)。点图(图 3e)不仅显示了特定标记基因、TE 和细胞类型之间的关联,还量化了它们的相对表达水平,为数据分析增加了更深的维度。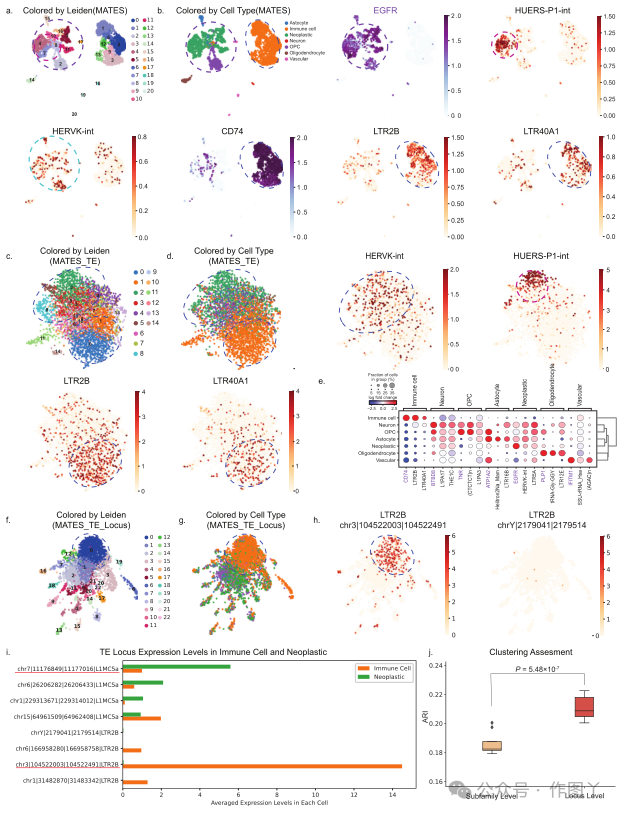
图 3
除了分析上述亚家族水平的 TE 表达外,MATES 的基因座水平 TE 定量分析还提供了更全面的细胞景观视图(图 3f-h)。这种方法有助于识别与先前在亚家族水平上识别的标记 TE 相对应的高表达 TE 基因座。值得注意的是,即使对于同一个 TE 亚家族,例如 LTR2B,不同的基因座也可能表现出不同的表达模式(图 3h,i),这强调了精确和基因座特异性 TE 定量的迫切必要性。chr3∣104522003∣104522491∣LTR2B(chrom∣start∣end∣TE)处的 LTR2B 基因座是免疫细胞中高表达的基因座特异性 TE 标记,与 CD166 基因接近,表明存在潜在的调控相互作用。CD166 对免疫细胞粘附和功能至关重要,可能受 LTR2B 通过其调控元件的影响。TE 可以通过提供启动子、增强子和转录因子结合位点来影响附近的基因表达,促进对免疫反应至关重要的快速动态基因表达变化。此外,TE 是表观遗传修饰的靶标,可进一步调节附近的基因并增强免疫细胞适应性。需要进一步的实验分析才能充分了解它们的相互作用。此外,与亚家族级别的定量方法相比,这种基因座特异性 TE 定量的应用显着提高了细胞聚类准确性,如图 3j 所示(P = 5.48 × 10−7),突出了其在分析细胞异质性和理解 TE 功能方面的关键作用及其相对于传统亚家族水平分析的优势。除了如上所示的对各种物种的普遍适用性之外,本研究提出的 MATES 模型还通过有效量化转录组数据和表观基因组数据中的 TE 展示了其多功能性。为了验证这种适应性,本研究将 MATES 应用于成年小鼠大脑的 10x 单细胞 ATAC-seq 数据集。这种与不同模式下的单细胞数据的兼容性至关重要,因为许多现有方法都是专门针对转录组数据量身定制的,并且可能不适用于其他单细胞模式。MATES 能够在不同的单细胞数据模式下量化 TE 基因座特定属性,例如染色质可及性,从而将其实用性扩展到转录组学之外。通过在 TE 亚家族水平上量化染色质可及性并结合标准单细胞 ATAC-seq 峰,MATES 促进了精细的细胞聚类和识别不同细胞群特有的 TE 标记(图 4a、b)。ERVK 家族中的 RMER16_MM 和 RLTR44B 等 TE 在巨噬细胞中表现出独特的可及性,而 MamRep434 和 MER124 则优先在星形胶质细胞中可及,这强调了 TE 在神经发生和星形胶质生成中的重要作用51。例如,MamRep434 对神经发生中的关键转录因子 Lhx2 基序的重大贡献象征着 TE 可及性在细胞身份和功能中的功能意义。仅利用 MATES 量化的 TE 表达数据进行聚类,本研究成功识别了 TE 生物标志物并在细胞组之间保持了清晰的界限(图 4c、d),证实了从 TE 量化中获得的见解植根于真正的生物现象。虽然将 TE 表达与基因或峰值计数等常规量化相结合通常会产生最佳的细胞嵌入质量和聚类,但仅 TE 分析对于强调除传统基因或峰值表达之外的 TE 的具体贡献至关重要。通过将基于 TE 的结果与常规基于基因或峰值的结果进行比较,本研究证明了 TE 定量具有高度信息量,并且可以产生与常规分析相似的一致细胞聚类结果。基于 TE 的簇和基于基因的簇之间的中位 ARI 为 0.309(P = 5.60 × 10−5),中位 NMI 为 0.438(P = 4.60 × 10−6)。图 4e 显示了所有细胞群的已识别特征 TE。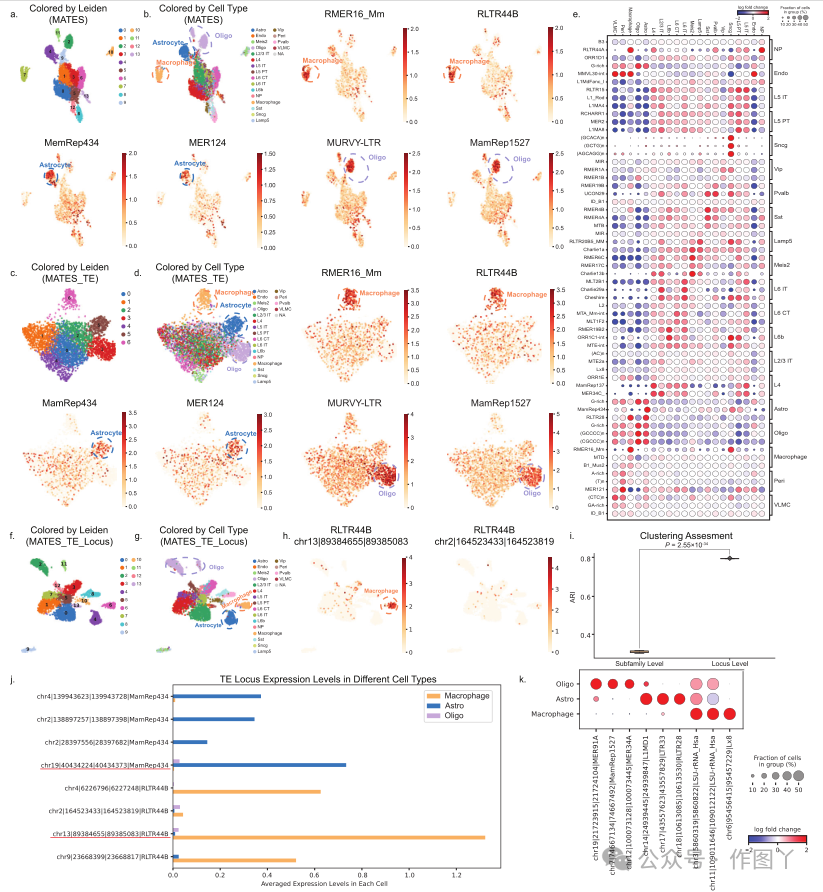
图 4
除了亚家族水平的 TE 定量和分析外,MATES 还提供基因座特异性 TE 定量,通过精确的 TE 基因座位置识别每个细胞群的特征 TE(图 4f-h)。与亚家族水平分析相比,基因座水平 TE 定量显示出明显更高的细胞聚类准确度(P = 2.55 × 10−34)(图 4i)。这强调了 MATES 在基因座水平 TE 定量中的有效性及其对了解数据中的细胞状态的潜在益处。在基因座水平识别的标记 TE 与在亚家族水平检测到的标记 TE 一致,验证了该方法的准确性并产生了关于 TE 对染色质可及性影响的基因座特异性见解。每个细胞群的这些基因座级 TE 生物标记物可能揭示与附近基因的相互作用及其对调节特定细胞类型的细胞状态的影响(图 4j、k)。例如,在这个小鼠大脑 scATAC 数据集中,基因座 chr13∣89384655∣89385083∣RLTR44B 在应该注释为小胶质细胞的细胞中表现出相当大的染色质可及性,尽管它们之前被注释为巨噬细胞(图 4h)。侧翼基因之一是 Edil3。定量蛋白质组学表明,与 WT 小鼠相比,APP-KI 小鼠分离的小胶质细胞中 Edil3 表达增加。该基因座还位于基因 Hapln1 的上游,该基因最近被鉴定为巨噬细胞相关调节剂,与癌症免疫疗法相关。这些发现支持该 RLTR44B 基因座在巨噬细胞/小胶质细胞群中的潜在作用。类似地,位于与星形胶质增生相关的 Sorbs1 基因附近的基因座 chr19∣40434224∣40434373∣MamRep434在表达高水平炎症标志物的精神分裂症患者中表现出表达升高 。此外,位于该转座因子 200kb 侧翼区域内的基因包括 Aldh18a1 和 Entpd19,它们在星形胶质细胞功能及其与非星形胶质细胞的相互作用中起关键作用。这些发现共同强调了这些已鉴定的标记 TE 基因座在影响星形胶质细胞相关基因表达方面的调控潜力。为了进一步证明 MATES 的广泛适用性,本研究将其应用于单细胞多组学数据集 (10x Multiome)。通过将 MATES 提供的 TE 量化与传统基因表达数据(来自 scRNA-seq 的 RNA 转录本)和匹配的可及性量化(来自 scATAC-seq 的染色质可及性)相结合,本研究区分了各种细胞群及其相关标记,如图 5a、b 所示。MATES 能够聚类不同的细胞,跨不同的测序技术识别不同的细胞群和 TE 生物标记,从而展示了 MATES 在利用多组学数据进行深入细胞分析方面的潜力(图 5c-f),结果强调了 MATES 内不同模式的协同作用。值得注意的是,当仅依赖 MATES 的 TE 量化(TE_scRNA 和 TE_scATAC)时,该方法可以有效捕获原代细胞群及其特征 TE 标记。相反,在基因座水平上的量化进一步提高了聚类性能(图 5g-l)。通过分析多组学数据,MATES 意外地发现某些 TE 在特定细胞亚群中具有唯一可辨别性,并且专属于特定模态(图 5m、n)。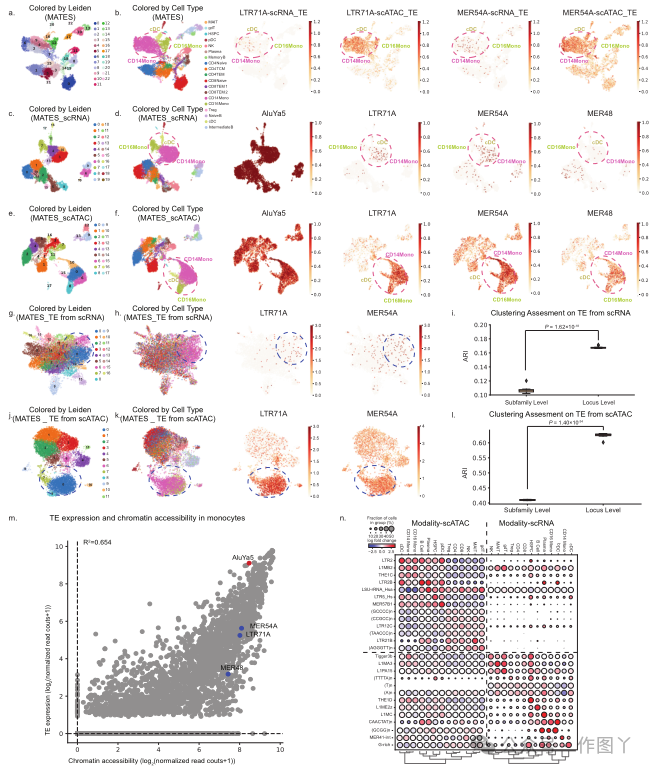
图 5
基因表达升高的 TE 通常表现出染色质可及性增加(用红点表示,例如已报道的 AluYa5)。相反,染色质可及性增强的转座子并不总是表示高表达水平(标记为蓝点)。例如,LTR71A 主要通过 scATAC-seq 检测到,而在 scRNAseq 数据集中不存在。在 TE 生物标志物(如 MER48 和 MER54A)中也观察到了类似的趋势,表明这些 TE 在未感染的单核细胞中可访问但处于转录休眠状态。这促使本研究提出,此类 TE 可能被引导至可能与单核细胞功能相关的“平衡”状态 。值得注意的是,这些 TE 中有几个之前被确定为“平衡”的 TE 在上调的 TE 中占据突出地位。通过超几何检验计算出的这种富集,显著的 p 值为 P = 5.91 × 10−24,是通过将通过 MATES 识别的标记 TE 与研究中报告的上调 TE 进行比较确定的。这一观察结果突出了 MATES 揭示 TE 生物学特征的能力。在这里,本研究
的结果强调,当通过 TE 棱镜进行分析时,染色质可及性和 RNA 丰度为单细胞状态提供了互补的见解(见图 5m)。 在整合基因和 TE 表达的分析中,MATES 表现出比 scTE 和 SoloTE 更好的性能,在用于化学重编程的 10x scRNA 数据集中尤其明显。在这种情况下,scTE 的有效性明显低于仅基因表达的方法,凸显了准确的多映射读取分配的重要性。MATES 在所有测试数据集中获得了更高的 ARI 和 NMI 分数,证明了这一点,如图 6a、b 的左侧面板所示。为了进一步展示和比较不同方法对 TE 定量的准确性,本研究进行了仅基于 TE 定量的细胞聚类分析,不包括基因表达数据。在这里,MATES 在聚类效率方面再次胜过 scTE 和 SoloTE,这反映在改进的 ARI 和 NMI 分数上(参见图 6a、b 的中间面板)。这一结果强调了 MATES 处理多映射读取的能力,并证明了其在 TE 定量中的重要性。在专注于多映射 TE 读取的场景中,MATES 管理这些具有挑战性的读取分配变得显而易见。如图 6a、b 的中间和右侧面板所示,MATES 在这些测试中始终优于 scTE 和 SoloTE。这种在不同测试条件下的持续改进证明了 MATES 在 TE 量化方面的稳健性和有效性。其在多映射读取分配方面的优势有助于提高后续细胞聚类任务的准确性。 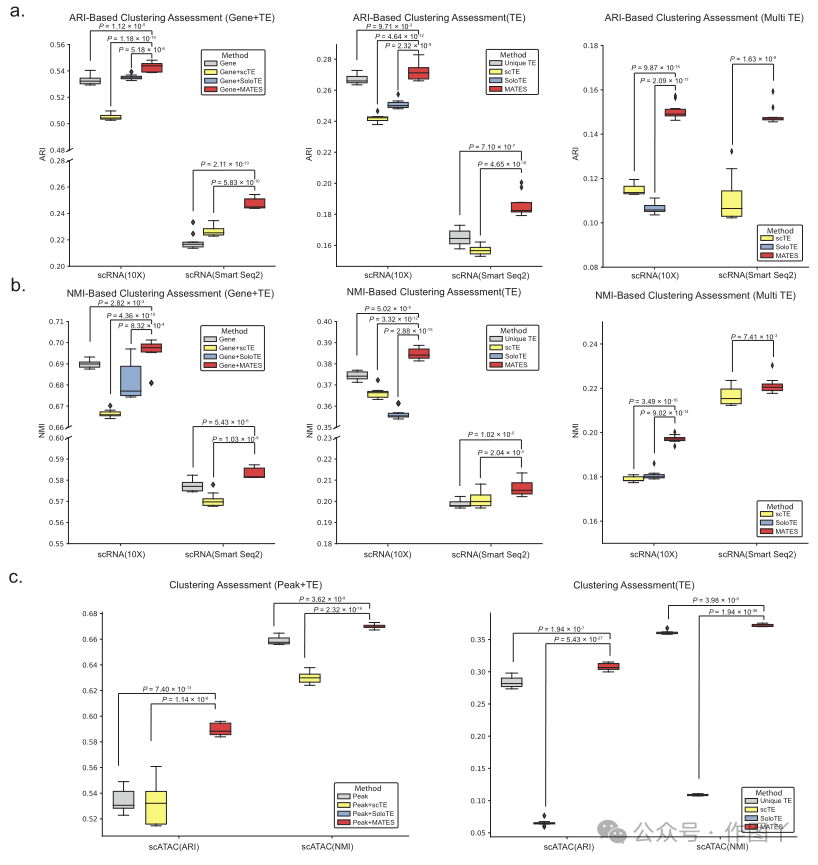
图 6
在本研究对 10x scATAC 小鼠脑数据集的基准分析中,本研究根据 scTE 和仅峰值量化方法评估了 MATES 方法。该评估旨在确定结合峰值和 TE 数据时的细胞聚类准确性。本研究的结果表明,MATES 在聚类准确性方面明显优于 scTE 和仅峰值方法,展示了其有效整合额外染色质可及性见解的能力。MATES 性能的一个关键方面是它对 TE 表达的全面量化,其中包括独特的映射读取和整体 TE 数据。这种方法大大超越了 scTE,有助于增强聚类过程。MATES 包含多重映射读取,尽管由于其在此 scATAC-seq 数据中的频率较低,因此不如 scRNA 数据集那么重要,但它为聚类分析提供了宝贵的见解,如图 6c 所示。这些发现不仅凸显了 MATES 的有效性,而且还证明了其在各种单细胞数据模式中的适应性和广泛应用的潜力。此外,本研究使用 2CLCs 10x scRNA 数据集将 MATES 与现有的基因座级 TE 定量方法进行了对比。由于 scTE 不支持基因座级 TE 定量,本研究仅将 MATES 的结果与 SoloTE 的结果进行了比较。基于基因座级 TE 的细胞聚类的 ARI 分数,其中 MATES 的 ARI 分数比 SoloTE 高 10.52%(P = 2.60 × 10−12)。这种改进的性能可能归因于 MATES 能够利用来自多映射读取的基因座级 TE 表达,而 SoloTE 只能量化来自唯一映射读取的基因座级 TE 表达。通过长读测序数据进行验证。为了验证本研究的 TE 定量方法 MATES 的准确性,本研究使用了来自 PacBio 和 Nanopore 平台的长读测序数据。PacBio 的 Sequel II 系统可生成高保真 (HiFi) 读数,准确度极高,通常超过 99%,非常适合需要精确碱基级分辨率的应用,例如识别特定的 TE 插入位点。纳米孔测序虽然通常以超长读数而闻名,但在本研究中提供了约 900 bp 的读数。准确率在 85% 到 95% 之间,它仍然为重复区域和 TE 结构提供了宝贵的见解。这两种长读测序技术都通过跨越长重复区域和捕获更多突变来增强本研究区分相似 TE 实例的能力。包括来自两个平台的数据集可以通过利用每种技术的互补优势来有力地验证 MATES。为了确定 MATES TE 定量的精度,本研究首先利用了来自纳米孔测序平台的黑色素瘤脑转移数据集。该数据集包括单细胞纳米孔 RNA 测序 (scNanoRNAseq)67,产生平均长度为 937 个碱基对 (bp) 的长读数据。这比从研究中的同一组细胞中获得的 10x scRNA-seq(真实测序读数)的 222 bp 的相应短读数据要长得多。纳米孔测序的较长读长有利于 TE 定量,因为它可以实现更准确的读段比对(即,纳米孔测序中的多重映射率降低约 1%,而同一研究中的 10x 短读测序中多重映射率超过 12%),从而提高了 TE 定量的可靠性。长读数据的这种精度水平是本研究通过 MATES 验证短读 TE 定量的基本事实。在通过纳米孔长读测序数据验证 MATES 时,本研究比较了单个细胞内通过每种方法(包括 MATES、scTE 和 SoloTE)量化的 TE 表达。为了提高相关性计算的准确性并准确反映短读和长读表达之间的关系,本研究仅包括明确定义的 TE 家族(DNA、LTR、RC、逆转录座子、SINE 和 LINE)。这是为了确保对转座因子进行有针对性和准确的分析,并且 R2 值真正代表了生物数据,不受非表达或异常读数造成的扭曲。如图 7a-c 所示,MATES 对 TE 的量化与真实长读数据(R2 = 0.7531)显示出很强的相关性,在从真实的 10x 短读数据量化亚家族级别的 TE 表达方面超过了 scTE(R2 = 0.6499)和 SoloTE(R2 = 0.6841)。此外,数据中的拟批量 TE 表达(通过对真实短读 10x 测序数据集中所有细胞的 TE 表达进行平均来确定)也通过相应的方法进行量化。MATES 与已建立的地面真实长读数据表现出更强的相关性,R2 值达到 0.9276。这一性能超过了其他方法,例如 scTE 和 SoloTE,它们的 R2 值分别达到 0.8591 和 0.8654。 图 7
图 7
通过受控模拟进行验证。尽管 Nanopore 和 PacBio 平台的长读测序提供了验证,但这些技术在捕获 Alu 重复等短 TE 方面存在局限性。为了解决这些限制,本研究使用模拟的 Alu 重复数据进行了基准测试,长度约为 300 bp(图 7d)。使用模拟数据作为基准可以更稳健地比较不同方法的性能,因为基准测试不受测序错误或其他技术限制的影响。有关构建模拟数据集的详细信息,请参阅方法中的“使用受控模拟进行验证”。然后将 MATES 的量化与模拟的基准进行比较,得出 R2 值为 0.7420(图 7e)。模拟数据由没有 UMI 的全长 RNA 测序读数组成,这是 SoloTE 处理和映射所必需的。因此,SoloTE 与这些数据不兼容,促使人们将重点放在比较亚家族级别的性能与 scTE 上。如图 7f 所示,与其他评估方法相比,MATES 的量化结果更接近真实值。此外,基因组数据中普遍存在的简单和低复杂度重复也很短。然而,简单重复对进化和人类疾病有重要影响。因此,本研究还在此验证了 MATES 在量化简单和低复杂度重复表达方面的有效性。簇特异性简单重复也在上述结果中成为顶级标记 TE(例如图 3e、图 4e 和图 5n),突出了它们的潜在作用。为了展示简单重复在基因座水平上的读取分配的准确性,本研究
进行了一项类似于 Alu 家族模拟的详细模拟研究。评估了 MATES 量化读取数与每个基因座的模拟读取数之间的相关性,实现了高相关性(R2 = 0.7479,P = 2.02 × 10−43)。尽管 scTE 在少数 TE 实例中表现出更好的性能,但 MATES 在大多数亚家族中普遍表现出更好的性能。 MATES 的引入可以聚焦 TE 的复杂动态,将曾经被认为是“垃圾读取”的内容转化为可以重新定义本研究对细胞多样性和动态理解的信息宝库。随着单细胞技术的发展,准确和精确的 TE 量化将变得越来越重要。MATES 有望成为这一努力中的重要资产,引导研究人员通过转座子的视角,探索基因组复杂性,在单细胞水平上发现生物学的新方面。
码字不易,欢迎读者分享或转发到朋友圈,任何公众号或其他媒体未经许可不得私自转载或抄袭。由于微信平台算法改版,公众号内容将不再以时间排序展示,建议设置“作图丫”公众号为星标,防止丢失。星标具体步骤为:(2)点击右上角的小点点,在弹出界面选择“设为星标”即可。