6月9日,安徽大学徐春燕副教授、石先阳教授和阿卜杜拉国王科技大学Zhongzhen Wang(共同通讯)在Water Research发表了题为“Robust S3Former Deep Learning Model for the Direct Diagnosis and Prediction of Natural Organic Matter (NOM) from Three-Dimensional Excitation-Emission-Matrix (3D-EEM) Data”的研究论文,开发了基于Transformer架构的S3Former模型,用于直接解析不同天然有机物(NOM)种类及其浓度范围的三维荧光光谱数据。该模型不仅能精准诊断单一及二元NOM组分,还可同步预测其浓度分布。
天然有机物(NOM)是水生环境的重要组成部分,其组成涵盖脂肪族化合物至显色芳香族物质,直接影响水体色度与气味,并在水处理过程中引发诸多负面效应。例如,部分NOM组分在混凝与消毒阶段会生成威胁人类健康及环境的消毒副产物(DBPs)。因此,对NOM种类与浓度的直接、快速、精准识别,是实现高效去除NOM并阻断DBP生成的关键。传统NOM诊断方法如总有机碳(TOC)、溶解性有机碳(DOC)及紫外光谱(UV254)等,难以全面解析NOM的复杂组成与动态变化。
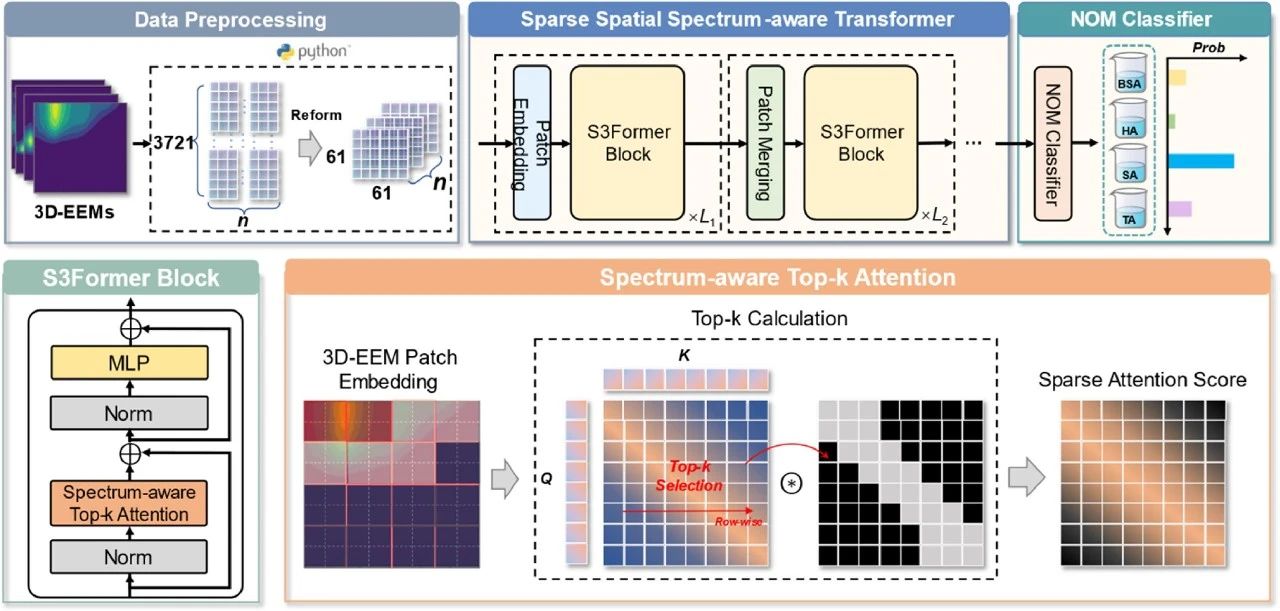
基于荧光光谱的三维激发-发射矩阵(3D-EEM)作为一种无损检测技术,已被广泛应用于水体NOM监测。然而,如何从3D-EEM数据中直接识别NOM种类及浓度仍面临挑战,尤其是在NOM混合体系及超滤分离等复杂场景下。本研究首先构建了包含多种工况的多任务学习3D-EEM光谱数据集,并集成具有稀疏空间谱(S3)感知机制的Transformer框架,开发了新型深度学习模型S3Former。该模型对单一及二元NOM种类的诊断准确率分别高达100%与97.2%,浓度预测准确率达75%与65.6%。其预测精度、稳定性及参数效率均显著优于十种卷积神经网络(CNN)模型及基础Transformer架构。进一步将S3Former应用于超滤产水NOM浓度区间预测这一更具挑战性场景(因NOM分子量分布变化),结果显示模型对腐殖酸的预测准确率仍达77%,展现出优异的鲁棒性。这些成果为提升水处理过程中水质自动化监控能力提供了创新技术支撑。
声明:本公众号仅用于分享前沿学术成果,无商业用途。如涉及侵权,请联系公众号后台删除!