为了方便各位同学交流学习,解决讨论问题,我们建立了一些微信群,作为互助交流的平台。2.告知:姓名-课题组-研究方向,由编辑审核后邀请至对应交流群(生长,物性,器件);欢迎投稿欢迎课题组投递中文宣传稿,免费宣传成果,发布招聘广告,具体联系人:13162018291(微信同号)
成果介绍
无标记分子成像技术能够构建生物组织的分子图谱,这对于理解复杂的生理和病理过程至关重要。包括正电子发射断层扫描(PET)、磁共振成像(MRI)、免疫分析和荧光显微镜在内的传统生物成像模式提供了宝贵的结构和功能信息,但在分子特异性、空间分辨率和标记要求方面仍存在局限。以进行性神经退行性变和区域特异性脑组织病变为特征的阿尔茨海默病(AD)正是这一需求的典型例证。
近日,莱斯大学黄声希教授团队提出了一种机器学习增强的高光谱拉曼成像框架,该框架实现了亚微米分辨率的无标记分子空间映射,构建了AD小鼠脑切片的全面分子图谱。通过将无监督与有监督机器学习(ML)算法与拉曼高光谱成像相结合,该框架高效提取了光谱变异、分子特征以及区域依赖性生化分布信息。生成的分子图谱显示,在AD小鼠海马体和皮层等特定脑区,Aβ42蛋白沉积显著增加,胆固醇与糖原代谢呈现区域特异性改变。这些结果证明了ML-拉曼成像技术具备超越经典Aβ病理学研究、捕获分子异质性的能力。该框架建立了一种可解释、数据驱动的空间分辨生化成像方法,将光谱学与人工智能相结合,实现了定量分子表征。该研究方法不仅适用于神经退行性疾病研究,还可广泛应用于异质性生物组织与纳米结构材料的分析,为探索复杂的化学与纳米尺度相互作用提供了多功能分析平台。
图文导读
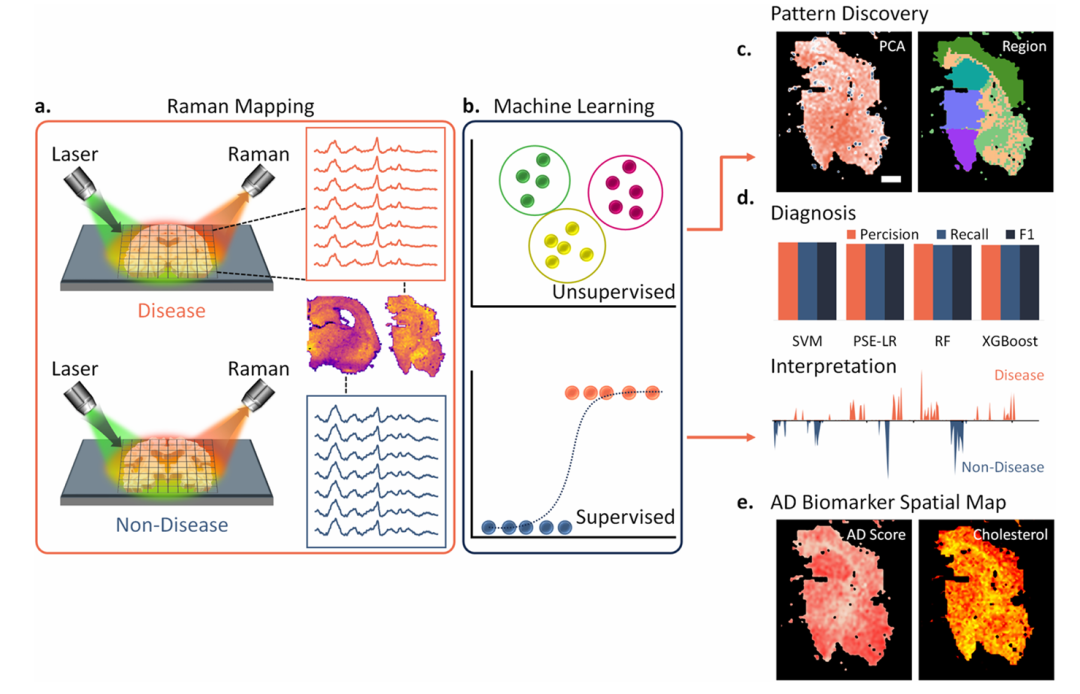
图1. ML-拉曼框架工作流程概览。(a) AD与非AD样本脑切片的高光谱拉曼成像。(b) 采用无监督与有监督ML算法的分析流程。(c) 基于K均值与主成分分析的分子特征提取与空间可视化。 (d) 运用支持向量机、泊松-稀疏编码逻辑回归、随机森林及XGBoost等监督式ML方法进行分类、特征重要性映射与生物标志物识别。(e) AD评分的空间映射及分子分布与疾病进展相关性分析。
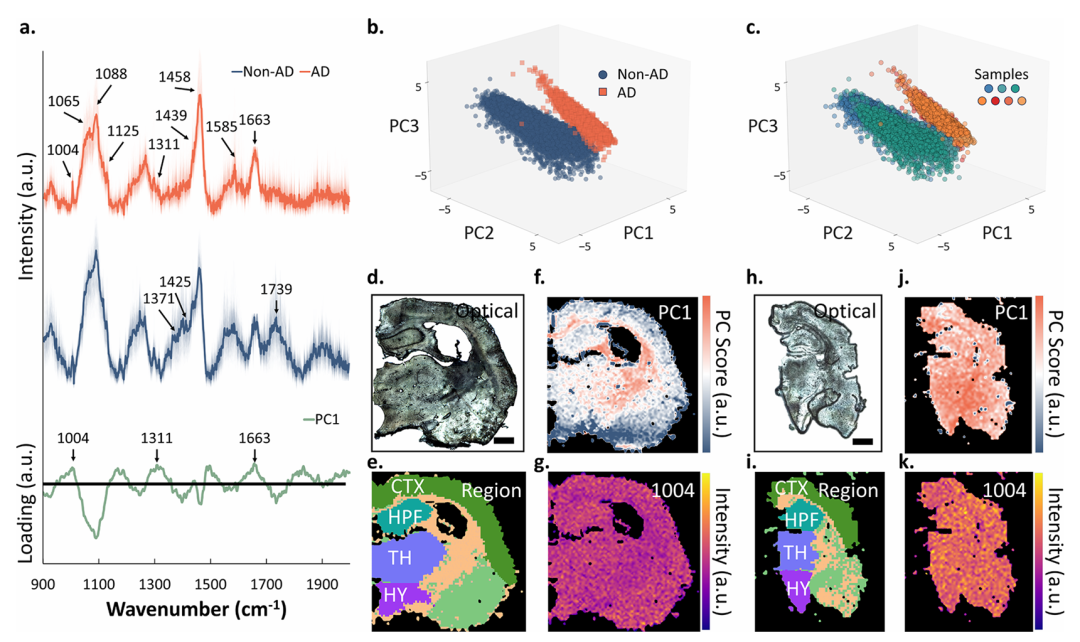
图2. 拉曼光谱与无监督机器学习分析。(a) AD与非AD脑切片的平均及单点拉曼光谱,并附对应光谱的主成分一载荷谱。(b) 按疾病状态(AD与非AD)颜色编码的主成分一、二、三得分分布图。(c) 按脑样本编号颜色编码的主成分一、二、三得分分布图。(d) 光学图像。(e) 基于区域边界K均值聚类标注的脑区划分。(f) 非AD样本的主成分一得分分布图。(g) 1004 cm⁻¹波数处的拉曼强度分布图。(h) 光学图像。(i) 基于区域边界K均值聚类标注的脑区划分。(j) AD样本的主成分一得分分布图。(k) 1004 cm⁻¹波数处的拉曼强度分布图。
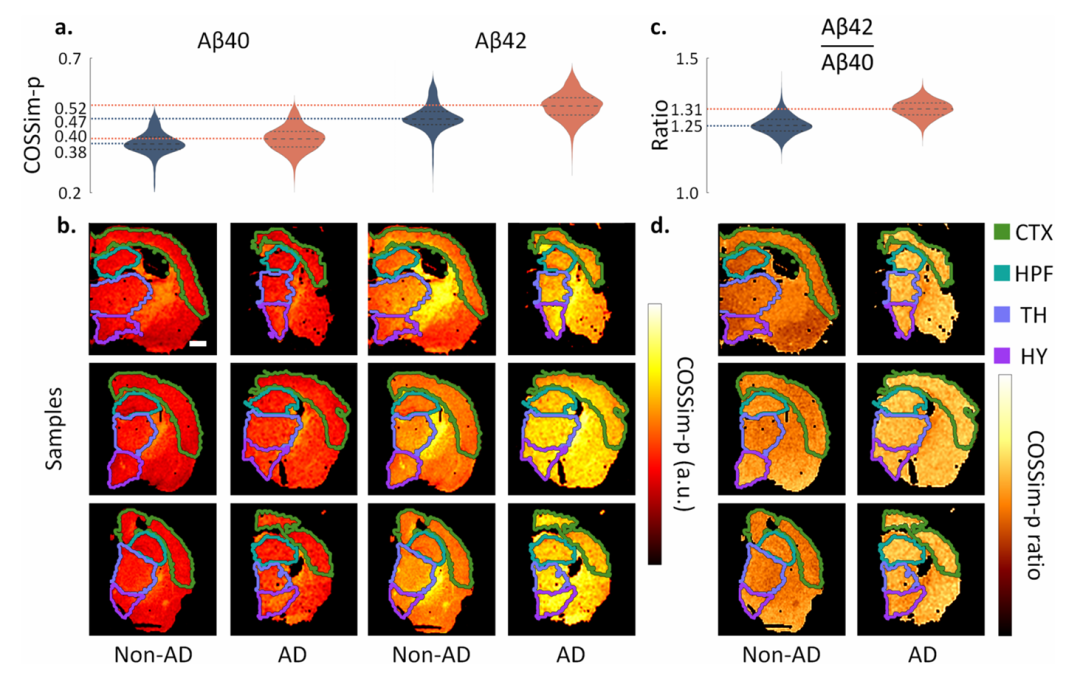
图3. Aβ40与Aβ42相对分子丰度的余弦相似度概率分析。(a) 非AD与AD脑切片中Aβ40及Aβ42的余弦相似度概率分布小提琴图。水平线代表第25、50(中位数)及第75百分位数。(b) 非AD与AD脑切片中Aβ40及Aβ42的余弦相似度概率空间分布图。(c) 非AD与AD脑切片中Aβ42/Aβ40余弦相似度概率比值的小提琴图。(d) 非AD与AD脑切片中Aβ42/Aβ40余弦相似度概率比值的空间分布图。
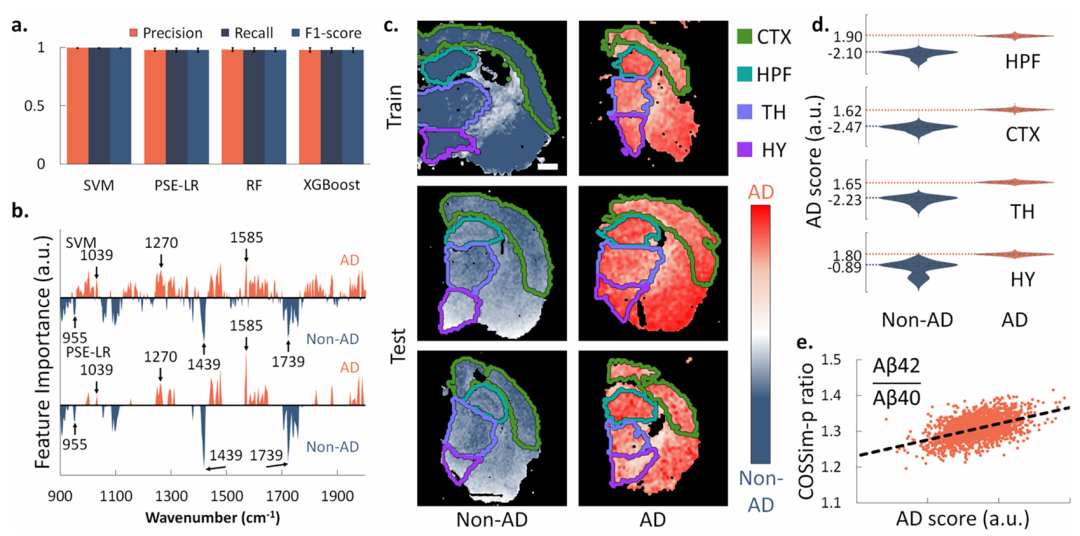
图4. AD与非AD脑切片的监督机器学习分析。(a) 使用支持向量机、泊松-稀疏编码逻辑回归、随机森林及XGBoost算法进行AD与非AD分类时,测试集的精确率、召回率与F1分数。交叉验证折叠间的方差以误差条形式呈现。(b) 支持向量机与泊松-稀疏编码逻辑回归生成的特征重要性分布图。(c) 泊松-稀疏编码逻辑回归对训练集与测试集生成的AD评分空间分布图。 (d) 海马结构、皮层、丘脑及下丘脑区域中非AD与AD样本AD评分的小提琴图。(e)
Aβ40/Aβ42余弦相似度概率比值与AD评分的相关性分析。
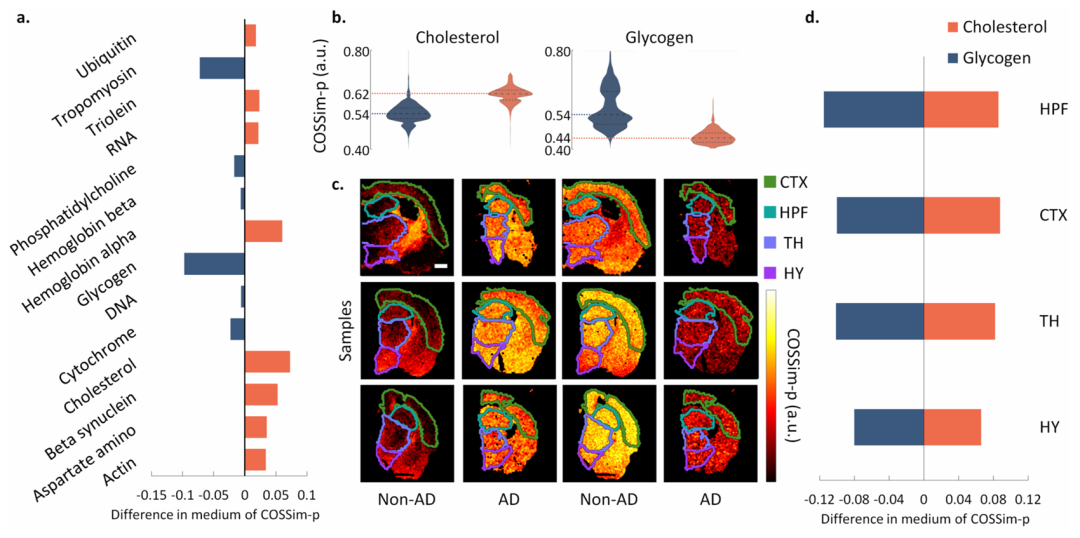
图5. 潜在生物标志物分子的识别。(a) AD与非AD样本间潜在生物标志物的余弦相似度概率中位数差异。(b) 非AD与AD脑切片中胆固醇与糖原的余弦相似度概率分布小提琴图。(c) 非AD与AD脑切片中胆固醇与糖原的余弦相似度概率空间分布图。(d) 海马结构、皮层、丘脑及下丘脑区域中AD与非AD样本间胆固醇与糖原的余弦相似度概率中位数差异。
结论与展望
综上所述,该研究基于机器学习增强的高光谱拉曼成像框架实现了阿尔茨海默病的无标记空间分辨分子图谱构建,揭示了Aβ42蛋白沉积增加以及胆固醇与糖原代谢在海马体和皮层等特定脑区的区域性改变。该研究方法不仅适用于阿尔茨海默病,更为广泛生物组织的分子复杂性探索提供了通用性分析平台。这些发现共同证明,拉曼高光谱成像技术与先进机器学习相结合,为疾病定量分子表征提供了强大且广泛适用的研究策略。
文献信息
Ziyang Wang, Jeewan C. Ranasinghe, Dennis C. Y. Chan, Ashley Gomm, Rudolph E. Tanzi, Can Zhang, Nanyin Zhang, and Shengxi Huang, “Machine Learning-Enhanced Hyperspectral Raman Imaging for Label-Free Molecular Atlas of Alzheimer’s Brain” ACS Applied Materials & Interfaces Article ASAP
文献链接:https://doi.org/10.1021/acsami.5c22623
上海昂维科技有限公司现
提供二维材料单晶和薄膜等耗材,器件和光刻掩膜版定制等微纳加工服务,以及各种测试分析,欢迎各位老师和同学咨询,竭
诚做好每一份服务。