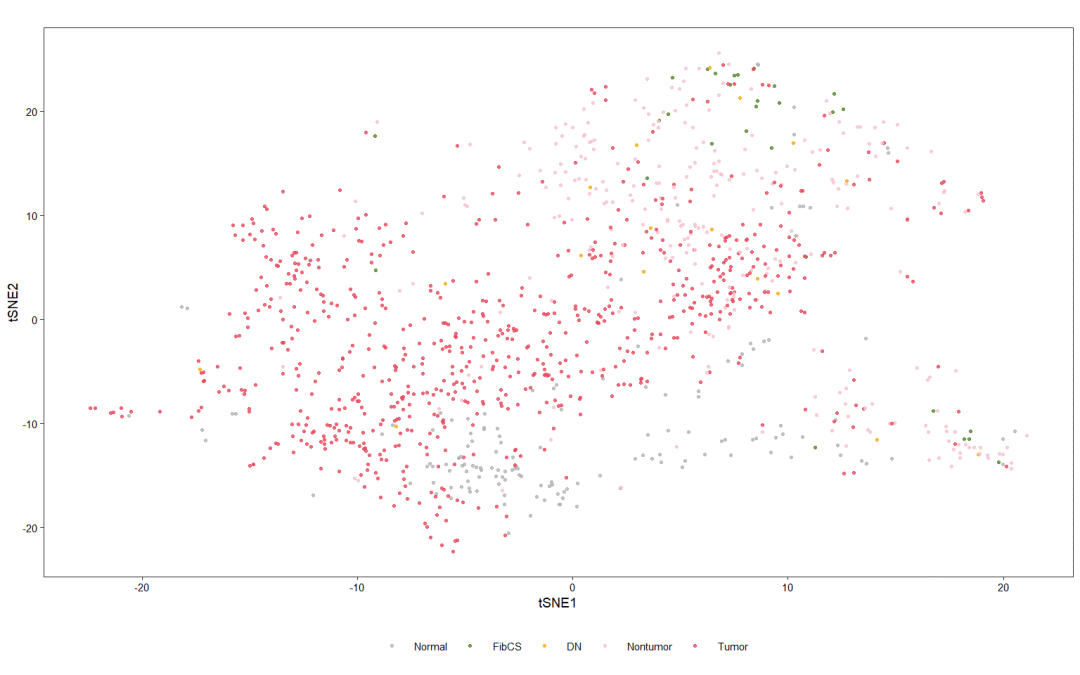
t-SNE(t-distributed stochastic neighbor embedding)是用于降维的一种机器学习算法,是由 Laurens van der Maaten 和 Geoffrey Hinton在08年提出来。并且,t-SNE是一种非线性降维算法,非常适用于高维数据降维到2维或者3维,进行可视化。相对于PCA来说,t-SNE可以说是一种更高级有效的方法,下面来实现Fig.2d的t-SNE图
文章来源:"Preoperative immune landscape predisposes adverse outcomes in hepatocellular carcinoma patients with liver transplantation" (2021,npj Precision Oncology),数据与代码全部公开在https://github.com/sangho1130/KOR_HCC。
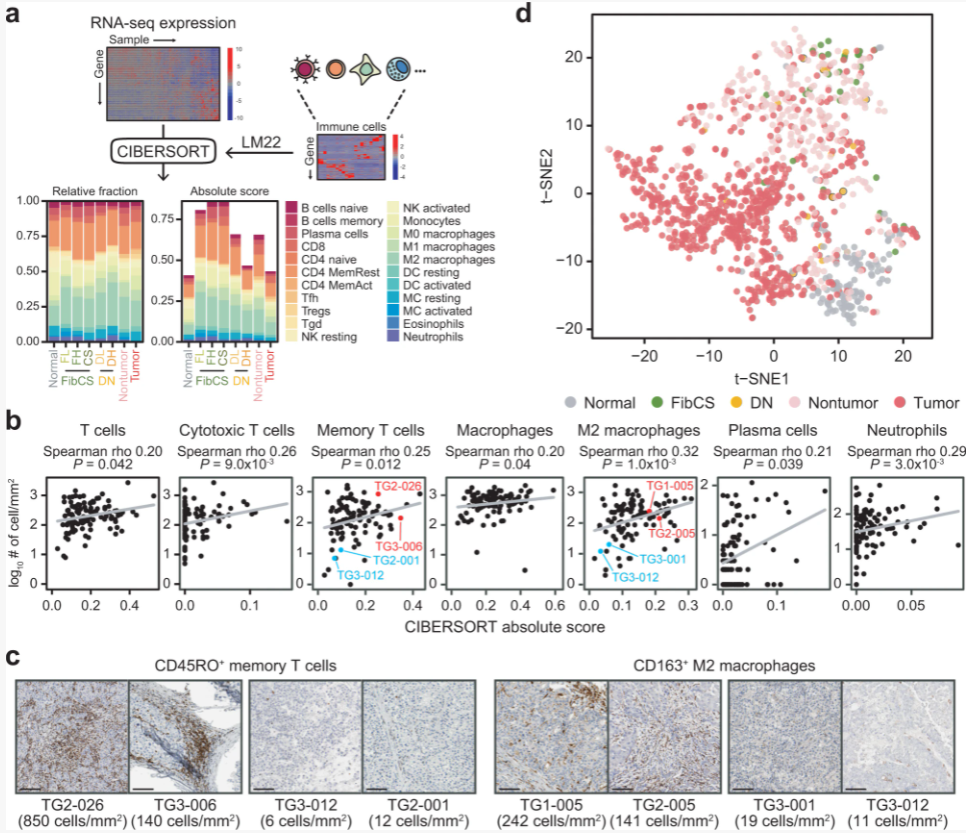
这里作者根CIBERSORT免疫分数进行t-SNE分析,将样本投影到二维坐标系。发现FibCS(肝纤维化)和DN(dysplastic nodule,癌前病变即发育不良结节)样本没有聚集在一起,并且与非肿瘤样本混合起来,且与肿瘤样本明显分离,推断出免疫浸润情况可以识别出癌前阶段的显著特征。
一、安装R包
主要用到Rtsne包,首先安装R包
install.packages("Rtsne") # Install Rtsne package from CRAN
也可以通过github安装
if(!require(devtools)) install.packages("devtools") # If not already installed
devtools::install_github("jkrijthe/Rtsne")
二、数据载入
rm(list = ls())
library(Rtsne)
library(ggplot2)
#library(rgl)
library(plyr)
table data # Rtsne() 在运行时默认会进行重复数据检测,如果不事先去除重复样本则会报错。
# data.uni = unique(data) # 去除重复数据
dataGrade data$Grade
head(data)
head(dataGrade)
二、tsne分析
基础用法如下:
tsne_out data,
dims = 2,
pca = FALSE,
perplexity = 10,
theta = 0.0,
max_iter = 1000,
verbose = F
)
参数解释:
data 用于降维的原始数据,其中行代表特征,列代表样本;pca 逻辑型变量,规定是否在t-SNE前预先进行PCA分析,默认为True。(pca参数表示是否对输入的原始数据进行PCA分析,然后使用PCA得到的topN主成分进行后续分析,t-SNE算法的计算量是特别大的,对于维度较高的数据数据,先采用PCA降维可以有效提高运行的效率,默认采用top50的主成分进行后续分析,当然也可以通过initial_dims参数修改这个值。)perplexity 这个值是一个正整数,且满足 3*perplexity < nrow(data) - 1 。作为计算数据点相似度的参数, perplexity 可以简单理解为对每个点具有的近邻数量的猜测,代表了平衡数据的局部和全局方面之间的程度,对生成的图像有复杂的影响。一般来说,随着perplexity 值的增加,形状越来越清晰。theta 计算速度与精确度之间的权衡,范围在0~1之间,越接近0越精确,默认0.5。这个参数影响最终计算结果,可以根据图像效果进行选取。max_iter 迭代次数,默认为1000,过小则结果未完全收敛,过大则浪费计算量。verbose 是否输出计算进度,在数据集较大的时候比较实用。
运算完成之后,结果保存在tsne这个对象中
说明书:https://cran.r-project.org/web/packages/Rtsne/Rtsne.pdf
实际分析:
# 由于t-SNE的结果具有随机性,因此在计算前必须设定固定的随机数种子,否则结果无法重复。
set.seed(0011) # 设置随机数种子
tsne
结果是一个有14个元素的list,其内容如下:
str(tsne)
# 其中的Y就是降维之后的二维空间对应的数据点,可以根据这个值进行可视化
scores rownames(scores) colnames(scores) head(scores)
三、设置分组
作者这个数据比较特殊,他是在分析完tsne之后再添加分组
# 新增grade列,为数据的分组
scores # mapvalues直接对数据的元素进行一一转换
scores$disease from = c('Catholic_Normal', 'GTEx',
'Catholic_CS', 'Catholic_FH', 'Catholic_FL', 'Catholic_DL', 'Catholic_DH',
'Catholic_T1', 'Catholic_T2', 'Catholic_T3T4', 'Catholic_Mixed',
'GSE77509_Nontumor', 'GSE77509_Tumor', 'GSE77509_PVTT',
'Riken_Nontumor', 'Riken_T1', 'Riken_T2', 'Riken_T3T4',
'TCGA_Nontumor', 'TCGA_T1', 'TCGA_T2', 'TCGA_T3T4'),
to = c('Normal', 'Normal',
'FibCS', 'FibCS', 'FibCS', 'DN', 'DN',
'Tumor', 'Tumor', 'Tumor', 'Tumor',
'Nontumor', 'Tumor', 'Tumor',
'Nontumor', 'Tumor', 'Tumor', 'Tumor',
'Nontumor', 'Tumor', 'Tumor', 'Tumor'))
# 转成因子,并设置顺序
scores$disease head(scores)
四、绘图
又回到我们散点图的画法了
plt geom_point(size = .5, alpha=0.8) +
scale_colour_manual(name="", values = c('Normal' = '#b3b2b2', 'FibCS' = '#528828', 'DN' = '#f2ae0e', 'Nontumor' = '#f5c2cf', 'Tumor' = '#e8495b')) +
theme_bw(base_size = 7) +
theme(axis.text = element_text(colour = 'black'), # 轴刻度值
panel.grid = element_blank(), # 空白背景
legend.position="bottom") + # 注释位置
labs(title = '')
plt
# ggsave('../results/Figure 2D 2dim.pdf', units = 'cm', width = 8, height = 6)
# scores.w # write.table(scores.w, '../results/Figure 2D tSNE projection.txt', quote = F, row.names = F, col.names = T, sep = '\t')
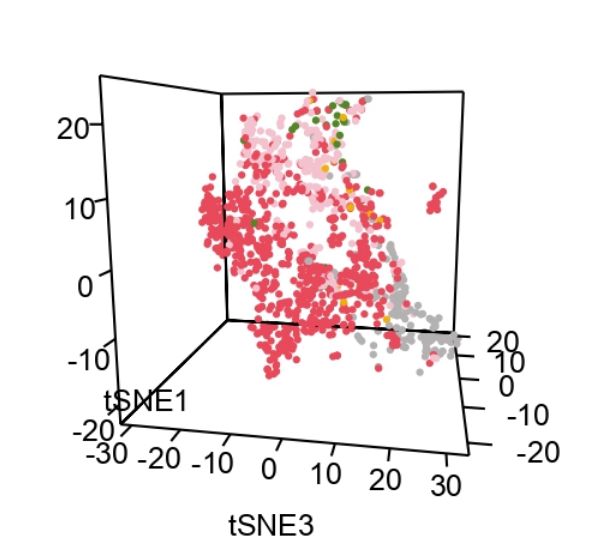
 五、交互式3D图
五、交互式3D图
这个就更炫酷了!
参考:http://www.sthda.com/english/wiki/a-complete-guide-to-3d-visualization-device-system-in-r-r-software-and-data-visualization
https://cran.r-project.org/web/packages/rgl/vignettes/rgl.html
library(rgl)
scores head(scores)
# 添加颜色
clustercolors table(clustercolors)
# 绘图
plot3d(scores[, 1:3], # xyz轴数据
col = clustercolors, # 颜色
type = 'p', # 形状。'p'表示点,'s'表示球面,'l'表示线,'h'表示从z = 0开始的线段,'n'表示空的线段
box = FALSE, # 外面是否绘制出箱子
size = 3) # 点的大小
# 加装饰
decorate3d(main = '', # 标题
box = FALSE, # 外面是否绘制出箱子
xlab = '', ylab = '', zlab = '') # xyz轴标签
# 加注释
legend3d("right", # 注释位置
legend = c('Normal', 'FibCS', 'DN', 'Nontumor', 'Tumor'), # 注释内容
col = c('#b3b2b2', '#528828', '#f2ae0e', '#f5c2cf', '#e8495b'), # 注释颜色
pch = 16, # 符号标识
cex=0.5, # 大小
inset=c(0.01)) # 相对位置,第一个参数为横轴,第二参数为纵轴
view3d(theta = 280, phi = 10, zoom = .9) # 将视点旋转到合适的位置
# 保存3D图
# rgl.postscript(filename = "../results/Figure 2D 3dim.pdf", fmt = "pdf", drawText = TRUE)
# rgl.close()
3D