为了到达它们的最终目的地,革兰氏阴性菌的外膜蛋白 (OMP) 从胞质溶胶开始进行一段多事的旅程。多种分子机器、分子伴侣、蛋白酶和其他酶促进 OMP 的易位和组装。这些助手通常联合,通常是短暂的,形成大的蛋白质组装体。
由于捕获和表征蛋白质-蛋白质相互作用(PPI),尤其是瞬态相互作用方面的实验挑战,它们还没有被很好地理解。使用 AF2Complex,佐治亚理工学院的研究人员引入了一个高通量、深度学习管道来识别大肠杆菌细胞包膜内的 PPI,并将其应用于 OMP 生物发生途径中的几种蛋白质。
在通过筛选约 1500 种包膜蛋白获得的最有信心的命中物中,研究人员不仅发现了预期的相互作用,还发现了具有深远影响的意想不到的相互作用。随后,该团队预测这些蛋白质复合物的原子结构。这些通常具有高置信度的结构解释了实验观察结果,并导致了关于伴侣如何协助从易位子中出现的新生前体 OMP 的机制假设,另一个伴侣如何防止它聚集并停靠到 β 桶组装端口,以及蛋白酶如何进行质量控制。
该研究以「Deep learning-driven insights into super protein complexes for outer membrane protein biogenesis in bacteria」为题,于 2022 年 12 月 28 日发布在《eLife》。

革兰氏阴性菌特有的结构成分是外膜 (OM),由不对称双层中的脂多糖和磷脂组成,嵌入脂蛋白和跨膜 β-桶蛋白。后一组 OM 蛋白 (OMP) 发挥着重要的功能作用,例如,通过其跨膜 β-桶孔蛋白与环境交换小分子。OMP 由胞质核糖体合成,跨内膜 (IM) 转运,最终通过 OMP 生物发生途径递送至 OM。
OMPs的易位和折叠涉及许多蛋白质,包括协同形成多个复合物的必需蛋白质和辅助蛋白质。两个核心复合体是 SecYEG translocon 和 β-桶组装机。SecYEG 是一种异源三聚体,由分泌通道 SecY 和两个额外的亚基 SecE 和 SecG 组成,锚定在 IM 上,负责将大多数细胞包膜蛋白移动到 IM 中。周质蛋白和 OM 蛋白通过依赖于 SecA 的易位途径进入 SecY 中的通道。它们拥有被 SecA 识别的信号肽,SecA 将蛋白质底物插入 SecY 并利用 ATP 水解产生的能量通过通道为其提供动力。位于底物 N 末端的信号肽包含一个疏水片段,一旦它离开 SecY 的侧门,该片段就会折叠成跨膜 α 螺旋。为了从 IM 中释放底物,I 型信号肽酶 (SPase I) 会裂解信号肽。此时,周质蛋白已到达其目的地,但 OMP 在伴侣蛋白 SurA 或 Skp 的护送下,继续其前往 OM 中隐藏的 BAM 的旅程。五个亚基构成 BAM,其中 BamA 在折叠 β-桶和释放成熟产物中起主要作用。
关于 OMP 生物发生途径有许多悬而未决的问题。在大肠杆菌中,SecYEG 转位子募集额外的蛋白质,如 SecA、SecDF、YidC 和 PpiD,以在不同情况下形成各种超复合物。研究人员不知道这些超复合物所有成员的身份,更不用说它们的原子结构了。SurA 在陪伴新生的 OMP 方面发挥着重要作用。它如何处理基板并将其交付给 BAM?同样,BAM 会招募额外的帮手,例如 BepA,但他们如何协同工作?
通过实验回答这些问题具有挑战性。近期,深度学习方法在预测蛋白质复合物的结构方面取得了巨大进展。在这里,佐治亚理工学院的研究人员使用一种这样的方法来解决上述问题。该方法的核心是基于 AlphaFold2 (AF2) 构建的 AF2Complex。使用 AF2Complex,研究人员结合了蛋白质-蛋白质相互作用 (PPI) 的虚拟筛选和超复合物建模,并将该策略应用于 OMP 生物发生途径中的几种重要蛋白质。

图示:大肠杆菌超级转运子 SecYEG/PpiD/YfgM。(来源:论文)通过将其应用于 OMP 生物发生途径中的几个关键蛋白质,该团队已经确定了它们的功能伙伴在每个查询筛选 PPI 的约 1450 种蛋白质的前 1% 排名中。由于顶级预测背后的高置信度结构,人们可以理解许多实验现象,特别是体内定点照片交联数据。例如,从 SecYEG 或 BAM 超复合物中发现的交联产物可以用预测的结构中揭示的直接物理相互作用来解释。此外,先前推测的构象被捕获为 SurA 和 BepA。最重要的是,这些揭示的原子结构提出了 OMP 生物发生途径各个步骤的机制假设。

图示:拟议的机制涉及大肠杆菌中的外膜蛋白 (OMP) 生物发生途径。(来源:论文)
一个意想不到的发现是 DsbA/PpiD 相互作用。众所周知,DsbA 关键地将其二硫键转移到由 SecYEG 易位的新生多肽,但易位子如何与 DsbA 相互作用?SecYEG/PpiD/YfgM/DsbA 的预测超复合物提供了一个令人信服的答案,原因有以下三个:
首先,DsbA 在 PpiD 上的瞬态驻留在易位子上显着提高了其效率,而不是在拥挤的周质中进行随机搜索。其次,它预测了 PpiD 的 PPIase 域的功能,该功能神秘地缺乏 PPIase 功能。第三,由于 PpiD 锚定在 IM 上,它可以使 DsbA 靠近 DsbB,DsbB 是一种回收 DsbA 的 IM 蛋白。值得注意的是,另外两个大肠杆菌分子伴侣 SurA 和 FkpA 各自拥有两个 PPIase 结构域,并且 AF2Complex 预测 DsbA 与 FkpA 相互作用(iScore = 0.47 对 PpiD 为 0.55)但不与 SurA 相互作用(iScore = 0.03)。
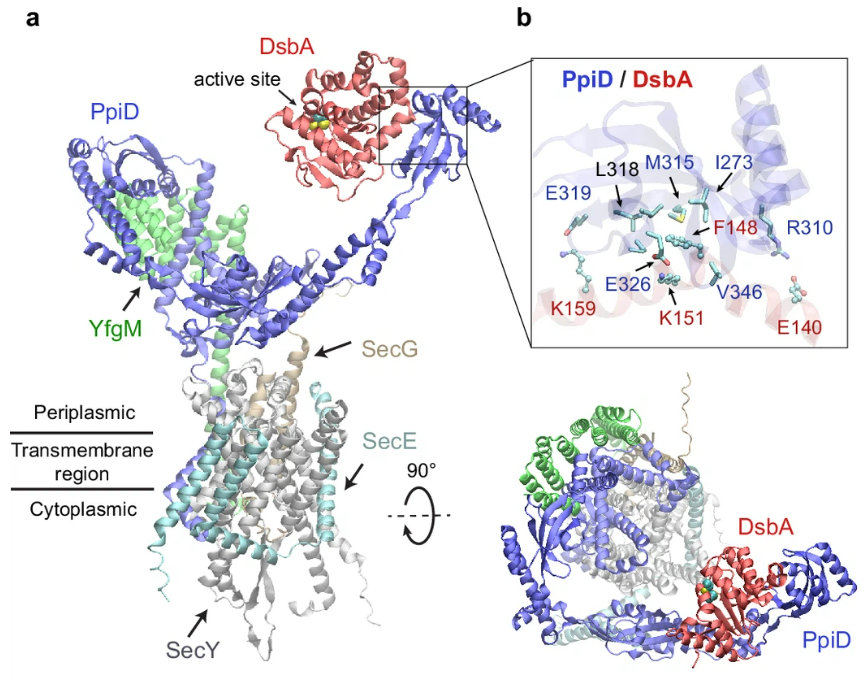
图示:SecYEG/PpiD/YfgM/DsbA 超复合体的结构模型。(来源:论文)
有趣的是,AF2 神经网络可以模拟伴随分子伴侣的部分展开的多肽,即使 AF2 是在折叠蛋白上训练的。该团队获得了看起来至少部分是物理的结构模型。一个例子是 PpiD/YfgM/LepB/proOmpA 模型,其中 proOmpA 被肽酶 LepB 切割。OmpA 的裂解丙氨酸距离 LepB 的催化三联体约 4 Å。考虑到只有 LepB 的 apo 或抑制剂结合结构可用,这个计算模型意味着 AF2 已经学习了物理表征。

图示:存在和不存在 OmpA 底物时 SurA 的结构模型。(来源:论文)
更有趣的例子是 SurA/OmpA 模型,其中 OmpA 的周质结构域折叠,但 β 桶结构域完全展开并松散地包裹在 SurA 周围。预测的结构与 SecB 结合的未折叠多肽的 NMR 结构相呼应,SecB 是一种参与 SecA 依赖性易位途径早期阶段的胞质伴侣。尽管由于弱相互作用导致置信度低,但预测的结构描绘了 SurA 如何防止 OmpA 聚集的图片。此外,由于它通过相对较少的分子间接触转运 OmpA,因此将 OmpA 从 SurA 解离所需的自由能很小。尽管有这些考虑,但研究人员表示这些预测的结构模型中可能存在伪影。
这些结果强化了这样一种观念,即深度学习是探索蛋白质构象集合和揭示生物系统分子机制的有前途的方法。先进的 AF2 深度学习模型、有效的 PPI 排名指标和针对大规模筛选优化的工作流程相结合,生成了蛋白质复合物的照明结构。
当然,这里没有描述其他有信心的 PPI 预测。虽然有些是混杂的相互作用,或者在物理上可能但在生物学上无关,或者只是错误的,但可能还有一些功能性相互作用有待探索。更一般地说,这些结果是基于深度学习的策略的一个例子,有助于阐明复杂生化途径的机制方面。
论文链接:https://elifesciences.org/articles/82885
相关报道:https://phys.org/news/2023-01-deep-tool-microscope-protein-interactions.html
人工智能 × [ 生物 神经科学 数学 物理 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。