
Prime editing是一种多功能的基因组编辑工具,但需要对Prime editing的pegRNA进行实验优化才能获得较高的编辑效率。实现高的prime editing速率通常需要通过手动测试多个pegRNA设计来优化pegRNA的特定部分,这是一个费力且耗时的过程。
近日来自瑞士苏黎世大学Michael Krauthammer团队和Gerald Schwank团队在国际知名期刊Nature Biotechnology杂志上在线发表了名为Predicting prime editing efficiency and product purity by deep learning的研究论文,该团队进行了高通量筛选,分析了高度多样化的13349个人类致病突变(包括碱基替换、插入和缺失)上92423个pegrna的编辑结果。基于此数据集,识别了影响prime editing的特征,并训练了PRIDICT。PRIDICT可靠地预测了所有小型基因改变的编辑率,预期编辑和非预期编辑的Spearman’s R分别为0.85和0.78。在内源性编辑位点和外部数据集上验证了PRIDICT,结果表明,PRIDICT得分高(>70)的pegrna与低(<70)的pegrna在体外不同类型的细胞中(12倍)和体内肝细胞中(10倍)的prime editing编辑效率显著提高,突出了PRIDICT在基础和转化研究应用中的价值。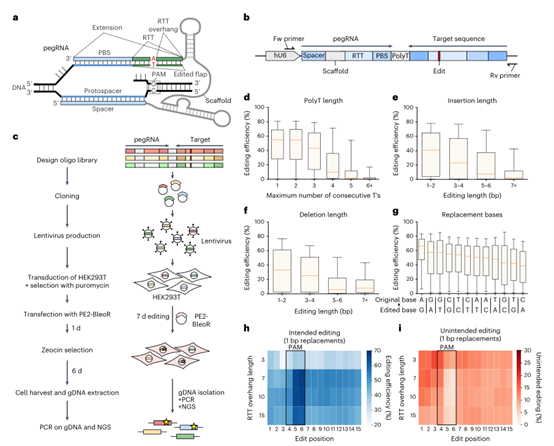
图1:高通量筛选prime editing系统的决定因素
为了评估高通量筛选的prime editing效率,该团队生成了一个“自靶向”慢病毒库,其中每个pegRNA都与其相应的目标位点配对。该文库由119,701个pegrna组成,针对不同的14,238个人类致病突变,包括所有可能的单碱基替换以及插入(最多24 bp)和删除(最多13bp)。为了测试不同pegRNA设计的效果,该团队针对每种疾病突变使用多个pegRNA; 它们包含多达三种不同的间隔序列,13 bp的恒定PBS和四种不同的RTTs。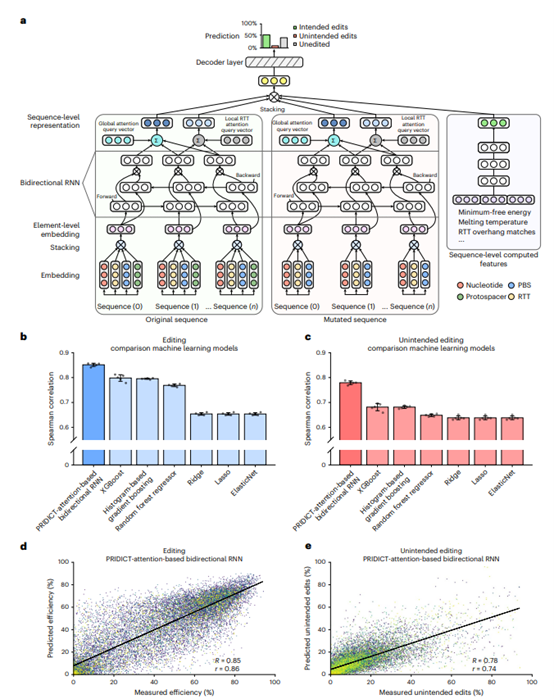
图2:开发机器学习算法来预测prime editing速率
接下来,该团队尝试使用prime editing结果数据集来开发能够预测主要编辑效率和意外(非预期)编辑率的机器学习模型。因此,该团队将数据集分成80%的训练序列和20%的测试序列。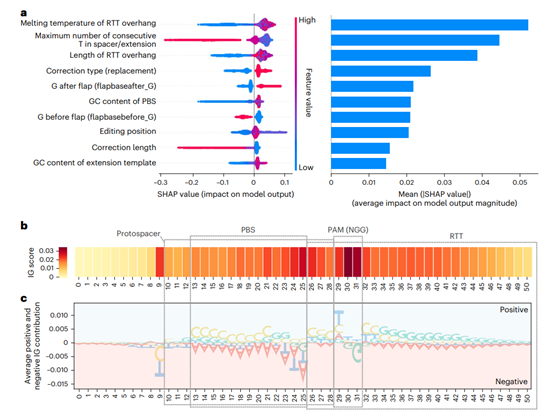
图3:pegRNA特征对机器学习模型的影响
对XGBoost模型测试数据集的SHAP分析可视化了影响编辑预测结果的前十大特征。SHAP值越高,编辑预测越高。对双向RNN模型中个体序列位置的重要性分析。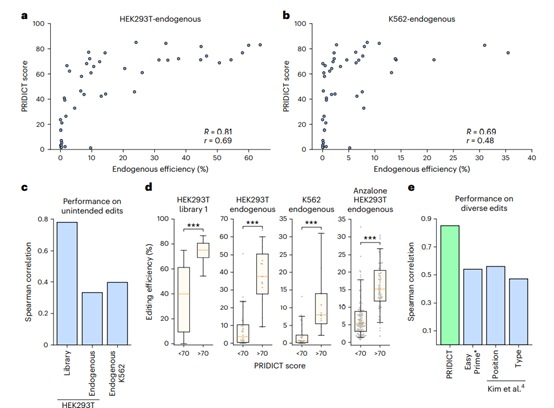
图4:PRIDICT在内源性位点和外部数据集上的验证
在prime editing预测工具的一个重要方面是在不同的实验设置下产生的数据集的适用性。因此,该团队评估了PRIDICT在以下数据上的性能:(1)在内源性基因组位点上,(2)在不同的细胞系中,(3)在其他实验室中产生。

图5:错配修复(MMR)抑制和pegRNA稳定模型验证
原文链接:
https://www.nature.com/articles/s41587-022-01613-7

为了能更有效地帮助广大的科研工作者获取相关信息,植物生物技术Pbj特建立微信群,Plant
Biotechnology
Journal投稿以及文献相关问题、公众号发布内容及公众号投稿问题都会集中在群内进行解答,同时鼓励在群内交流学术、碰撞思维。为了保证群内良好的讨论环境,请先添加小编微信,扫描二维码添加,之后我们会及时邀请您进群。小提示:
添加小编微信时及
进群后请务必备注学校或单位+姓名,PI在结尾注明,我们会邀请您进入PI群。

