*仅供医学专业人士阅读参考


年后看《流浪地球2》时,朋友突然问我:技术都到这儿了,不能利用550W来设计点儿新蛋白,治疗这些辐射病吗?
我:啊?(内心:你小子看个电影还挺勤学好问哈?)
回到此刻的现实中,利用人工智能来“定制”全新蛋白质,确实已经不是什么未曾设想的道路。
近日,顶刊《自然》发表了蛋白质学领域里程碑式的最新研究成果。
美国华盛顿大学David Baker教授领导的研究团队,开发了一种基于深度学习的人工智能算法——Family-wide Hallucination,用于蛋白质结构预测和设计。
在这项研究中,他们成功利用Family-wide Hallucination设计出全新的荧光素酶LuxSit,并完成将其从头合成和性能优化。与天然荧光素酶相比,LuxSit表现出了更加优越的活性、稳定性、底物特异性,能够有效催化底物发光[1]。
Family-wide Hallucination的研发,对于从头设计蛋白质具有重大意义。

论文首页截图
作为蛋白质学领域的杰出科学家和领军人物,David Baker在蛋白质折叠预测、蛋白质与小分子结合、自组装蛋白纳米颗粒、蛋白质设计等方向都有着深入研究和众多贡献[2]。
早在1998 年时,David Baker及其团队就开发出Rosetta平台来预测蛋白质结构,基于物理学原理,对蛋白质的构象进行能量最小化计算,以预测最稳定的三维结构,并在后续的时间里不断拓展Rosetta的应用,如蛋白质设计。
2008年时,David Baker实验室还创造性地开发了蛋白质结构预测和设计的“硬核”小游戏Foldit,动员科研人员、非科研人员一起参与到这个领域[3]。
近几年,他们研发的深度算法RoseTTAFold与备受关注的AlphaFold 2并驾齐驱,仅根据氨基酸序列即可快速准确地预测蛋白质结构,成为迄今为止最准确的蛋白质结构预测算法之一 [4]。
去年,他们推出的深度学习算法ProteinMPNN ,可快速根据给出的蛋白质三维结构“逆推”出可能的氨基酸序列,序列恢复率达52.4%(Rosetta为32.9%)[5,6]。

[5]
虽然蛋白结构预测方面有了RoseTTAFold、AlphaFold 2等人工智能算法的相助,但要想实现从头设计全新蛋白质,仍然是个费时费力的艰巨任务。目前,即使是借助机器学习算法,蛋白质设计也主要是把自然界中现有的蛋白质拿来改造,以进行优化或获得特定功能,而非真正地设计并创造一个全新的蛋白质。
致力于在蛋白质学领域掀起人工智能革命的David Baker实验室,始终没有停下探索的脚步。在这项研究中,他们尝试利用深度学习算法Family-wide Hallucination,为荧光素DTZ“量身定做”一个全新的荧光素酶。
萤光素酶是一种通过酶促氧化反应使其底物荧光素产生生物发光的酶,常用于报告基因检测来研究基因表达、蛋白质定位、蛋白质相互作用等生物学问题。不过,现有的荧光素酶存在一些限制,例如亮度不够高、对氧气敏感、发光波长有限等,影响其在特定场景下的应用,设计新的荧光素酶可以为生物学研究和应用提供更好的工具和平台。
在从头设计荧光素酶时,如何将催化位点和底物结合口袋引入蛋白质主链骨架是个难点,而且已知的蛋白质主链骨架数量也较为有限。这次,David Baker团队将组装过程分成几步来进行。
首先,他们将现有荧光素酶家族的结构数据与蛋白质序列信息,输入给Family-wide Hallucination。通过深度学习,Family-wide Hallucination将这些已知的“知识”进行转化,生成上千个具有DTZ底物结合口袋样结构的、全新的、理想的蛋白质三维结构骨架,及其相应的氨基酸序列。
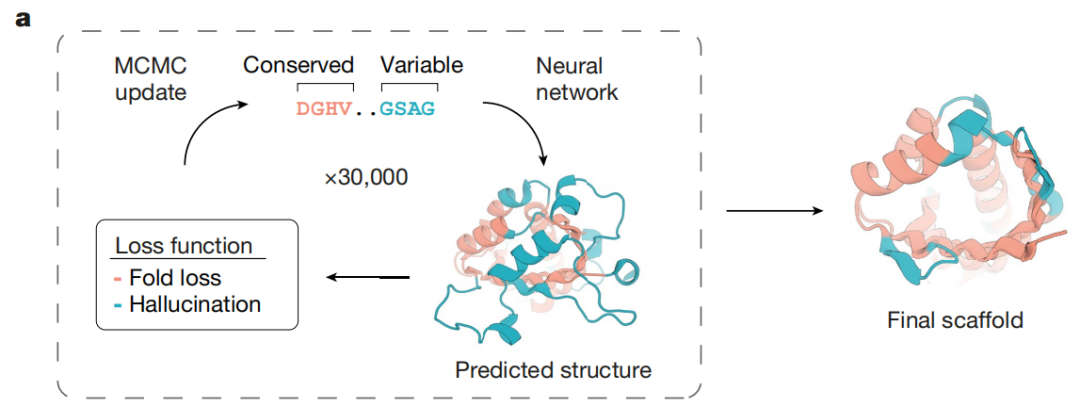
先让Family-wide Hallucination给出大量全新蛋白框架
随后,基于目标荧光素酶可能的反应机制和酶催化反应过渡态,研究者们设计了催化位点,并引入到由Family-wide Hallucination生成的这些框架中。借助Rosetta软件,他们对这些组装的蛋白结构和功能进行预测和优化,并根据配体结合能、形状互补性等进行筛选,得到7648种候选蛋白。
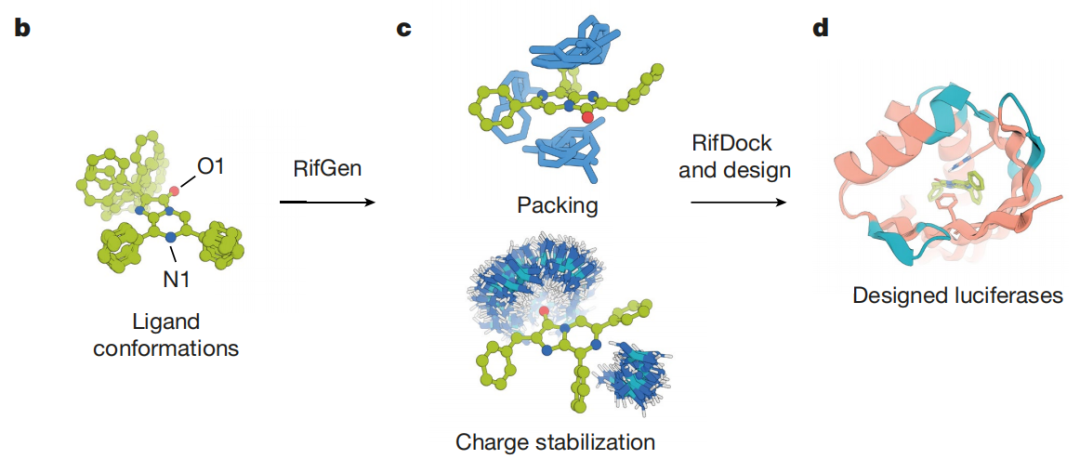
研究人员设计催化位点后放进去,以及其它一些序列、结构优化
接下来,研究者们合成并在大肠杆菌中表达这些候选蛋白。经过克隆与活性鉴定后,最终有3个候选蛋白杀出重围,即有效的、以DTZ为底物的荧光素酶。
其中,活性最强的荧光素酶被研究者们命名为LuxSit(拉丁语“让光存在”)。LuxSit比已有的任何荧光素酶都小,仅有117个氨基酸残基(13.9 kDa),可在大肠杆菌中高表达,为可溶性单体,且耐高温(熔解温度高于95℃),作用于DTZ时荧光发射峰位于480nm左右。
进一步性能优化后,研究者们得到非常高效、具有高度底物特异性的LuxSit-i,催化底物后产生的发光肉眼可见,比自然界中存在的海肾荧光素酶 (RLuc)催化底物发生的生物发光还要亮。
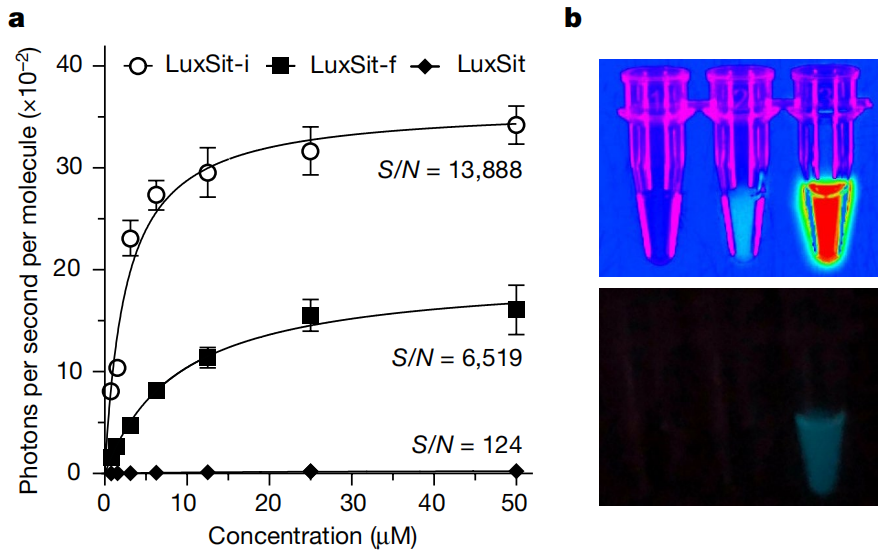
LuxSit和优化过的LuxSit-i、LuxSit-f,其中LuxSit-i性能最好
不仅如此,研究人员再次利用相同的方式从头设计了另一组以荧光素h-CTZ为特异性底物的荧光素酶。不过这次,他们借助了更多人工智能的力量,在对组装上催化位点的蛋白质结构进行优化时,研究者们使用了深度学习算法AlphaFold 2和ProteinMPNN,得到46种候选蛋白。
经鉴定,这46种候选蛋白中有2种为有效设计。这相比于首次尝试,从头设计荧光素酶的成功率增加了十倍,从 0.04%(DTZ:3/7648)提高到4.35%(h-CTZ :2/46)。研究者们认为,这一改进可能是得益于设计LuxSit时所得到的的经验,以及 ProteinMPNN的使用。
总体来说,David Baker团队研发的深度学习算法Family-wide Hallucination,能够将已知的蛋白质结构和功能信息,用于生成全新的、拥有目标性能的、理想的蛋白酶结构。在这项研究中,他们利用Family-wide Hallucination从头设计了全新的荧光素酶,具有体积小、高度稳定、在细胞中表达良好、高度特异性等优点。
这项研究成果证明,基于深度学习的算法能够为蛋白质工程领域带来翻天覆地的改变,或可极大促进酶制剂、生物传感器等生物制品或技术的研究和应用。
参考文献:
[1]https://www.nature.com/articles/s41586-023-05696-3#citeas
[2]https://www.bakerlab.org/
[3]https://fold.it/
[4]Baek, M., DiMaio, F., Anishchenko, I., Dauparas, J., Ovchinnikov, S., Lee, G. R., Wang, J., Cong, Q., Kinch, L. N., Schaeffer, R. D., Millán, C., Park, H., Adams, C., Glassman, C. R., DeGiovanni, A., Pereira, J. H., Rodrigues, A. V., van Dijk, A. A., Ebrecht, A. C., Opperman, D. J., … Baker, D. (2021). Accurate prediction of protein structures and interactions using a three-track neural network. Science (New York, N.Y.), 373(6557), 871–876. https://doi.org/10.1126/science.abj8754
[5]Dauparas, J., Anishchenko, I., Bennett, N., Bai, H., Ragotte, R. J., Milles, L. F., Wicky, B. I. M., Courbet, A., de Haas, R. J., Bethel, N., Leung, P. J. Y., Huddy, T. F., Pellock, S., Tischer, D., Chan, F., Koepnick, B., Nguyen, H., Kang, A., Sankaran, B., Bera, A. K., … Baker, D. (2022). Robust deep learning-based protein sequence design using ProteinMPNN. Science (New York, N.Y.), 378(6615), 49–56. https://doi.org/10.1126/science.add2187
[6]https://www.bakerlab.org/2022/09/16/proteinmpnn-excels-at-creating-new-proteins/


本文作者丨张艾迪