大家好,欢迎来看今天的文献解读!今天我们带来一篇非常有意思的文章,标题为“Development of a machine learning-based radiomics signature for estimating breast cancer TME phenotypes and predicting anti-PD-1/PD-L1 immunotherapy response”,发表在Breast Cancer Research上。这篇文章探讨了如何利用机器学习和放射组学技术来预测乳腺癌患者对免疫治疗的反应,特别是在肿瘤微环境(TME)表型的评估上。随着乳腺癌在女性中发病率的上升,如何提高免疫治疗的效果和预测患者的临床反应显得尤为重要。想知道这篇研究带来哪些新发现和潜在的应用吗?那就继续往下看吧!
这项研究的亮点在于,作者构建了一个基于放射组学的生物标志物,用以预测乳腺癌患者的TME表型,并通过多组学数据进行独立验证。研究采用了来自Cancer Genome Atlas (TCGA)和Cancer Imaging Archive (TCIA)的多组数据,通过结合RNA-seq数据与动态对比增强MRI图像,成功建立了一个放射组学特征签名。这一方法首次将机器学习应用于乳腺癌TME表型的评估,并在多个独立的验证集中显示出良好的预测性能。这一研究不仅为乳腺癌的个体化治疗开辟了新的方向,也为未来的临床研究提供了新的思路。
数据来源
这项研究的数据主要来源于Cancer Genome Atlas (TCGA)和Cancer Imaging Archive (TCIA)数据库,包括对1089名乳腺癌患者的转录组数据,以及94名患者的动态对比增强MRI图像。这些数据支持了放射组学特征签名的构建与验证。此外,研究还利用了来自广东省人民医院的免疫表型队列和免疫治疗队列,以评估该签名的临床预测能力。所有数据均经过伦理委员会的批准,并严格遵循了赫尔辛基宣言的伦理标准。
题目: Development of a machine learning-based radiomics signature for estimating breast cancer TME phenotypes and predicting anti-PD-1/PD-L1 immunotherapy response
杂志: Breast Cancer Research
研究设计与数据来源
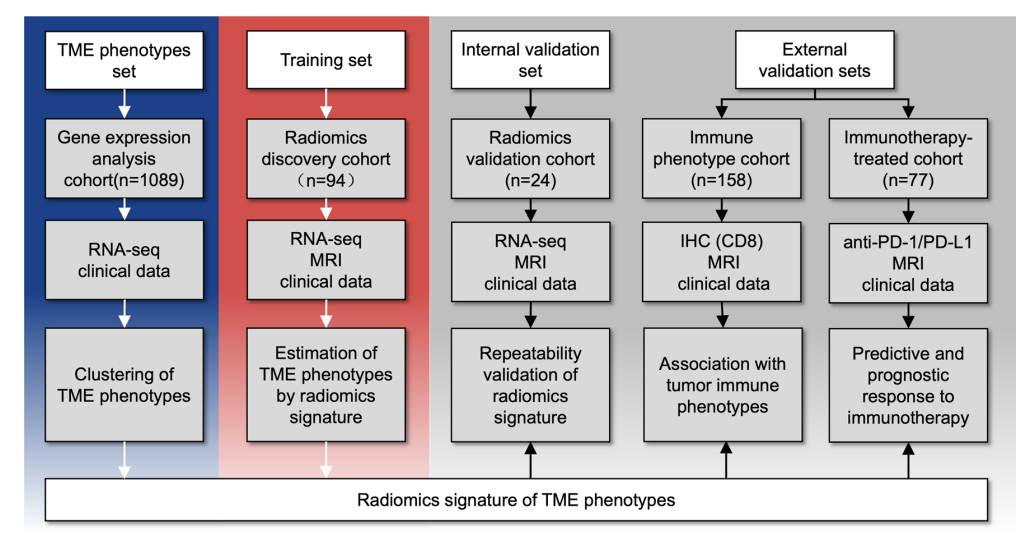
在本研究中,图1清晰展示了整体设计和不同队列的组成。Gene expression analysis cohort结合了RNA-seq和临床数据,旨在开发肿瘤微环境(TME)表型。Radiomics discovery cohort作为训练集,用于训练TME表型的Radiomics特征,而Radiomics validation cohort则负责进行重复性验证。同时,Immune phenotype cohort根据CD8免疫组化结果将患者分为免疫炎性和免疫沙漠瘤两种类型。最后,Immunotherapy-treated cohort接受抗PD-1/PD-L1治疗,以预测免疫治疗的预后反应。这一设计框架为后续实验提供了明确的指导,帮助研究者深入分析免疫表型与免疫治疗反应之间的关系,从而提升预测模型的临床适用性。
肿瘤区域的分割与特征提取

图2展示了肿瘤区域的分割过程,采用ITK-SNAP软件手动标记MRI图像中的肿瘤边界,并通过Python进行自动识别,形成肿瘤的核心区域及其周围的2mm外周区域,最终形成4mm的肿瘤浸润扩展环。这种方法确保了肿瘤边界的准确性,为后续的特征提取奠定了基础。结合人工标记与自动化处理,研究者能够精确获取与TME相关的特征数据,确保结果的可靠性与一致性。
TME表型的聚类分析及预后价值
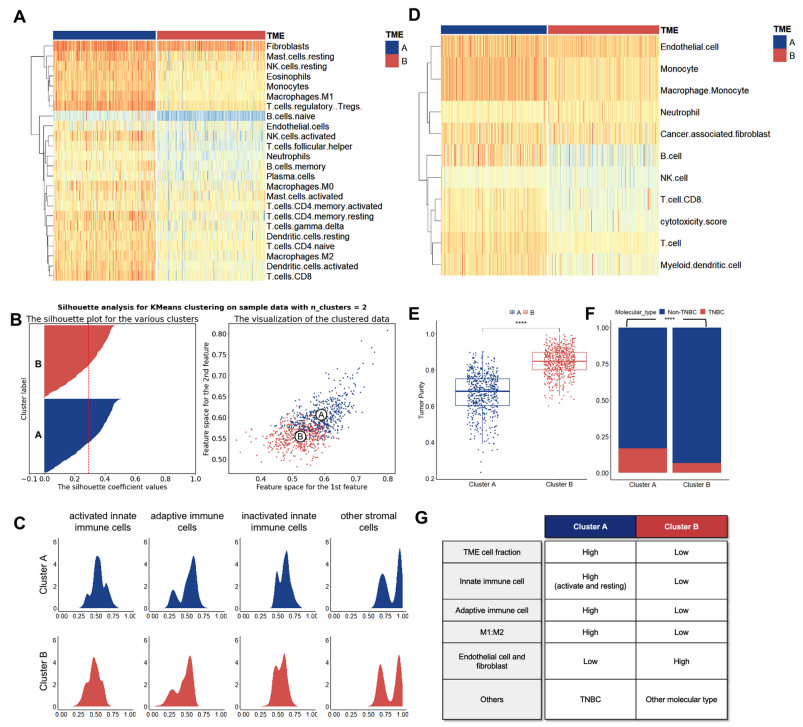
在图3中,乳腺癌TME表型的景观通过k-means聚类分析被分为两个异质性集群:Cluster A(免疫炎性集群)和Cluster B(免疫沙漠集群)。Cluster A显示出较高的免疫细胞浸润,而Cluster B则表现出较低的细胞浸润水平。这一分析对于理解乳腺癌生物学特性和治疗反应至关重要,强调了TME在乳腺癌预后中的重要性。

进一步的分析在图4中体现,Kaplan-Meier生存曲线表明Cluster A的整体生存率明显优于Cluster B,免疫细胞浸润程度与生存率呈正相关。这一结果显著表明TME的不同表型与患者预后密切相关,为未来的治疗策略提供了重要参考。
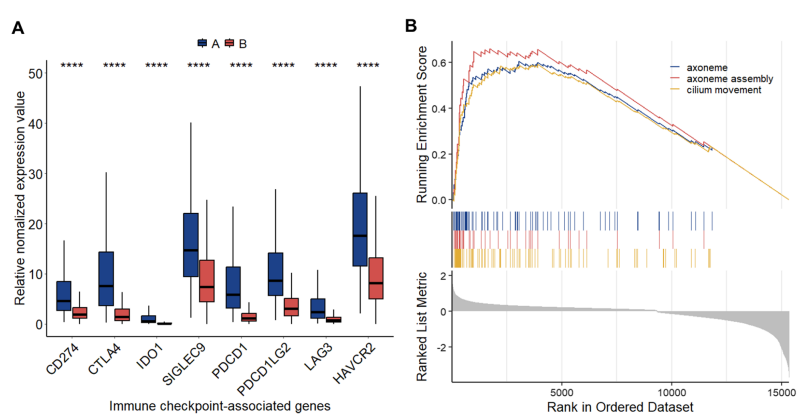
图5则探讨了乳腺癌中免疫调节因子的激活情况,评估了包括CD274、CTLA4等在内的免疫检查点相关基因,结果显示Cluster A中的免疫检查点基因表达显著高于Cluster B,GSEA分析进一步揭示了Cluster A中与免疫反应相关的通路的富集。这些发现强调了免疫逃逸机制的重要性,并为未来的治疗策略提供了潜在靶点。
Radiomics特征的预测能力与临床应用
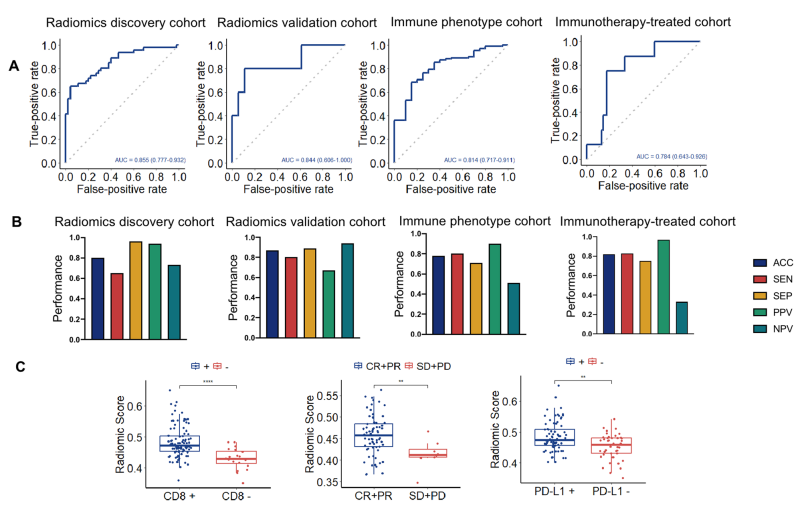
在图6中,基于Radiomics的TME表型预测性能得到了验证。该图展示了Radiomics signature在训练集和三个独立验证集中的AUC结果,表现出良好的预测能力。通过混淆矩阵的评估,研究者分析了模型的敏感性和特异性,表明该签名在不同队列中均表现出色,强调了Radiomics在个性化治疗中的重要性。
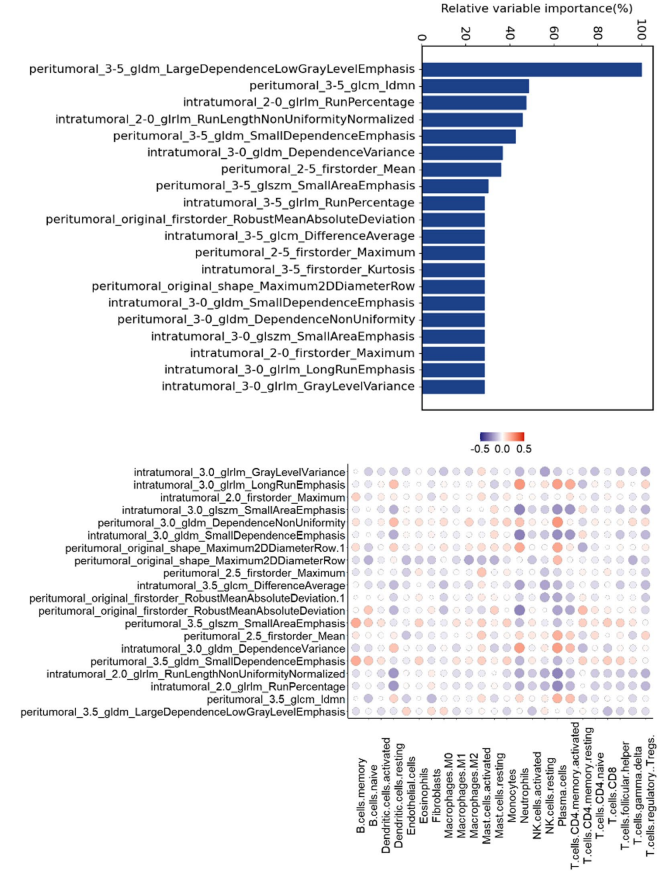
最后,图7探讨了Radiomics特征与24种免疫细胞及基质细胞浸润的关系,利用随机森林算法评估特征的重要性,结果显示大多数细胞类型与Radiomics特征之间存在显著关联。这一分析为理解Radiomics特征与TME之间的相互作用提供了重要视角,并为临床应用Radiomics进行免疫治疗反应预测奠定了理论基础,助力提高个体化治疗的效果。
总结
这篇文章探讨了利用机器学习和放射组学技术来评估乳腺癌患者的肿瘤微环境(TME)表型,并预测其对抗PD-1/PD-L1免疫治疗的反应。研究通过分析来自TCGA和TCIA的多组数据,首次构建了放射组学特征签名,旨在提高乳腺癌的个体化治疗效果。研究表明,TME表型的聚类分析与患者预后密切相关,免疫炎性集群的生存率明显优于免疫沙漠集群。此外,放射组学特征在多个验证集中展现出良好的预测能力,为未来的临床研究提供了新的思路,强调了放射组学在个性化免疫治疗中的潜在应用价值。

