本文用 APEX 模型从全球毒液库筛选386种新抗菌肽,91.4%验证有效。
美国宾夕法尼亚大学的研究团队整合四大毒液数据库构建全球毒液数据库,应用了一种名为 APEX 的序列到功能深度学习模型,专门用于系统性挖掘毒液蛋白质组中的潜在抗菌候选物,最终筛选出 386 条具有抗菌潜力且与已知 AMPs 序列相似度低的候选肽。
1928 年青霉素的发现,为人类对抗细菌感染开启了全新纪元。这类抗生素显著提升了人类应对感染性疾病的能力,但随着抗生素在医疗、农业等领域的广泛应用乃至过度使用,细菌的耐药性问题逐渐显现,并以惊人的速度恶化。
进入 21 世纪,抗生素耐药性已成为全球公共卫生领域的重大挑战。世界卫生组织的数据显示,每年全球约有 500 万人直接或间接死于耐药性感染,而这一危机的核心推手是抗生素耐药性病原体的快速滋生与蔓延。其中,被 WHO 列为「优先病原体」的革兰氏阴性菌尤为棘手,它们凭借极强的耐药进化能力,在与人类的「抗菌战役」中持续占据上风。更严峻的是,过去数十年间,新型抗生素的研发陷入停滞:高昂的研发成本、漫长的临床试验周期,让众多制药企业对这一领域望而却步,新抗生素的问世数量锐减,远远跟不上耐药菌的进化速度。
在这一困境下,科学家们开始将目光投向自然界中一个充满潜力的「宝库」——动物毒液。历经数百万年的自然进化,动物毒液孕育了极其丰富的分子多样性,其中包含的生物活性肽和蛋白质,既能与广泛的生物靶点相互作用,更展现出显著的抗菌活性。事实上,毒液在生物医学领域的应用早有成功先例——从锥螺毒液中提取的齐考诺肽(商品名 Prialt®),已成为治疗慢性疼痛的重要镇痛药;从蛇毒中研发的卡托普利,则是临床常用的降压药物。这些案例为毒液在抗菌领域的探索提供了重要启示。
在此背景下,美国宾夕法尼亚大学的研究团队以高通量方式从全球毒液蛋白中筛选抗菌候选肽,从全球动物毒液中挖掘出 386 种结构全新的抗菌肽,其中 91.4% 的实验验证分子展现出强效抗菌活性,为新一代抗生素的发现提供了突破性思路。
相关研究成果以「Computational exploration of global venoms for antimicrobial discovery with Venomics artificial intelligence」为题,发表于 Nature Communications。
研究亮点:
* 该研究构建全球毒液数据库并生成加密肽候选库,共计收录 16,123 条毒液蛋白,并生成了超过 4 千万条毒液加密肽;
* 该研究使用深度学习模型 APEX 预测每条肽对 34 种细菌的最小抑菌浓度,最终筛选出 386 条具有抗菌潜力且与已知 AMPs 序列相似度低的候选肽;
* 研究团队从 386 条 VEPs 中合成并测试了 58 条肽,其中 91.4%(53 条)对至少一种病原菌具有显著抑制效果。
论文地址:
https://www.nature.com/articles/s41467-025-60051-6[*](https://arxiv.org/pdf/2507.09466*)
数据库:收集 1.6 万种毒液蛋白质
该研究为了寻找新的抗菌候选物,从 ConoServer、ArachnoServer、ISOB 和 VenomZone 4 个数据库中收集了毒液蛋白质序列,总共收集到了 16,123 种毒液蛋白质。其中,ConoServer 专注于芋螺肽,ArachnoServer 包含蜘蛛毒素,ISOB 是孟加拉国本土蛇类毒液数据库,VenomZone 则是从 UniProtKB 整理而来,涵盖了蛇、蝎子、蜘蛛、芋螺、海葵和昆虫等 6 个分类群的蛋白质,在该研究中由 UniProt 代表。
这些数据集具有各自独特的特点和作用。如下图所示,通过对不同数据库的物种重叠分析发现,UniProt 包含的独特物种数量最多,达到 699 个,这表明它具有更广泛的蛋白质多样性。而 ConoServer 和 ArachnoServer 包含的独特子集相对较小,ISOB 则没有独特物种。这种多样性的存在使得研究人员能够从多个角度挖掘毒液中的抗菌肽,综合不同来源的蛋白质信息,提高发现新型抗菌肽的可能性。
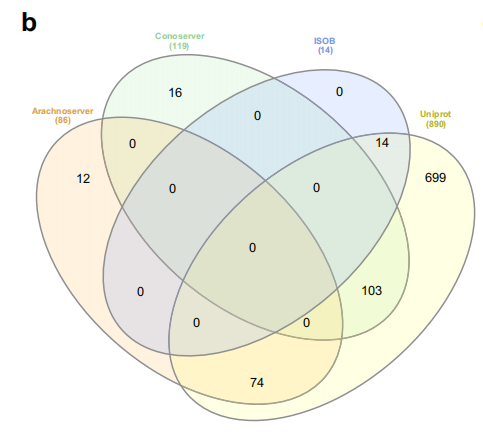
四大数据库的 Venn 图
如下图所示,研究人员对这些毒液蛋白质进行处理,生成了超 4 千万个毒液加密肽(VEPs)。然后使用 APEX 深度学习模型对这些肽的抗菌活性进行预测,预测每个肽对细菌菌株的最小抑菌浓度(MIC)值,并以中位数 MIC 作为抗菌活性的衡量标准。通过进一步筛选,研究人员最终确定了 7,379 个中位数 MIC ≤ 32 μmol L⁻¹ 的 VEPs,再经过与已知抗菌肽的序列相似性过滤,得到了 386 个候选物。这些候选物在结构和功能上与已知抗菌肽不同,为后续的实验验证提供了有价值的目标。
APEX 模型预测:多源毒液蛋白质组数据整合与抗菌候选肽的筛选流程
APEX 模型是一款专注于抗菌肽潜力挖掘与预测的深度学习工具,其核心优势在于具备细菌菌株特异性抗菌活性预测能力。经过系统训练,该模型可针对 34 种不同细菌菌株,精准预测肽的最小抑菌浓度(MIC)值——这一指标是衡量抗菌剂效力的关键参数。
模型的训练数据集涵盖两部分:内部积累的肽序列数据,以及从 DBAASP 数据库获取的公开抗菌肽(AMPs)信息。这种多元化的数据支撑,使 APEX 在解析肽序列与抗菌活性之间的复杂映射关系时表现突出,为后续筛选奠定了扎实的算法基础。
在架构设计上,APEX 采用多目标任务模型框架,基于「三步走」策略,从毒液蛋白中筛选抗菌肽候选分子。首先,用滑动窗口法(sliding window method)构建肽库,截取长度在 8 至 50 个氨基酸之间的片段;其次,利用 APEX 模型预测这些肽对不同细菌菌株的最小抑菌浓度(MIC);最后,依据序列相似性进一步精选,获得一组结构新颖且活性优异的毒液加密肽(VEPs)。
抗菌肽(AMPs)的挖掘框架
这种设计使其在处理海量毒液加密肽(VEPs)时,能够精准识别出具有潜在抗菌活性的候选分子。相比传统依赖实验筛选的方式,APEX 大幅提升了抗菌肽的发现效率,同时降低了对资源密集型生化测定的依赖,为研究人员提供了一种更快捷、经济的筛选路径。
在实际应用中,使用 APEX 模型需要一定的技术准备。研究人员需要搭建一个合适的运行环境,包括安装 Python 3.9以及特定版本的 PyTorch 和一系列依赖库,如 numpy、scipy 和 matplotlib 等。一旦环境搭建完成,将候选肽序列保存在文本文件中,通过运行指定的命令,模型能够生成一个包含 MIC 预测结果的 CSV 文件。通过对这些预测结果的分析,研究人员可以迅速锁定具有较高抗菌潜力的肽段,进而进行深入的实验验证。
VEPs 的抗菌活性验证与作用机制解析
为验证毒液加密肽(VEPs)的抗菌活性,研究团队开展了一系列实验。在抗菌活性测定中,58 种 VEPs 接受了针对多种病原菌的测试,结果如下图所示,其中 53 种(占比 91.4%)对至少一种病原菌表现出活性。尤为值得关注的是,所有来自 ArachnoServer 数据库的 VEPs 均展现出抗菌活性,这一结果有力印证了该类肽段强大的抗菌潜力。

毒液中活性抗菌剂的 Heatmap
不过,部分来自 UniProt 数据库的 VEPs 效力相对有限。例如,UniprotKB-2 未表现出任何抗菌活性,UniprotKB-6 和 UniprotKB-11 仅对粪肠球菌有抑制作用。进一步分析揭示,这些低活性肽段普遍具有较低的疏水性和净电荷——这一发现提示,疏水性与净电荷在促进肽段与细菌膜的相互作用中扮演着关键角色,其水平可能直接影响 VEPs 的抗菌效能。
在二级结构研究中,通过圆二色光谱(CD)分析,团队观察到 VEPs 的结构变化具有显著环境依赖性。在水溶液中,VEPs 主要以无规则结构存在;而在模拟细菌膜环境(如 SDS 胶束体系)或螺旋诱导介质(如三氟乙醇/水混合溶液)中,它们会发生明显的构象转变,从无序结构折叠为 α-螺旋。这种结构特征与典型抗菌肽的行为高度一致,暗示 VEPs 可能特别适应与生物膜相关的功能,这也为其抗菌机制提供了重要线索。
作用机制研究方面,团队通过荧光测定法评估了 VEPs 对细菌外膜和细胞质膜的影响。结果如下图所示,23 种 VEPs 能有效渗透细菌外膜,其中 Arachnoserver-18、ConoServer-6 和 ConoServer-7 的渗透活性尤为突出。在细胞质膜去极化实验中,大多数测试 VEPs 的去极化效果优于对照组,但相较于部分已知肽家族则稍弱。综合来看,VEPs 的抗菌作用主要通过诱导细胞质膜去极化实现,这一机制与多种已知抗菌肽及加密肽的作用模式相符,进一步支持了其作为新型抗菌剂的合理性。
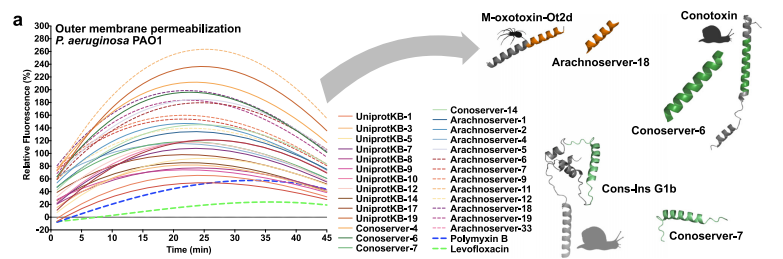
毒液中抗菌肽的作用机制研究
学界与业界协同探索,助力新型低耐药性抗菌药物研发
为开发不易引发耐药性的新型抗菌药物,从基础研究到产业转化,学术界与企业界的协同探索正不断解锁毒液抗菌肽的应用潜力。
在学术界,各国研究团队围绕毒液抗菌肽的挖掘、设计与机制解析展开深入攻关。新加坡南洋理工大学慕宇光团队针对传统模型在蛋白相互作用预测中受限于数据量和特征维度的问题,构建了目前最大的基于结构的蛋白质相互作用数据集,并开发出 ProAffinity-GNN 深度学习框架。该模型创新性融合蛋白质语言模型与图神经网络,将序列信息与空间结构特征深度耦合,不仅在蛋白-蛋白亲和力预测精度上超越现有方法,其强大的泛化能力更为毒液肽与细菌靶点相互作用的高效预测提供了技术范式。与此同时,对作用机制的解析为抗菌肽的优化设计提供了理论支撑。
山东大学齐鲁医学院团队借助人工智能领域的扩散模型,模拟自然界肽段进化的「随机扰动-定向筛选」过程,成功生成 40 种全新多肽序列,其中 25 种展现出明确的抗菌或抗真菌活性。
在企业界,以色列 Bountica 公司则聚焦食品防腐领域的痛点,将毒液来源的抗真菌肽通过微生物发酵技术规模化生产。其开发的 VenomShield 系列产品,仅需百万分之一的添加量就能抑制面包、果汁中的霉菌与酵母菌生长,保质期延长至传统防腐剂的 2 倍以上。
中国上海高科生物工程有限公司与复旦大学合作,共建了全球 3 个相对完整的抗菌蛋白数据库。这些数据库不仅为研发新型生物抗菌酶、抗菌肽提供了高效工具,更通过系统化的数据挖掘加速了抗耐药微生物新物质的研究。
由此可见,毒液抗菌肽的研究与转化正展现出跨越基础科学与产业应用的强大生命力。随着深度学习模型对序列-功能关系的解析愈发精准,以及合成生物学技术对肽段稳定性与表达效率的持续优化,这类源自自然界「生存智慧」的分子,有望在未来成为对抗抗生素耐药性的关键力量,为全球公共卫生安全提供新的保障。
参考文章:
1、https://mp.weixin.qq.com/s/lwYWWIe9-Az22jlhiZ747A
2、https://mp.weixin.qq.com/s/sGHRf-ebaSRiqGiUxMMueA
3、https://mp.weixin.qq.com/s/0mJltGuaKTUwYDGK4wD9UA
4、https://mp.weixin.qq.com/s/R6Y_-38saZwqSSow0LNhzw