——背景——
在一个40个残基的序列中,氨基酸的排列比地球上的原子还要多,加上蛋白质复杂的二级、三级结构,这一巨大的化学搜索空间阻碍了人类设计功能性蛋白,而多肽,尤其是功能性的线性多肽,在设计上具有复杂度低的优势。MIT的L.Pentelute教授实验室和Cambridge的Rafael Gómez-Bombarelli教授实验室合作发表在Nature Chemistry近期名为Deeplearning to design nuclear-targeting abiotic miniproteins的工作,展示了机器学习如何使非生物核靶向线性多肽的从头设计能够将反义寡聚体输送到细胞核,将高通量实验与定向进化启发的深度学习方法相结合,其中自然和非自然残基的分子结构表示为拓扑指纹。该模型能够预测训练数据集之外的活动,同时破译和可视化序列-活动预测。被称为“Mach”的预测的微蛋白的平均质量达到10 kDa,比细胞中任何先前已知的变体更有效,并且还可以将蛋白质输送到细胞质中。Mach微蛋白无毒,能在小鼠体内有效地传递反义物质。这些结果表明,深度学习可以破译设计原理,产生高度活跃的生物分子,而这些分子不太可能被经验方法发现。
——准备工作——
Mini protein库的合成
作者首先通过化学手段将PMO与57种多肽进行组合偶联构筑了600个迷你蛋白,每个偶联物的多肽包含有三个部分:细胞膜靶向肽、非天然肽以及环肽。他们通过高通量实验测定了定量转染效果,并以此作为数据训练预测模型。
线性多肽可合成性的预测
作者结合此前发表的工作中的多肽可合成性预测模型也被运用到本工作中,该模型设计思路与本工作模型相似,都是主要利用分子指纹信息进行性质预测。作者在肽表达和合成参数的基础上,训练了一个深度神经网络模型来预测肽合成中归一化的第一偶联的UV - vis出峰的积分、高度和宽度。
 图1. 可合成性预测示意图,通过合成与计算结果进行循环优化:a.合成与性质测定;b.机器学习模型;c.序列优化
图1. 可合成性预测示意图,通过合成与计算结果进行循环优化:a.合成与性质测定;b.机器学习模型;c.序列优化
——方法——
算法亮点
本工作的亮点是通过RDkit提供的分子描述符对多肽结构进行描述,而此前主要的描述方法是一种在残基水平上的one-hot法,旧方法对多肽性质的描述非常有限,只能区分多肽的种类信息。为了更进一步完整化、准确化输入模型的多肽信息,作者采取了一种分子描述符的氨基酸表征方式:每个残基由一个向量构成,该向量中包含了此残基的结构信息,并且在此基础上,选出跟氨基酸结构信息相关的191个描述符组成向量作为输入的结构信息,向量的顺序组合则构成了整个多肽的“指纹”。在对不同的模型进行尝试后,他们发现基于卷积神经网络的模型具有良好的拟合以及预测效果。
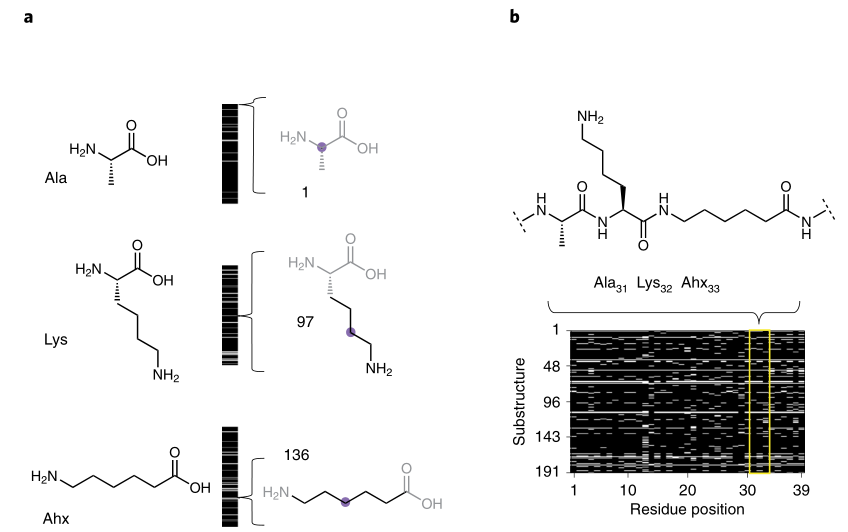
图2. a.分子指纹对应的结构信息示意图;b.多肽分子指纹矩阵示意图
算法模型构架:generator–predictor–optimizer loop
算法由三部分组成:generator、predictor、optimizer,对多肽序列进行循环优化。
首先由一个RNN模型结合序列的先验知识生成具有活性潜力的多肽序列,然后将多肽的分子指纹矩阵输入到CNN-predictor里进行活性预测,通过一个遗传算法对序列进行突变然后通过优化打分最终得到高活性序列。
打分项如下:
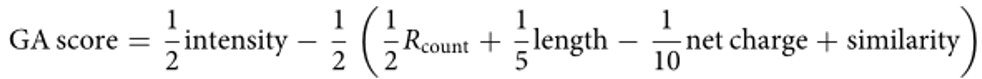
Intensity为CNN-predictor的活性预测值,Rcount为精氨酸的个数,作为罚分项,多肽的长度作为罚分项,带电量作为奖励项,序列相似性作为罚分项。
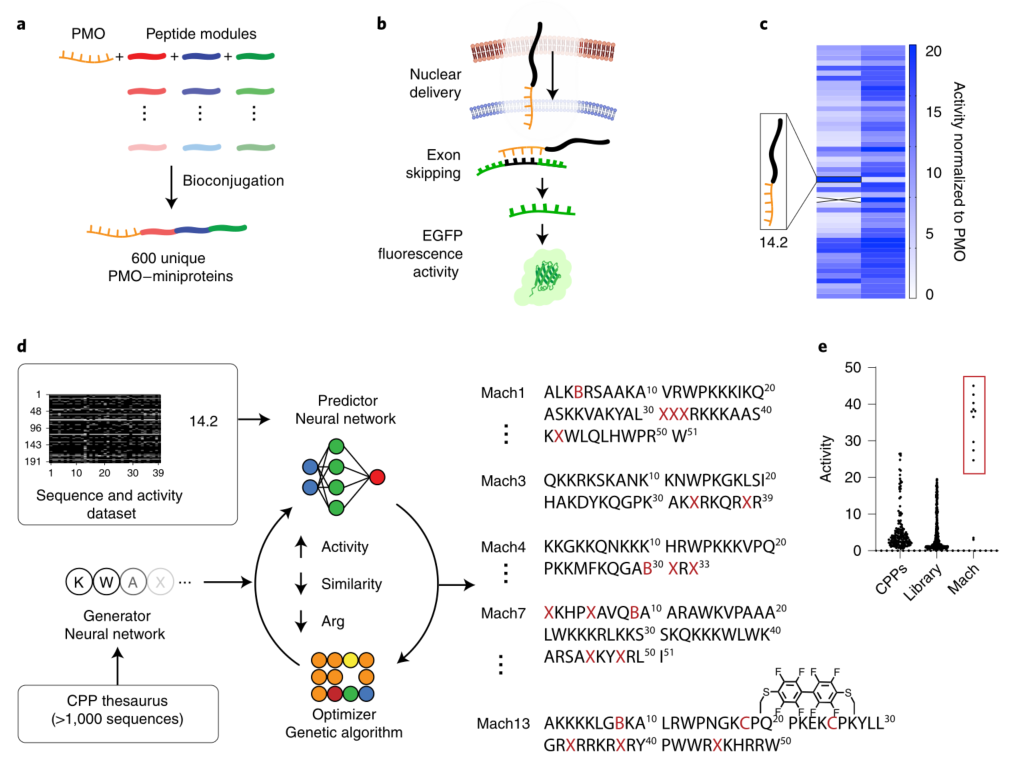
图3. d.三元模型循环优化序列示意图;e.Mach序列活性与优化前多肽数据库活性的对比
——结果与总结——
Mach数据集预测结果对比:
作者得到的优化后的序列组成了Mach数据集,通过实验结果可以很清晰地观察到Mach数据集中的多肽精氨酸数目下降(即细胞毒性降低),活性提高,达到了预期的效果。
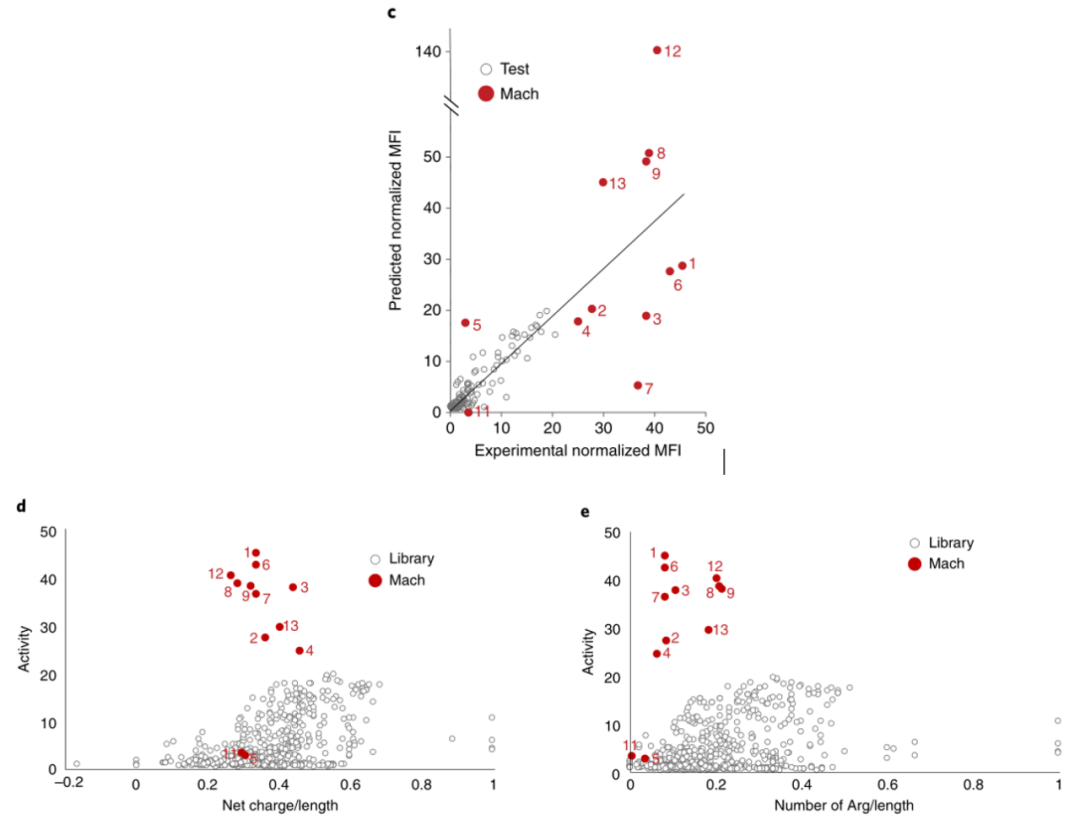
图4. c.优化前后MFI对比图;d.优化前后活性与平均电荷数对比图;e.优化前后活性与平均精氨酸个数对比图
对分子描述符部分的分析:
作者从网络中取出分子描述符各个区段的参数,用数值绝对值大小代表某个描述符的激活程度,并发现:
1. 在某些结构位点所代表的描述符的激活程度要明显高于其他位点;
2. 不同长度的多肽主要的激活位点都位于链的碳端;
3. 不同长度的多肽激活程度较高的描述符位置基本一致。
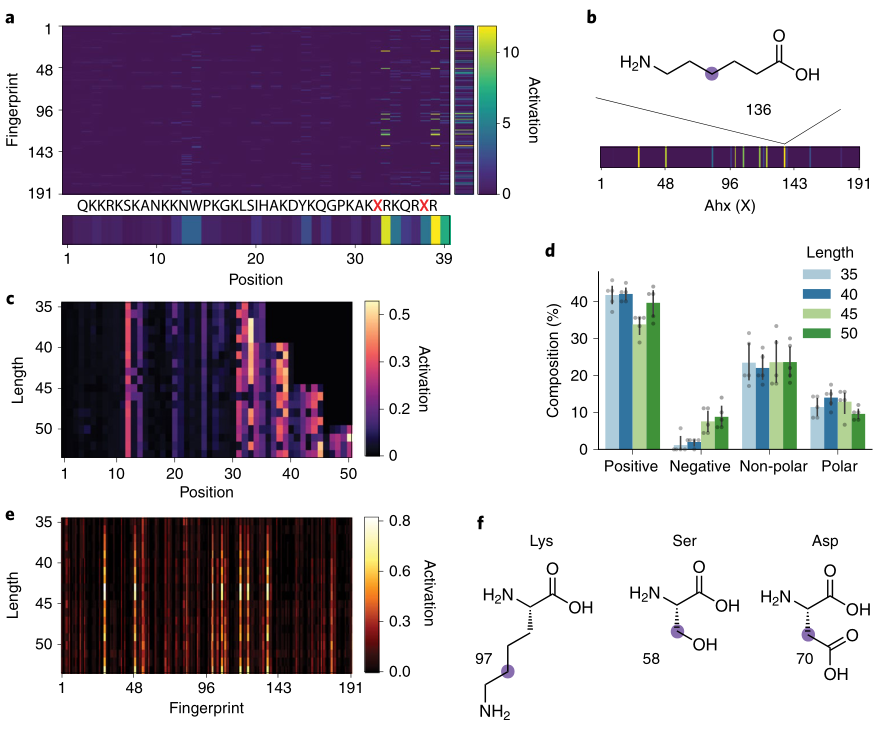
图5. a-b.氨基酸活性结构位点与分子描述符向量的对照图;c.不同长度多肽活性氨基酸位置的对比示意图;d.氨基酸长度与带电量的对比示意图;e-f氨基酸长度与描述符激活程度的示意图
因此可以认为,分子描述符能够较为细致地反应氨基酸内部的结构信息,从而能使模型学到更多的与活性和毒性相对应先验知识,从而更加准确的预测结构活性并进行序列优化,得到活性更高的序列。
作者还通过将遗传算法优化器的进化方向指向相反的方向,即最小化MFI,但保持其他约束不变,生成了一个非活性序列(Mach11),其氨基酸组成与活性预测相似。合成后,Mach11偶联物表现出较低的实验活性(如图4.c中所示),这证明了该模型在预测独特序列活性方面的鲁棒性。
参考文献:
[1] Rafael Gómez-Bombarelli and Bradley L. Pentelute. "Deep learning to design nuclear-targeting abiotic miniproteins" Nature Chemistry. 13 (2021): 992-1000. https://doi.org/10.1038/s41557-021-00766-3
[2] Rafael Gómez-Bombarelli and BradleyL. Pentelute. "Deep Learning for Prediction and Optimization of Fast-Flow Peptide Synthesis" ACS Central Science. 6,(2020): 2277−2286. https://dx.doi.org/10.1021/acscentsci.0c00979
点击左下角的"阅读原文"即可查看原文章。
作者:王凡灏
审稿:刘佳乐
编辑:黄志贤
GoDesign
ID:Molecular_Design_Lab
( 扫描下方二维码可以订阅哦!)

本文为GoDesign原创编译,如需转载,请在公众号后台留言。