胶质瘤是最常见的脑部恶性肿瘤,患者的平均生存时间为18月。对患者的生存时间基于分子标志预测,可以找出具有治疗潜力的患者。通过汇总多个模型的预测结果,可以构建更加具有鲁棒性,范化能力更强的模型,这在机器学习中被称为集成学习(ensemble learning),9月22日来自青岛大学研究者的论文“The genetic algorithm-aided three-stage ensemble learning method identified a robust survival risk score in patients with glioma”,影响因子13.99。介绍了结合遗传算法,集成学习的生存时间预测模型。该文将详细介绍其算法原理。

算法的训练数据,来自11个公开数据库的集合,包含患者的RNA表达谱和生存时间。其中来自TCGA和CGGA693这两个样本量较大的数据集被用作训练数据集,而全部数据集(总计1191个样本)被当作测试数据集。
模型训练的第一阶段,使用TCGA数据集通过基因配对(gene-pairing)算法进行特征提取,以避免批次效应;
模型训练的第二阶段,在CGGA693数据集上,通过10叠交叉验证,在47个基础预测模型(例如随机森林,决策树,支持向量机)上通过对参数的网格搜索,找出最优的模型参数组合;
模型训练的第三阶段,在TCGA数据集上,通过遗传算法,从第二阶段训练得出的模型中,找出最优的三个模型,作为最终进入集成学习框架的三个基础模型。之后在所有11个来源的数据集上,对训练的模型进行评价。
具体来看每一阶段的训练过程,根据医学信息,选取217个关键基因,之后通过Cox回归,找出203个关键基因,用于进一步的特征工程。之后将剩下了的203个基因两两配对,形成203*202个基因对,根据下图的公式,进行配对。
之后对于每个样本,判断配对所处的频率所在的相对位置,选取位于20-80%的基因对(去除那些在该批次中的表达量大多是存在相关性,即A在大多数时大于或小于B的基因对),以避免批次差异。经此过程选出的6300个基因对,通过与生存时间做Lasso回归,选出最终用做特征的40个基因对。
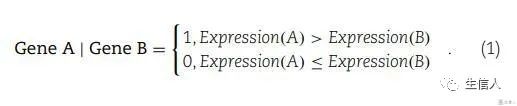
a是各个基因对PCA降维后的结果,b与c是各个数据集上,基因组合在去批次效应之前的降维结果,d是去除异常值之后,对剩余的基因对表达量的降维结果,可见去批次效应之前,降维结果显示不同批次聚在一起,去除后则相对均一。之后的原始的预测模型训练,在新数据集CGGA693上利用上一阶段得到的特征,在47个模型上,通过10折交叉验证和网格搜索,依次寻找每个模型下的预测准确性最高的参数组合。
在第三阶段,依旧使用同一批数据,使用遗传算法,找出采取那些模型的组合,能够以更高的效能进行生存时间预测。以选取那些模型组合作为基因型,以集成学习后预测误差为表型。
最初选取随机组合的预测模型作为基因型,通过不断的模拟进化过程(让模型组合经过点突变和重组),再通过其预测准确性作为适应度,选择下一代模型组合的“物种池”,以此找出选取那些模型组合进行集成学习时,预测的准确性最高。最后在全部数据集组成的样本集合上,评价模型的准确性。
预测过程中,考察了诊断后从第一年到第五年的生存率(使用ROC评价),并通过泛癌症分析,在五种不同的癌症下,使用训练的模型进行预测。
评价时先按照生存率预测,将患者分为两组,计算不同风险组的Kaplan–Meier s生存时间曲线,在11个数据集中的每一个,以及组合后测试集上,可以看到两组的生存率有显著差异。
之后判断针对个体患者的1年,3年,5年的生存率预测,通过ROC曲线评价预测准确性,结果见图4。在所有样本的数据集上,第一年生存率ROC为0.705, 第三年为0.825,第五年为0.839图4 不同数据集上生存概率预测的ROC曲线
为了说明模型预测的鲁棒性,通过将样本和标签置换(npermutation test),重新计算ROC,发现新预测的结果和之前的结果是相近的,这说明模型是学到了数据间的特征,而非仅仅记住数据标签。

该论文的亮点,不仅在于提供了一个简单易用的,对胶质瘤及其它癌症患者基于转录组数据预测预后效果(生存率)的在线工具,更在于提出了一种可以借鉴的生信分析思路,即先通过搜集不同来源的公开数据集,之后通过判断组间数据的相关性,选出不存在组间依赖的特征集合,以此去除批次效应。之后批量训练各类预测模型,再通过诸如遗传算法的组合优化算法,找出使用那些分类器集合后的预测效果最佳。通过类似的套路,可以对各种癌症,使用多组学,多来源进行类似的模型构建,预期也会产生比单一预测模型,或基于所有基础预测模型的集成学习更优的结果。