DNA甲基化(DNA methylation)为DNA化学修饰的一种形式,能够在不改变DNA序列的前提下,改变遗传表现。作为最早发现的基因表观修饰方式之一,DNA甲基化在维持正常细胞功能、染色体结构、X染色体失活、基因印记、胚胎发育过程、衰老以及疾病的发生中起着重要的作用。近些年,结合数据驱动的人工智能算法,利用DNA序列建模实现DNA甲基化预测,为加速甲基化的发现提供一种重要手段。然而,目前大部分的方法存在的问题在于模型缺乏可解释性,无法从序列层面建立起甲基化功能的对应关系。

近日,魏乐义教授团队在生物信息领域顶级期刊Genome Biology(影响因子: 17.906)上发表了题为”iDNA-ABF: multi-scale deep biological language learning model for the interpretable prediction of DNA methylations”的研究成果。该论文提出了一种基于多尺度深度学习的生物序列与功能语义模型iDNA-ABF实现对DNA甲基化的可解释性预测。在多物种数据集及人类细胞系数据集中,iDNA-ABF均展现出较好的性能。通过引入对抗训练策略,增强了模型的鲁棒性从而让模型在不同大小的训练集上都获得较优异的性能,也可以预防出现大模型的过拟合问题。另一方面,通过引入attention机制,iDNA-ABF获得了良好的可解释性,能够有效从序列层面建立起甲基化功能的关系,从而为生物学家的研究和发现提供启发。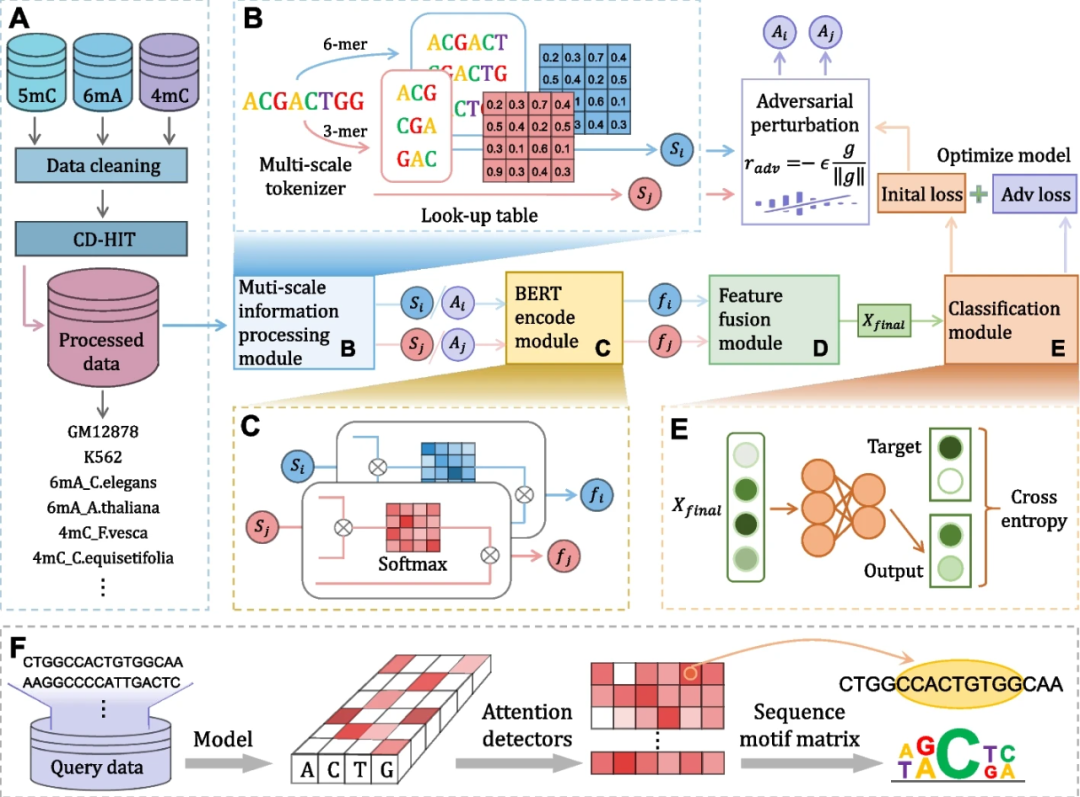
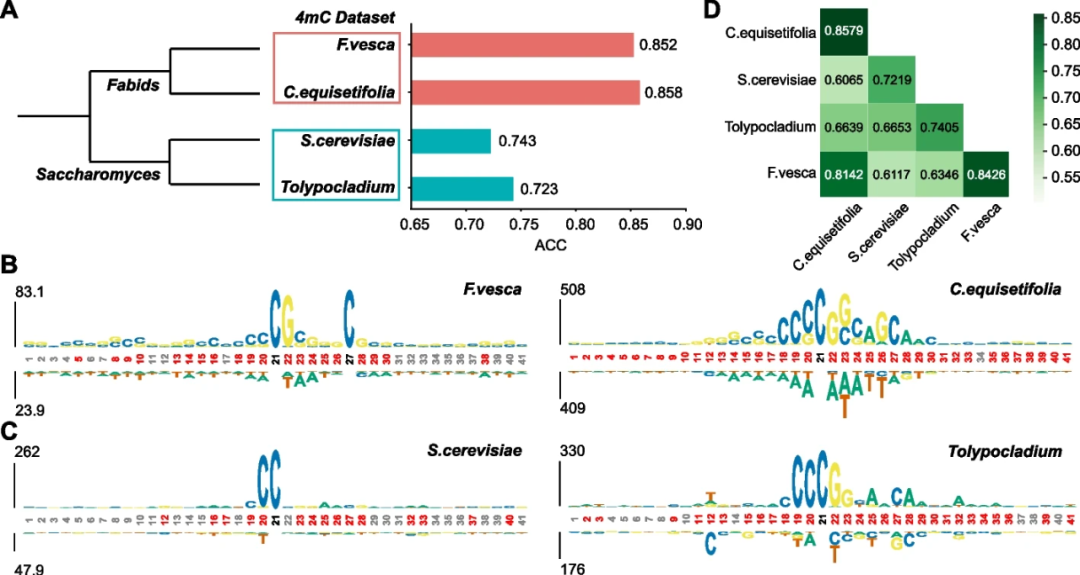
在多物种数据集中,iDNA-ABF表现出强大的性能提取和表示能力,在测试的17个数据集中,有15个数据集的指标都超过了现有方法。有趣的是,研究人员对于同一种甲基化的不同物种数据集做了进化分析,研究人员发现,在进化层面上越具有亲缘关系的物种,预测模型表现出来的性能越接近(见下图),说明在序列水平上甲基化模式跟物种亲缘关系可能呈现正相关关系;通过对于数据集进行motif分析,进一步证实这一点。
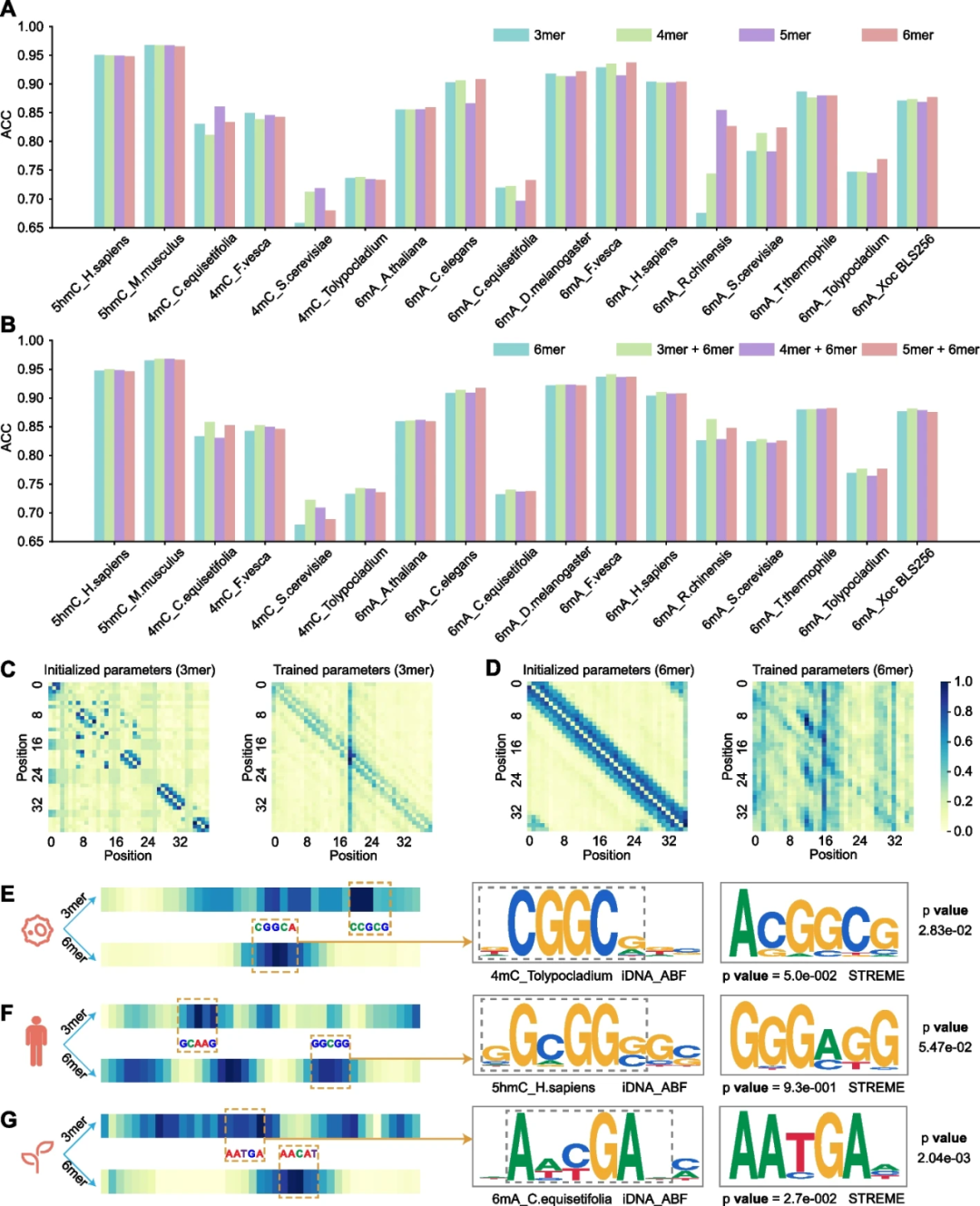
为了充分展示模型的信息学习机制,在这篇工作中研究人员利用iDNA-ABF的attention机制对模型学习的内容进行了深度分析。首先,研究人员研究不同尺度的分词策略在信息捕捉方面的影响。通过对不同尺度的可视化(下图CD),可以看出训练模型后的3mer模块更加关注局部信息,6mer模块更加关注全局信息。因此,多尺度模型在一定程度上能够相互补充信息,从而达到提取更好的特征表示。接着,为了更好可视化模型学习的内容,研究人员将不同权重的累和归结为提取到的motif,再将其与传统的STREME算法提取得到的motif进行对比(见下图EFG),可以明显的看出其中的相似性。通过可解释分析,更加说明了iDNA-ABF学习到了有效的知识表达,能够充分从数据中提取有效的关键序列信息。
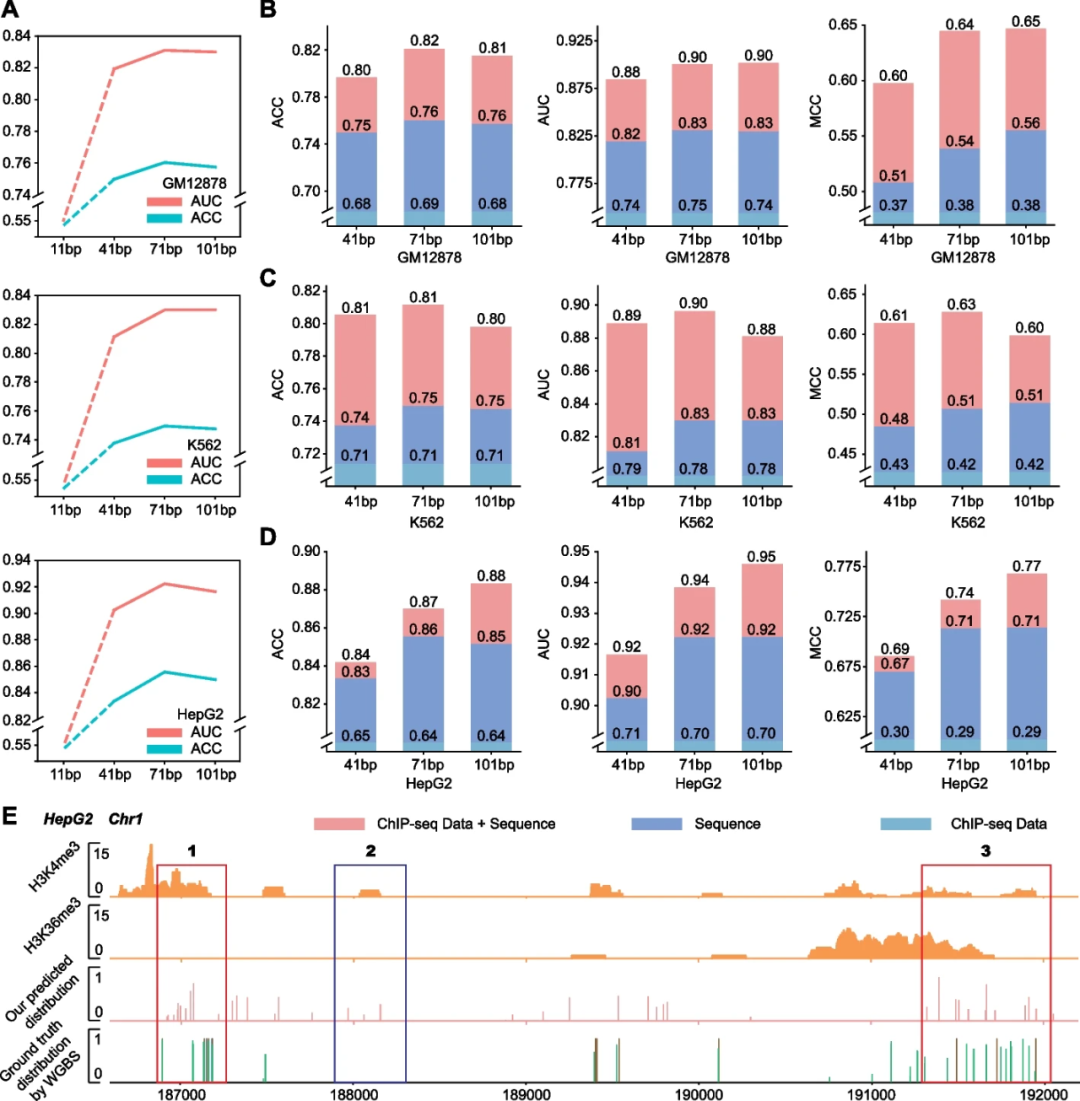
除了研究不同物种的甲基化模式,研究人员还进一步研究了在同一个基因组不同的细胞系间,DNA序列与甲基化的关系。由于5mC是一种非常重要的甲基化类型,研究人员重新构造了新的人类细胞系数据集,并围绕该数据集进行了详细的讨论。人类细胞系的数据集主要选取了三种cell line分别进行了研究。首先,通过控制输入序列的长短,来研究序列长度输入对模型的影响,从结果看出,随着序列输入长度的不断增加,模型的表现更加优异直至收敛,说明输入充分的信息能够让模型学习到更加准确的预测结果。其次,考虑到很多下一代测序技术对甲基化的影响,研究人员还加入了ChIP-seq数据对模型进行数据增强。结果表明,融合ChIP-seq的数据能够有效提高模型性能;另一方面,表明了序列信息能够起到正面增益的效果。最后,作者在真实的cell line序列数据上(HepG2 Chr1)进行了预测,得到的效果比较而言也十分优异,预测得到的区域和ground truth也能基本吻合。特别的是,研究人员发现iDNA-ABF还能挖掘出一些符合ChIP-seq信号,但不是ground truth的特异性序列区域,这些可以作为潜在的甲基化区域,为生物实验的验证提供更好的切入点。
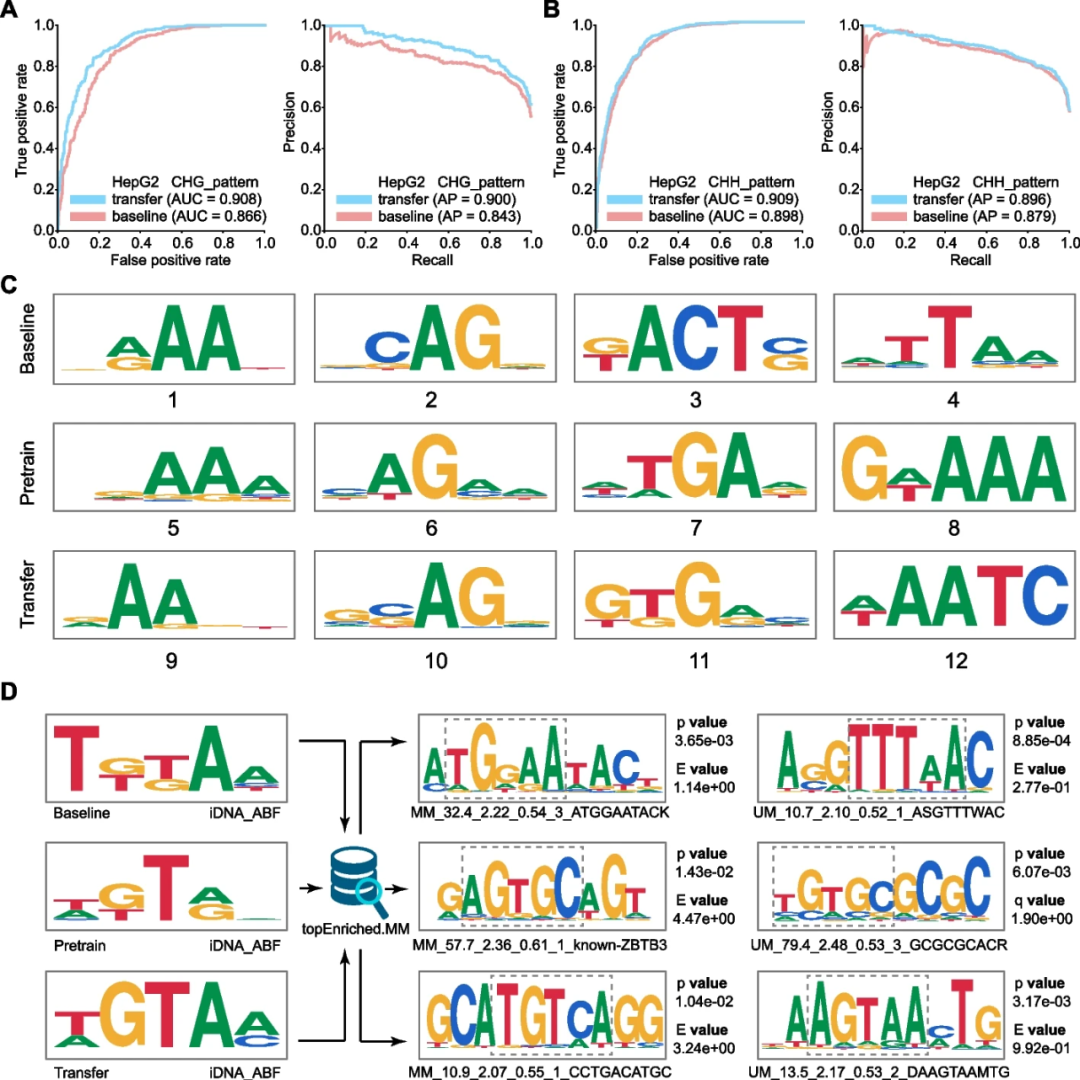
最后,研究人员进一步对模型在不同甲基化序列模式(如:CpG较为常见、CHH较为罕见)的迁移学习能力做了深入研究。迁移结果表明iDNA-ABF具有良好的迁移学习能力,能够有效提升罕见序列模式的预测性能,从而能有效缓解因为训练样本(罕见甲基化模式)不足带来学习不充分的问题。另一方面,
通过深入分析性能提升的原因,研究人员展示通过迁移学习,模型能捕捉不同甲基化模式的特异性(如下图)。重要的是,模型学习到的motif是具有显著生物功能性的,表明模型能充分挖掘到甲基化的功能语义。
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-022-02780-1
https://github.com/FakeEnd/iDNA_ABF