显微镜是表征细胞和亚细胞结构和动力学的有力工具。虽然显微镜技术的进步为在细菌的单细胞水平上定量和精确地测量形态和分子现象提供了巨大的希望,但对于显微镜图像的定量分析仍然是一个挑战,因为许多细菌只能在可见光波长的范围内被显现。两年前,科学家们开发出一种基于深度学习的分割方法,名为Cellpose,它可以从各种图像类型中精确分割细胞,并且不需要模型重新训练或进行参数调整。然而,该方法仍受到独立于细胞形态或光学特性方法的限制。近日,发表在《Nature Methods》上的一项新研究中,来自华盛顿大学的研究团队开发了深度神经网络图像分割算法——Omnipose。经过大型细菌图像数据库训练的Omnipose在描述和量化混合微生物培养物中的各种细菌方面表现良好,并消除了使用其前身工具Cellpose时可能出现的错误。
众所周知,细菌除了身材微小以外,还具有多种形态。虽然普遍研究的细菌基本都近似于棒状或球状,但还有的细菌是其他形态,如螺旋状。此外,微流体装置使研究人员可以捕捉细菌对抗生素等各种治疗的反应,但在这一过程中通常能导致细菌产生不规则的形态。这些形态特性会使现有深度学习工具很难从显微图像中判断出存在哪些细菌。在这项新研究中,该团队开发了深度神经网络图像分割算法Omnipose。独特的网络输出(例如距离场的梯度)允许Omnipose准确分割现有算法(包括其前身 Cellpose)产生错误的细胞。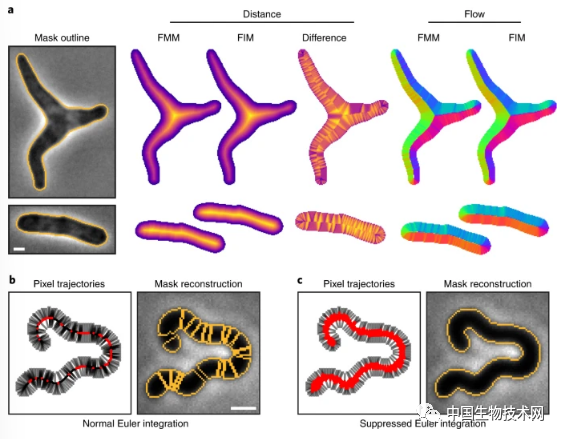
研究表明,Omnipose在混合细菌培养物、抗生素处理的细胞,以及细长或分支形态的细胞上实现了前所未有的分割性能。此外,Omnipose在克服由于不同细菌的光学特性差异而造成的识别问题方面做得更出色。 该算法增强的细胞分割性能促进了对微生物系统的独特见解。Omnipose可以准确识别中毒的大肠杆菌细胞
不过,在将拥挤的微生物群落3D样本以2D方式再现时,Omnipose还存在一些限制。例如,图像中的元素重叠会产生错觉效果。此外,当细菌大小低于某个阈值时,Omnipose很难检测出来。
尽管如此,在分析快速生长的拟南芥根原始数据集中的细胞时,Omnipose可以显示出一些在3D样本中优于现有方法的优势。虽然还存在一些缺点,但是研究人员认为,Omnipose可以是一种帮助解答细菌细胞生物学中各种问题的解决方案。为了验证Omnipose是否也能成为其他依赖显微镜的生物科学甚至非生命科学领域的多功能工具,该团队尝试用Omnipose对秀丽隐杆线虫(C.elegans)的显微镜图像进行分析。C.elegans是遗传学、神经科学、发育和微生物行为研究中一种重要的模式生物。C.elegans以细菌为食,如同一些细菌一样,C. elegans也是线状,可以自由扭曲成各种形态。测试显示,无论是伸展、收缩还是其他各种运动,Omnipose都能识别出C.elegans。研究人员表示,这种能力在延时追踪线虫运动期间的神经研究中可能会非常有用。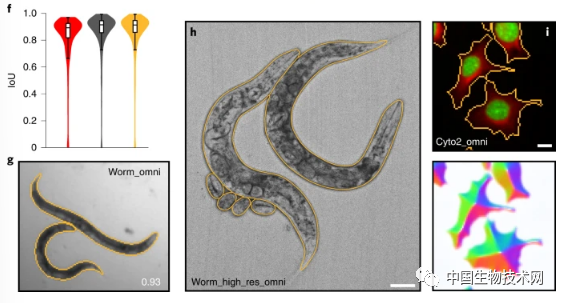
目前,该团队已经公开了Omnipose的源代码、训练数据和模型,还提供了关于安装和使用Omnipose的完整文档。他们预计,Omnipose在不同的细胞形态和模式下的优秀表现,可能会从显微镜图像中解锁以前无法获得的信息。Omnipose甚至可能会改变生物图像分析的游戏规则。论文链接:
https://www.nature.com/articles/s41592-022-01639-4
Omnipose完整文档:
https://omnipose.readthedocs.io/
来源:中国生物技术网 2022-10-30