通过组蛋白修饰对转录控制的定量表征受到许多计算研究的挑战,但其中大多数只关注启动子周围的狭窄和线性基因组区域,留下了改进的空间。
韩国首尔大学的研究人员提出了 Chromoformer,这是一种基于 Transformer 的三维染色质构象感知深度学习架构,它在基因调控中组蛋白代码的定量破译方面实现了最先进的性能。Chromoformer 架构的核心本质在于注意力操作的三个变体,每个变体都专门模拟转录调控的个体层次,涉及从核心启动子到通过三维染色质相互作用与启动子接触的远端元件。
对 Chromoformer 的深入解释表明,它自适应地利用了与转录起始和延伸相关的组蛋白修饰之间的长程依赖性。研究表明,Chromoformer 可以捕获转录工厂和 Polycomb 基团的定量动力学。总之,这项研究突出了基于注意力的表观基因组中复杂相互作用的深度建模的巨大优势。
该研究以「Learning the histone codes with large genomic windows and three-dimensional chromatin interactions using transformer」为题,于 2022 年 11 月 5 日发布在《Nature Communications》。

基因表达的控制由不同组的调节因子进行,包括转录因子、共激活因子、辅助抑制因子以及基因组序列元件。然而,这些因素相互作用背后的基本前提是在相关基因组区域中组蛋白尾部或组蛋白修饰(HMs)的共价修饰的适当配置,因为它们在染色质可及性的调节中起关键作用。因此,可以设想一定数量的 HM 及其组合编码了附近基因组区域的调节潜力。
这个概念被称为「组蛋白密码假说」。已经有许多计算和定量方法来破解由 HM 编码的基因表达的调控代码。它们中的大多数是预测模型,利用转录起始位点(TSS)周围启动子的 HMs 水平来预测相应基因的表达水平。值得注意的是,最近的研究表明,在这项任务中,深度学习模型与传统机器学习模型相比具有卓越的性能。
两个挑战
迄今为止,深度学习已经在计算生物学的各个领域取得了显著的突破,从表征结合 DNA 和 RNA 结合蛋白的特异性,到长期存在的基于氨基酸序列的蛋白质结构预测问题。如果没有新的模型架构的发明以及它们对复杂生物学问题的巧妙应用,就无法在生物学中取得深度学习的这些成功。从这个意义上说,组蛋白代码的高度复杂性确实使其成为深度学习的一个很好的目标,如现有方法所示,但它们仍然存在两个主要的限制,推动了新方法的开发。
首先,他们只能在 TSS 周围使用狭窄的基因组窗口。这是因为这些模型所基于的深度学习架构,例如卷积神经网络(CNN)和循环神经网络(RNN),在对长序列内的依赖关系进行建模时效果不佳。CNN 高度专业于学习数据的局部模式,但对它们来说学习模式之间的远距离依赖关系具有挑战性。
尽管 RNN 架构是为对序列数据进行建模而开发的,但由于嵌入在单个位置的信息逐渐被稀释并被污染,而模型计算沿着两个遥远位置之间的位置传播,RNN 架构也难以清楚地捕获远程依赖关系。事实上,诸如门控循环单元或长短期记忆 (LSTM) 之类的 RNN 单元的高级形式部分地改善了这个问题,但是由于循环而对长序列进行建模的内在低效率仍然存在。
其次,大多数深度学习模型不考虑由三维(3D)染色质折叠介导的远端顺式调节,尽管众所周知,核心启动子和远端顺式调节元件之间的物理相互作用会严重调节基因表达。换言之,组蛋白代码所传达的调控信息不仅可以在本地传播,还可以通过 3D 染色质相互作用在遥远的基因组位点之间跳跃。
幸运的是,Hi-C 等高通量测量技术的最新进展成功地提供了千碱基规模的 3D 染色质相互作用的高分辨率视图,并为研究人员提供了前所未有的机会,来利用这些有价值的信息来模拟基因调控的综合观点。很少有新兴研究明确考虑 3D 染色质相互作用来预测基因表达。一个这样的例子是 GC-MERGE,这是一种图神经网络 (GNN),用于在相互作用的基因组区域之间传播信息以预测基因的表达水平。
虽然它是一个概念验证模型,不能应用于没有任何染色质相互作用的基因,并且只能执行 10 kbp 基因组 bin 级别的预测,但不能在基因级别进行预测,它仍然强调了对远端基因组区域以及启动子的表观基因组环境进行建模的前景。
Transformer 也许是药方
与此同时,最初为自然语言处理而开发的名为 Transformer 的深度学习模型架构,在理解 DNA 序列、氨基酸序列甚至它们的比对的潜在语法方面表现出巨大的潜力。在这项研究中,研究人员注意到 Transformer 架构的两个主要功能非常适合解决上述两个挑战。
首先,Transformer 可以精确地建模序列数据中的长期依赖关系。这是通过向输入序列添加位置编码来优雅地完成的。这些包含位置信息的输入特征被独立处理,并被输入到随后的自注意力模块中,该模块计算输入特征之间的所有成对依赖关系。因此,可以在不受位于对之间的特征干扰的情况下捕获远程依赖关系。
其次,Transformer 架构也可以应用于建模无序的实体集以及它们之间的交互。值得注意的是,对于大多数深度学习架构来说,这并不简单,因为包含它们的操作取决于输入位置。另一方面,构成变换器的操作基本上是置换不变的。
输入特征之间的交互仅在 self-attention 操作中考虑,所有其他操作都以位置方式完成,因此它们可以应用于模型的无序特征集。总之,Transformer 架构的这两个优势使其成为组蛋白代码定量建模的有希望的选择,因为它允许研究人员同时在多个远端调控区域利用 TSS 附近更宽的基因组窗口和组蛋白代码。
基于Transformer 的深度学习架构 Chromoformer

图示:Chromoformer 模型架构。(来源:论文)
在这里,首尔大学的研究人员提出了一种名为 Chromoformer 的基于 Transformer 的深度学习架构,以模拟组蛋白代码在基因表达调控中的定量作用。Chromoformer 通过对涉及核心启动子和 pCRE 的三级顺式调节层次进行建模,极大地提高了基因表达预测的性能。
通过对 self-attention 权重、潜在嵌入动力学和几个特征消融研究的分析,研究人员对 Chromoformer 模型的行为提供了深入的生物学解释。

图示:促成 Chromoformer 卓越性能的因素。(来源:论文)
由于 Transformer 能够理解序列中的远距离依赖性,Chromoformer 可以成功地学习关注基因体内的特定区域,在该区域中,与基因表达相关的 HM 在高表达和低表达基因之间最为独特。有趣的是,对基因体的关注程度取决于 TSS 的表观遗传背景,这意味着 Chromoformer 模型捕获了放置在 TSS 和基因体的 HM 的远距离依赖性。
另一方面,通过使用 Transformer 对一组无序特征中的成对关系进行建模,Chromoformer 可以了解由组蛋白代码介导的信息如何通过 3D 染色质折叠从 pCRE 传播到核心启动子以调节基因表达。对模型学习的组蛋白密码的潜在表示的分析强调,持家基因和细胞类型特异性基因的表达通过与增强子的相互作用得到加强,而发育基因的表达主要通过与 PRC2 结合的消音器的相互作用受到抑制。
该团队使用 3D 染色质相互作用的预编译知识来指导 Chromoformer 学习。那些通过实验测量的交互频率被用来确定将参与模型训练的 pCRE 的优先级,这些 pCRE 被显式地注入到自我注意得分矩阵中。
然而,仅从基因组序列信息中推断 pCRE 和核心启动子之间的相互作用频率似乎也是可能的。这是因为顺式调控相互作用的特异性很大程度上取决于 DNA 结合蛋白对 DNA 序列基序的识别,包括转录因子或 CCCTC 结合因子 (CTCF),它们作为分隔 3D 基因组构象的绝缘体。因此,嵌入基因组中的那些结合基序可以作为隐藏的词汇表,允许仅基于 DNA 序列推断所需的染色质构象。

图示:Chromoformer 学习的顺式调节特征。(来源:论文)
同时,来自最近名为 Enformer 的模型的结果强烈支持,当使用更广泛的序列信息时,pCRE 的这种从头排序更有效,从而表明通过使用 Transformer 架构整合基因组和表观基因组特征,实现基因表达调控的完全数据驱动建模的令人兴奋的可能性。该团队将这种基于 Transformer 的多组学集成作为进一步的工作。
从活跃的 TSS 跳转到基因体的嵌入 Transformer 所学到的注意力表明,在预测稳态基因表达水平时,放置在基因体上的 HM 确实是有用的,即使不是最关键的信息。从这个结果中,研究人员考虑使用分布在单个基因中的整个组蛋白代码景观,可以进一步提高稳态 mRNA 水平的预测准确性的可能性。
此外,由于研究所用的 H3K36me3 的外显子比内含子丰富得多,因此利用全长基因注释将是模型训练的另一个有效指导。由于基因长度和外显子-内含子分布显示出很大的可变性,研究人员需要对这种生物学先验知识进行一些巧妙的表示。
同样,Transformer 架构将是最强大的选择之一,因为可以灵活地应用掩码来处理可变长度输入,还可以扩展位置编码以形成复合编码,同时包含基因组位置和基因结构注释的信息。
为 Chromoformer 模型提出的训练方案具有高度可扩展性。例如,该团队展示了 Chromoformer 模型可以针对来自人类以外物种的细胞类型进行训练,即小鼠胚胎干细胞,使用相关的组蛋白 ChIP-seq 和 Hi-C 配置文件,通过跨物种预测性能证明了两个物种之间组蛋白代码语法之间的总体相似性。
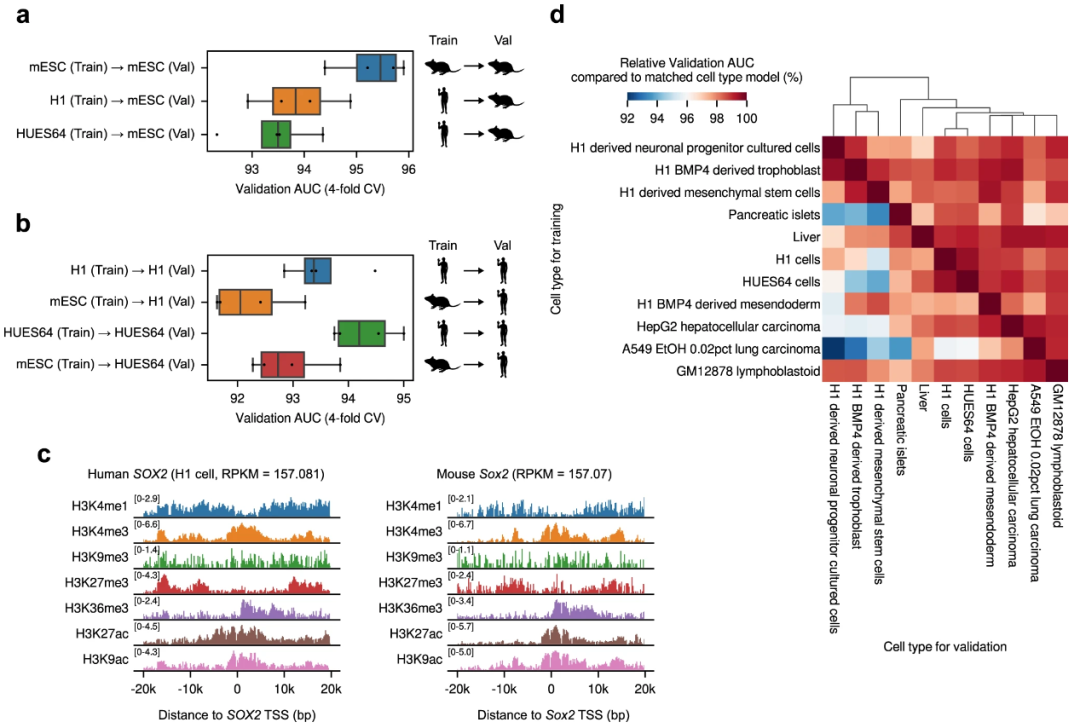
图示:Chromoformer 的跨物种和跨细胞类型预测性能。(来源:论文)
此外,跨细胞类型预测实验表明,在一种细胞类型中训练的 Chromoformer 模型在一定程度上仍适用于其他细胞类型(相对验证AUC > 92%),相似细胞类型的交叉预测性能更高。
这意味着以细胞类型特异性方式训练的 Chromoformer 不仅学习了基因调控的细胞类型特异性特征,而且仍然捕获了可普遍应用于其他细胞类型的一般规则。如果它可以表示为全基因组信号值的数组,则可以扩展显色体训练以包含任何额外的表观基因组特征。这些特征包括转录因子 ChIP-seq 信号或用于隔间识别的第一主成分 (PC1) 信号。

图示:在 Chromoformer 训练中加入 CTCF 结合信号。(来源:论文)
论文中介绍,CTCF 结合是 3D 基因组结构的关键决定因素,并且通过远端增强子 - 启动子相互作用在基因激活中也突出了启动子 - 近端 CTCF 结合,如上图所示。该团队证明,包括 CTCF 在内的 Chromoformer-clf 性能有边际但一致的增加,而嵌入 Transformer 的 Chromoformer-clf 模型的增加更大。
另一方面,使用 PC1 值作为附加特征通知 Chromoformer 细胞类型特异性基因组划分状态不会导致显著的整体性能提升。尽管区室化与基因表达水平相关,但由于关联的绝对水平(皮尔逊相关系数 0.12–0.19)不够大,该团队认为,区室级特征的预测能力没有超过基因级 HM 特征的预测能力。
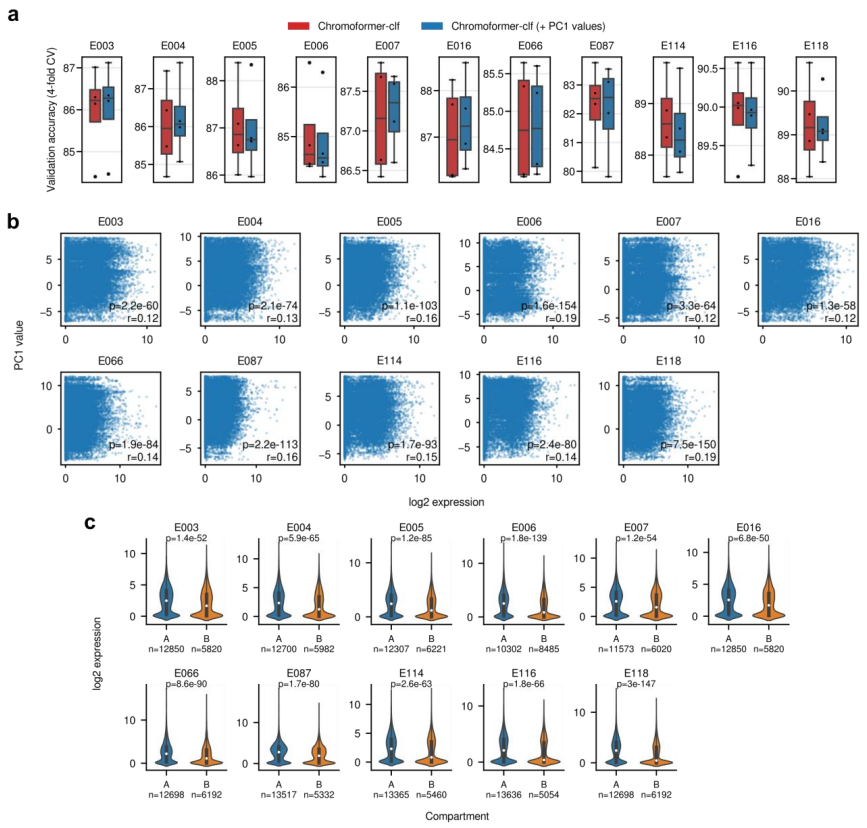
图示:在 Chromoformer 训练中纳入基因组划分状态。(来源:论文)总之,Chromoformer 是另一个示例性应用,它强调了 Transformer 架构在生物序列建模方面的巨大潜力。该研究还强调了开发有效嵌入生物先验知识的专业深度学习架构的重要性,不仅可以提高预测任务的性能,还可以定量表征生物实体之间的复杂关系。
论文链接:https://www.nature.com/articles/s41467-022-34152-5
人工智能 × [ 生物 神经科学 数学 物理 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。