多重免疫荧光成像允许以亚细胞分辨率对细胞环境进行多维分子分析。然而,从这些丰富的数据集中识别和表征与疾病相关的微环境具有挑战性。

在这里, 斯坦福大学James Zou教授提出了空间细胞图形建模 (SPACE-GM),这是一种几何深度学习框架,它采用图形神经网络 (GNN) 将细胞生态位结构或微环境灵活地建模为子图。子图的每个节点对应于一个由其多路复用蛋白质水平表示的细胞,并且边缘捕获邻居关系。作者将SPACE-GM应用于三个临床CODEX数据集,并表明它识别出与疾病相关的微环境,可以准确预测患者水平的表型。图深度学习模型在预测患者结果方面比基于细胞类型的局部组成对空间数据建模的深度学习方法更准确,并且它深入了解了肿瘤细胞和粒细胞的空间划分对患者的预后影响。局部图还可以帮助分析通过空间转录组学和其他组学技术表征的组织学样本中的疾病相关基序。该成果近期发表在《Nature Biomedical Engineering》。
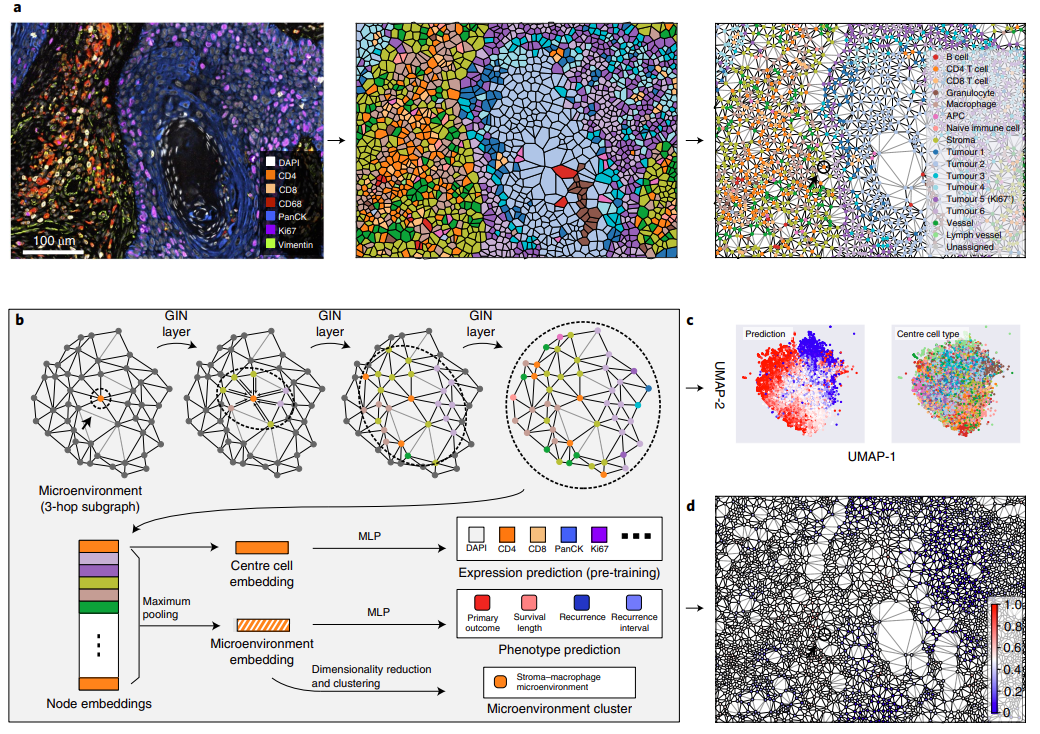
几何深度学习模型细胞微环境
以图形表示作为输入,作者提出SPACE-GM作为一种几何深度学习工具,可以读取TME中的空间细胞群落结构。SPACE-GM使用图同构网络 (GIN) 作为主干,使用多个多层感知器 (MLP) 作为预测头(方法)。根据经验,作者发现3跳邻域(平均对应40个单元)是一个合适的选择,因为子图的大小和模型性能是平衡的。此后将这些3跳本地子图称为“微环境”,这可能与在其他环境中研究的TME不同。相应地,SPACE-GM中采用了三层GIN,它基于节点特征(例如,单热编码的细胞类型)和边缘(图连接)为中心细胞和微环境构建嵌入。然后可以将嵌入通过预测头传递以生成对细胞特性(例如,中心细胞表达谱)和患者水平表型特性(例如,患者的生存结果)的估计。
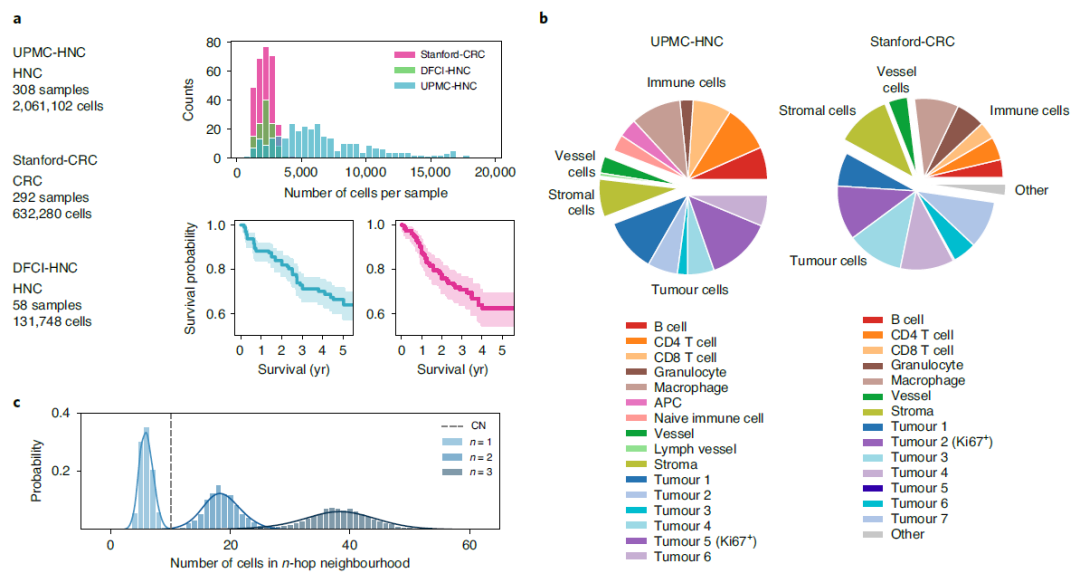
使用SPACE-GM定义疾病相关的细胞微环境
受SPACE-GM优于基于成分的基线方法的启发,作者进一步研究了成分之外的细胞群落结构的特征如何预测临床结果。SPACE-GM的预测准确度表明,它的嵌入空间(学习用数值向量表示每个微环境)提供了表型的信息。UMAP的可视化显示患者表型在嵌入空间中被很好地分离。作者使用来自UPMC-HNC数据集的主要结果任务作为示例来演示如何解释SPACE-GM嵌入以生成与患者结果相关的生物学假设。首先基于SPACE-GM嵌入(方法)对所有微环境进行聚类。簇显示出不同的细胞类型富集模式和预测值。
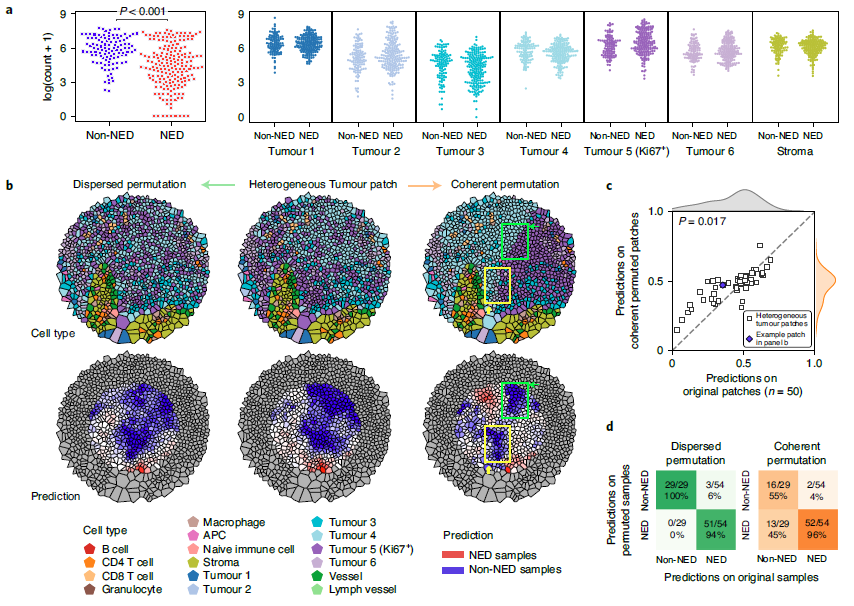
图3:微环境中节点的排列有助于识别影响预测的细胞间相互作用。总结:这项工作表明,空间细胞图是一种灵活的空间生物学建模方法。在此类图上训练的图神经网络可以识别预测患者对癌症治疗反应的空间基序。对来自分析的空间基序的后续计算和实验研究应该揭示更多关于肿瘤细胞之间相互作用的生物学见解,并促进下游诊断和治疗工具的开发。SPACE-GM可以识别可预测患者表型的关键空间结构。然而,由于细胞分类的局限性,将这些学习到的特征直接应用于具有非重叠细胞类型的未见数据集仍然是一个悬而未决的问题。作者希望探索的潜在解决方案包括根据人类细胞联合体的努力调整综合细胞类型或应用能够容纳看不见的细胞类型的方法。随着内容丰富的空间组学数据变得更加可用,作者期望诸如SPACE-GM之类的方法能够揭示生物学并识别有用的生物标志物。https://www.nature.com/articles/s41551-022-00951-w