基于信使 RNA (mRNA) 的药物具有巨大的潜力,正如它们作为 COVID-19 疫苗的快速部署所证明的那样。然而,mRNA 分子的全球分布受到其热稳定性的限制,这从根本上受限于 RNA 分子对称为在线水解的化学降解反应的固有不稳定性。预测 RNA 分子的降解是设计更稳定的基于 RNA 的疗法的关键任务。
在这里,斯坦福大学的研究人员描述了 Kaggle 上的众包机器学习竞赛(Stanford OpenVaccine),涉及对 6,043 种不同的 102-130 核苷酸 RNA 结构的单核苷酸分辨率测量,这些 RNA 结构本身是通过 RNA 设计平台 Eterna 上的众包征集的。整个实验在不到 6 个月的时间内完成,获胜模型中 41% 的核苷酸水平预测在实况测量的实验误差范围内。
此外,这些模型普遍适用于盲目预测更长的 mRNA 分子(504-1,588 个核苷酸)的正交降解数据,与之前发布的模型相比具有更高的准确性。这些结果表明,此类模型可以非常准确地表示在线水解,支持它们用于设计稳定的信使 RNA。两个众包平台的集成,一个用于数据集创建,另一个用于机器学习,可能有助于解决其他需要在快速时间尺度上进行科学发现的紧迫问题。
该研究以「Deep learning models for predicting RNA degradation via dual crowdsourcing」为题,于 2022 年 12 月 14 日发布在《Nature Machine Intelligence》。

基于信使 RNA (mRNA) 的疗法作为模块化治疗平台显示出巨大的前景,可以传递和翻译任何蛋白质,基于 mRNA 的疫苗针对严重急性呼吸系统综合症冠状病毒 2 (SARS-CoV-2)的快速部署就证明了这一点。然而,RNA 的化学不稳定性对基于 RNA 的疗法的稳定性设置了基本限制,其中 RNA 水解特别为基于脂质纳米颗粒 (LNP) 的制剂的稳定性设置了限制因素。LNP 制剂中的水解会降低运输和储存过程中剩余的 mRNA 的量,并且疫苗注射后体内的水解会限制随时间产生的所得蛋白质的量。开发耐热 RNA 疗法的更好方法将允许增加它们分布的公平性,降低它们的成本并可能增加它们的效力。
同义序列设计的前景是通往货架稳定性更高的 mRNA 疗法的未充分探索的途径。一个简单的计算表明,存在 10^633 个 mRNA 序列,它们都编码 SARS-CoV-2 刺突蛋白抗原。由于可用于给定治疗靶标的 mRNA 序列数量是天文数字,因此这些序列中的一些可能具有结构特征,使其比第一代 mRNA 疫苗制剂更耐水解。事实上,初步结果表明,可以通过优化候选 RNA 序列,为模型蛋白质系统设计更稳定的 mRNA,并使用 RNA 水解模型进行评分。这些初步研究表明,与未优化的 mRNA 相比,稳定的 mRNA 可以产生等量的蛋白质,并且对于某些设计,可以产生更多的蛋白质。预计这些设计策略能够产生不会激活双链 RNA 免疫传感器(如 RIG-I)的 mRNA。这些策略还证明了与由修饰的核苷酸合成的 mRNA 的相容性,包括用于 mRNA 疫苗制剂的假尿苷。
然而,任何此类 mRNA 设计算法的潜力都受到预测 RNA 降解的基础模型准确性的限制。以前的 RNA 降解模型假设任何 RNA 核苷酸连接被切割的概率与 5' 核苷酸未配对的概率成正比。使用该模型的计算研究表明,通过序列设计至少可以将稳定性提高两倍,同时保持与可译性、免疫原性和全局结构相关的序列和特征的广泛多样性。然而,降解不太可能仅取决于核苷酸未配对的概率:局部序列和结构特异性背景可能差异很大,自然界中发现的核酶 RNA 证明了这一点,其序列采用经历自我分裂的特定结构。

图:用于创建 RNA 降解预测模型的双众包设置。(来源:论文)
斯坦福大学的研究人员希望了解在模型开发的短时间内可实现的 RNA 降解的最大预测能力。为此,他们结合了两个众包平台:RNA 设计平台 Eterna 和机器学习竞赛平台 Kaggle。「RNA 设计」的问题涉及设计具有特定目标特性的 RNA 序列,例如特定的整体结构、目标功能(例如传感器活性),或者在这种情况下,具有高化学稳定性。研究人员使用了在 Eterna 平台上设计的短 RNA 片段的降解数据,其中包含多种序列和结构,并假设众包获得机器学习架构的问题将产生一个模型,该模型能够表达由此产生的序列复杂性和结构相关的退化模式(图 1a)。研究人员假设这种「双重众包」将导致对开发的模型进行严格和独立的测试,最大限度地减少设计测试结构的个人(Eterna 参与者)与构建模型的个人(Kaggle 参与者)之间的假设共享,并导致在独立数据集上具有更好的普遍性。
由此产生的模型受到了两次盲目预测挑战。第一个是在 Kaggle 竞赛的背景下,参与者旨在预测的 RNA 结构探测和降解数据直到比赛宣布后才获得。用于这些数据的实验方法 In-line-seq 允许测量单个核苷酸连接的降解率。然而,这种方法依赖于探测短 RNA 片段,无法扩展以对感兴趣的蛋白质靶标的全长 mRNA 进行单核苷酸降解测量。其他实验方法,如 PERSIST-seq 已被开发用于表征每个 mRNA 分子的总体降解率,这是在设计稳定的基于 RNA 的疗法时要最小化的主要兴趣值。原则上,长度为 N 的 mRNA 分子的总降解率等于骨架中每个二核苷酸键的降解率之和: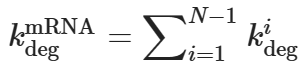 ,其中
,其中 是核苷酸连接 i 的降解。mRNA的半衰期计算如下,
是核苷酸连接 i 的降解。mRNA的半衰期计算如下,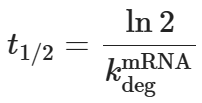 。
。
研究人员通过比较每个核苷酸的总降解率与测序剩余的整个构建体的丰度来凭经验测试上述模型,并发现高度一致(扩展数据图 1)。使用上述 ansatz,在第二个盲目挑战中测试了生成的模型,该挑战预测编码各种模型蛋白质的全长 mRNA 的整体降解,使用 PERSIST-seq 进行实验测试。这些模型还证明了在预测这些总体降解率方面比现有方法具有更高的预测能力。因此,这些模型立即可用于指导低降解 mRNA 分子的设计。模型性能分析表明,预测 RNA 降解模式的任务受到可用数据量以及用于创建输入特征的结构预测工具的准确性的限制。实验数据和二级结构预测的进一步发展,与此处开发的网络架构相结合,将进一步推进 RNA 降解预测和治疗设计。

图:竞赛中使用的深度学习策略。(来源:论文)
讨论
OpenVaccine 竞赛独特地利用了两个互补的众包平台的资源:Kaggle 和 Eterna。Kaggle 竞赛的参与者的任务是预测单个 RNA 核苷酸的稳定性测量值。及时开发稳定的 COVID-19 mRNA 疫苗的紧迫性要求比赛在相对较短的三周时间内进行,而不是三个月,这在 Kaggle 比赛中更为常见。
此处介绍的模型可立即用于 mRNA 设计,因为它们可以在随机 mRNA 设计算法中调用,以最大限度地减少预测的降解。可能还有进一步的机会利用自然语言处理的进步来使用此处介绍的数据集来使用文本生成方法生成 mRNA 设计。本次比赛中使用的降解数据来自用未修饰的核苷酸合成的 RNA,但 mRNA 疫苗是用修饰的核苷酸配制的,包括假尿苷或 N-1-甲基假尿苷。修饰的核苷酸通常具有不同的潜在热力学,因此需要开发数据集和预测模型来预测结构和由此产生的用修饰核苷酸配制的 mRNA 的稳定性。In-line-seq 方法可以使用带有修饰核苷酸的 RNA 来执行,生成的数据可用于重新训练具有此处介绍的架构的模型。如果不为修饰的核苷酸开发全新的热力学参数,就有可能开发有原则的启发式算法,使模型适应用修饰的核苷酸合成的 mRNA。例如,Leppek 团队修改了假尿苷的 DegScore 模型,将所有尿苷降解测量值设置为零以模拟假尿苷的稳定作用,并看到相关性得到适度改善。

图:Kaggle 模型在全长 mRNA 降解的独立测试中表现出改进的性能。(来源:论文)
数据集相对较小的 Kaggle 比赛可能会严重过度拟合公共排行榜,这通常会导致在宣布未见过的测试集的结果时排行榜的「重组」。在这场比赛中,变动很小——大多数顶级团队在私人排行榜上的排名与他们在公共排行榜上的排名接近。由于私人排行榜是根据比赛开始时尚未收集的数据确定的,因此这一结果表明这些模型是稳健且可推广的。
斯坦福大学的研究人员展示了前两个模型概括为预测全长 mRNA 分子降解的任务,这些分子比用于训练的结构长十倍。研究人员推测,使用单独的、独立收集的数据集进行私人排行榜测试——真正的盲目预测挑战——对于确保普遍性很重要。获胜的解决方案都结合了常用于建模一维序列数据的神经网络架构,包括多头注意力、循环神经网络(LSTM 和 GRU)和一维 CNN。伪标签的有效性有两个含义:更多的数据可能会有益于任何未来的建模工作,并且所使用的简单架构有足够的能力从更多的数据中受益。

此处介绍的模型的一个研究不足的方面是训练对多种数据类型的影响。研究人员认为,由于 SHAPE 反应性比退化数据类型具有更高的信噪比,具有允许数据类型之间权重共享的体系结构的模型也受益于学习预测 SHAPE 反应性。在不同时对 SHAPE 数据进行训练的情况下直接预测 RNA 降解可能会导致模型性能变差。相反,此处介绍的模型架构也可能被证明在仅预测 SHAPE 反应性数据方面具有有用的生物学应用。模型开发的未来方向包括在来自更多不同实验来源的更大的化学映射数据集上训练此类模型,并将其集成到 RNA 结构预测的推理框架中。
最后,在这项工作中开发的用于预测 RNA 水解的模型可能被证明可用于计算识别已经进化为具有抗降解性的天然 RNA 类别。这种未来的生物信息学分析可能会提出全新的生物学启发方法来设计抗水解 RNA 疗法。更直接的是,计算设计 mRNA 序列以优化本研究中发现的预测降解稳定性,并通过实验测试此类序列是否确实足够稳定以实现 mRNA 疫苗的更广泛分布将引起强烈兴趣。神经网络预测属性的计算机设计是一个活跃的研究领域,研究人员推测进一步的双众包研究可能有助于加速进展。
论文链接:
https://www.nature.com/articles/s42256-022-00571-8
人工智能 × [ 生物 神经科学 数学 物理 材料 ]
「ScienceAI」关注人工智能与其他前沿技术及基础科学的交叉研究与融合发展。
欢迎关注标星,并点击右下角点赞和在看。
点击阅读原文,加入专业从业者社区,以获得更多交流合作机会及服务。