
点评 | 戴琼海(中国工程院院士)、李栋(中国科学院生物物理所)
责编 | 兮
提高荧光图像的空间分辨率一直是光学显微成像领域的重要目标。2008年Mats Gustafsson等发明了一种实用化的超分辨成像技术“三维结构光照明显微镜” (3D SIM)【1】。它利用三光束干涉产生的莫尔条纹来激发样品以获得超越衍射极限的空间频率信息,并将这些信息编码在采集到的荧光图像之中。对这些原始图像进行解调和数学重建可以实现荧光超分辨成像。与宽场显微镜 (wide-field microscopy) 相比,3D SIM在三个维度上的空间分辨率都提高了一倍。虽然分辨率提升低于其他超分辨技术(PALM/STORM、STED/MINFLUX),3D SIM具有大视场、高速成像、无荧光基团标记限制等独特优势,并且在深度成像中具有真正的轴向切片能力,尤其适合活细胞成像,因此成为了当今细胞生物学、微生物学、活体显微成像领域中的一项重要研究工具。然而,3D SIM的轴向分辨率仍然局限于300 nm,远低于其100~120 nm的横向分辨率。这就导致3D SIM的重建结果是各向异性的:研究人员可以清楚地观察到横向平面 (x-y) 上的二维细节,但轴向平面 (z) 上的三维特征仍然是模糊的。
在名为I5S的后续工作中,Mats 和Lin Shao 使用了两个对置物镜产生的六光束干涉,可以进一步提高轴向分辨率【2】。由对置物镜收集到的双路荧光信号同时在像面产生干涉 —— 这种结合了相干激发和相干探测的成像技术得到了100 nm的各向同性分辨率。令人可惜的是,I5S较高的系统复杂度和操作难度制约了它的推广。此后,SIM 领域大多数的技术进展都集中在提高系统横向 (x-y) 分辨率上,而轴向 (z) 分辨率上的推动却鲜有人问津。但是大多数生物样品信息是存在于三维的,只关注x-y平面上的二维图像会忽视z方向上的重要信息。因此增加超分辨显微镜的轴向分辨率、获得各向同性的光学超分辨能力,对生命科学研究有着重要价值。
在这一领域的研究过程中,美国国立卫生研究院 (NIH) 的Hari Shroff研究团队近年来进行了广泛合作并取得了一系列卓越成果(MSIM: Nat Methods (2012); iSIM: Nat Methods (2013); Two-photon SIM: Nat Methods (2017); TIRF-iSIM: Nat Methods (2018); Multiview SIM: Nature (2021))。
为了进一步解决轴向分辨率问题,2023年1月26日,现已加入美国霍华德休斯医学研究所 (HHMI) 的Hari Shroff研究团队在Nature Biotechnology杂志以长文 (Article) 形式发表题为Three-dimensional structured illumination microscopy with enhanced axial resolution的研究论文,分别从光学和计算的角度出发,提出了两种互补的实用化方法来提高3D SIM的轴向分辨率:
(1)在3D SIM系统中只增加一个模块,就获得了160 nm 轴向分辨率,是3D SIM的两倍,宽场显微镜的四倍。
(2)在不改变3D SIM系统的情况下,开发了一种新的深度学习方案并获得了120 nm各向同性分辨率,同时与深度学习去噪相结合,最终实现了活细胞内跨越一百个时间点的四维超分辨成像。

研究团队首先在3D SIM中引入“驻波显微镜” (standing-wave microscopes)【3】的概念,将一个反射镜直接浸入样品液体中并垂直于物镜光轴,可以实现比3D SIM的三光束干涉有更多轴向高频分量的四光束干涉成像 (4-beam SIM)。与之前提出的设计相比【2, 4】,4-beam SIM具有以下优势:(1) 从双物镜改为单物镜,极大地降低了系统的搭建难度和维护成本;(2) 不再需要对多个光学元件进行主动反馈控制,而只需对反射镜和样品位置本身进行调节,进一步降低了实现难度;(3) 用于产生驻波的中心光束只在样品液体内反射一次,避免了折射率不匹配和光学器件引入的波前畸变,从而最大程度地保证了干涉面的大视场和平坦度;(4) 因为反射镜和样品之间的相干距离很短,所以常用激光器即可满足系统需求,从而避免了对已有的3D SIM进行改造。
本研究中的4-beam SIM在对3D SIM的轴向分辨率提高了一倍的同时,保留了3D SIM对宽场显微镜上横向分辨率的两倍优势,即120 nm 横向分辨率和160 nm轴向分辨率(近各向同性成像)。该研究团队展示了4-beam SIM能够在不同样品中提供清晰的轴向视图,包括100 nm小球、细菌外膜、线粒体外膜和其他双色标记样品(图1和2)。
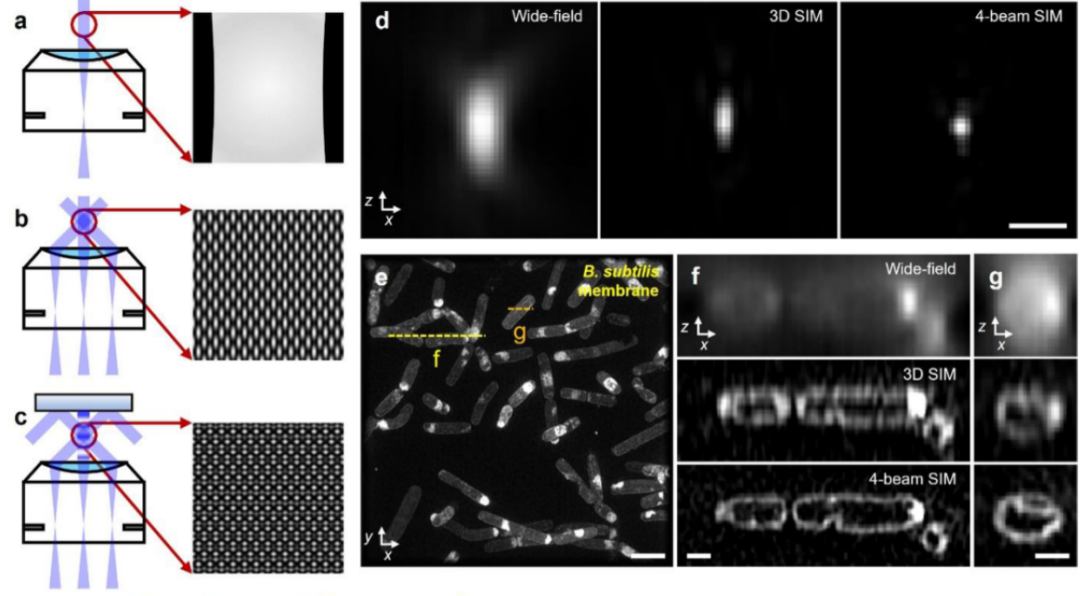
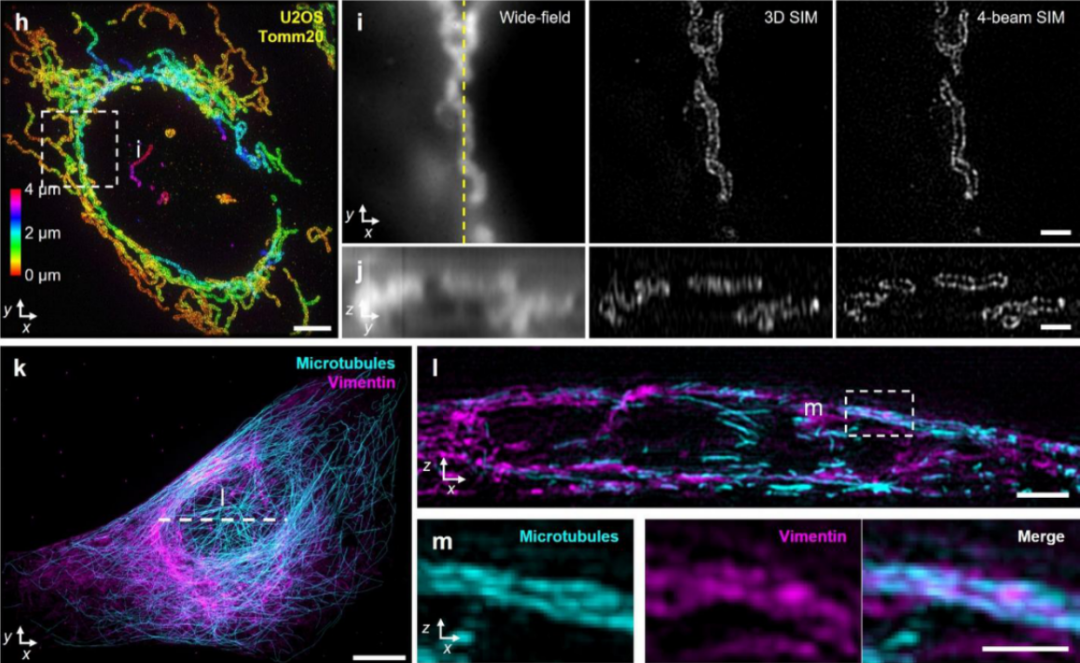
图1. 4-beam SIM实现近各向同性的超分辨成像。a-c) 物镜后焦面和样品平面的光路示意图。宽场显微镜(单光束照明,a),3D SIM(三光束干涉,b)和4-beam SIM(四光束干涉,c)。d) 100 nm绿色荧光小球的轴向横截面视图,分别由宽场显微镜(左)、3D SIM(中)和4-beam SIM(右)进行成像。e) 细菌外膜染色的最大强度投影。f, g) 对应e中虚线的轴向视图,从上到下依次为宽场显微镜、3D SIM和4-beam SIM。h) 在U2OS细胞中对线粒体外膜的抗体标记(颜色深度表示的最大强度投影)。i) 对应h中虚线区域的放大图。j) 对应i中虚线的轴向视图。从左到右依次展示了宽场显微镜、3D SIM和 4-beam SIM的成像效果。从d、f、g、j四幅图中可以看出四光束干涉对于z方向分辨率的显著提升。k) 在U2OS细胞中对微管(青色)和波形蛋白(紫红色)进行双色标记。l) 对应k中虚线的轴向视图。m) 对应l中虚线区域的放大图,可以从z轴方向清晰地观察到两种结构在细胞内部的分布与排列。比例尺:500 nm d, f, g); 4 µm h); 2 µm e, l); 1 µm i, j, m); 10 µm k)。
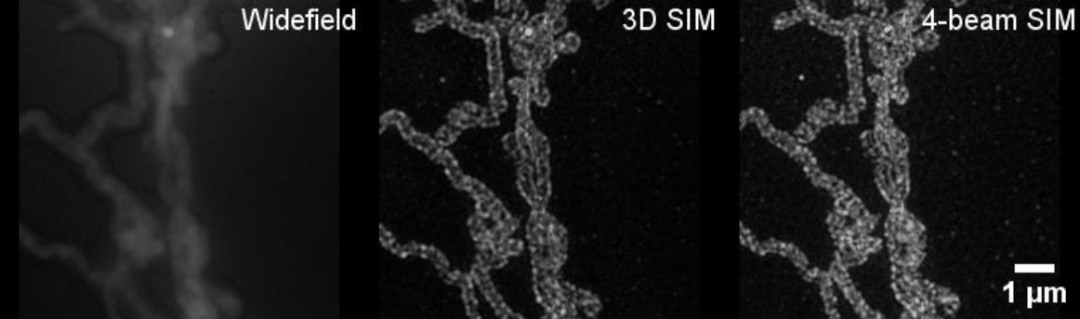
图2. U2OS细胞中线粒体外膜标记的三维投影动画。
此外,研究团队发现利用上述方法对细胞进行长时间活体三维成像 (4D) 仍然具有较大难度。即使在4微米的厚度内进行成像,4-beam SIM也需要多于1,000张的原始图像,所以采集过程中积累的光漂白 (photobleaching) 和光毒性 (phototoxicity) 成为了主要限制。相较于4-beam SIM,在同样的成像深度条件下,3D SIM(1)只需要不到一半的原始图像,(2)成像效果受波前畸变的影响更小,(3)并且可以实现持续的4D活体成像【5】。因此,研究团队考虑设计另一种在不引入额外照明剂量的条件下提高3D SIM轴向分辨率的方法。这一问题包含了两个子问题:1.如何对低信噪比3D SIM图像中的噪声进行滤除?2. 如何提高3D SIM图像的轴向分辨率?
2021年,Shroff团队的陈记稷博士(NIH)等使用three-dimensional residual channel attention networks (3D RCAN) 【6】的深度学习方法,展示了它对4D荧光图像强大的去噪能力。当前团队在此基础上发现,如果直接用神经网络对3D SIM的原始图像或重建图像去噪能一定程度上提高信噪比,但恢复到的图像并不完美。因此,该研究整合了3D SIM中经典的维纳滤波重建算法和两步3D RCAN去噪模型,最终获得了远优于单步去噪模型的结果。
另一方面,研究团队的吴一聪博士(NIH)和韩晓霏博士(清华大学)在近期的工作中提出了一种增强荧光图像横向分辨率的新策略 (体积分辨率提高10倍!吴一聪等《自然》发文报道共聚焦显微镜新突破:https://blog.sciencenet.cn/blog-3423233-1314124.html)【7】。这个方法在content-aware image restoration networks (CARE) [8] 深度学习的基础上,通过旋转-预测-融合的重建策略将一维超分辨扩展到整个二维图像。本研究利用类似的方法来提高3D SIM的轴向分辨率:由于在轴向视图中x轴总是比z轴具有更好的分辨率,所以本研究通过学习x轴上较高的分辨率来提高z轴甚至其他任意方向的分辨率。具体上,沿x轴模糊处理图像得到类似z轴效果,以此训练一个神经网络来逆转这种人工模糊。然后将得到的神经网络应用于6个沿y轴旋转不同角度的轴向视图,将结果反向旋转回原始角度后即得到沿6个方向的超分辨图像。最后通过融合这些图像即可实现一个具有各向同性分辨率的深度学习结果。此外,研究团队还验证了使用另一种深度学习方法Richardson-Lucy Network (RLN) 也可用于提高轴向分辨率(详见BioArt报道:Nat Methods | 深度学习结合成像物理模型显著提高荧光显微图像质量)。该研究团队以4-beam SIM的超分辨图像为参照,对深度学习模型的预测结果进行了验证。结果表明,参照和预测图像具有高度的相似性,表明了此深度学习模型的可靠性(图3a-c)。
最后,该研究团队结合了上述的去噪模型和轴向分辨率增强模型。整个深度学习框架突破了现有超分辨显微成像技术的瓶颈,实现了从低信噪比输入到120 nm各向同性分辨率的高信噪比输出,展示出了对不同生物样品进行各向同性超分辨4D活体成像的强大能力(图3d-f)。
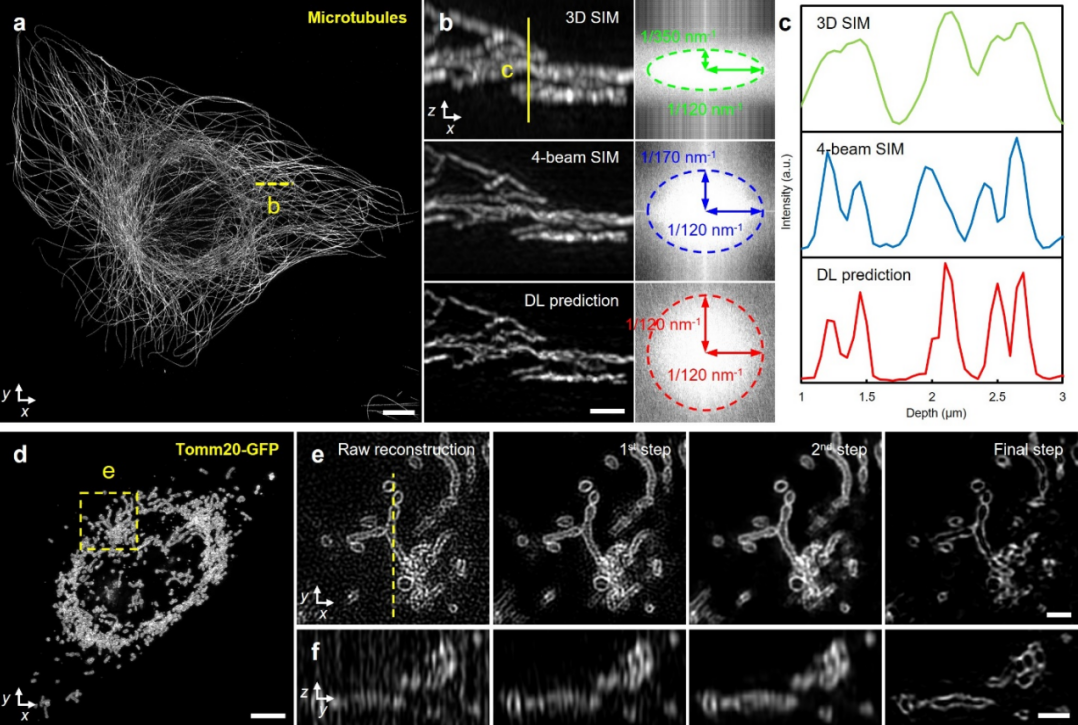
图3. 深度学习实现具有各向同性分辨率的 4D 超分辨率成像。a) 在U2OS细胞中对微管进行抗体标记(最大强度投影)。b)图左:对应a中虚线的轴向视图;图右:左图的傅里叶变换;从上到下依次为:3D SIM、4-beam SIM和深度学习结果。c) 沿b中实线所绘的信号强度图,可以清楚地看到与 3D SIM 相比,4-beam SIM 和深度学习都提高了区分精细结构的能力。d) Tomm20-GFP标记的活U2OS细胞(最大强度投影)。e) 对应d中虚线区域的放大图。f) 对应e中虚线的轴向视图。e和f充分展示了原始图像通过去噪模型和分辨率增强模型,逐步提升图像质量的过程。比例尺:5 µm a, d); 500 nm b, e, f)。
综上所述,Hari Shroff研究团队从光学系统和深度学习两条路径出发,显著地提高了结构光照明超分辨显微镜的成像能力,拓展了该技术在生物样品中的应用。团队的后续工作旨在开发更多新技术来提高显微镜的空间分辨率、时间分辨率和成像深度,并进一步推动深度学习与显微成像领域的融合与创新,为生命科学的发展提供有价值的研究工具。
霍华德休斯医学研究所(HHMI/Janelia Research Campus)李雪松博士为该论文第一作者, 并与美国国立卫生研究院生物医学成像和生物工程研究所(NIH/NIBIB)吴一聪博士为该文章共同通讯作者。
戴琼海(中国工程院院士、清华大学自动化系、清华大学脑与认知科学研究院、清华-IDG/麦戈文脑科学研究院):
大约400年前,列文虎克用自己磨制的透镜改良了光学显微镜,打开了人类观察微观生物世界的大门。近年来,随着成像理论与技术的进步,光学显微镜不断向着更高的时空分辨率与更大的成像尺度迈进,逐渐成为支撑脑科学、细胞生物学等生命科学领域发展的重要观测手段。作为当前应用最广泛的三维超分辨成像技术之一,三维结构光照明超分辨显微镜(3D-SIM)借助三束光干涉产生的激发光对生物样本信息进行调制,再通过数学解析算法进行解调重建,相比于常见的宽场显微镜,可以将横向(x-y)和轴向(z)分辨率同时提升一倍。然而由于光学显微镜固有的轴向频域信息缺失问题,3D-SIM的轴向分辨率仅为横向的三分之一左右,造成空间分辨率的各向异性。
为解决这一问题,美国霍华德休斯医学研究所(HHMI)Hari Shroff研究团队的李雪松博士和吴一聪博士从光学系统设计和计算成像两个角度出发,提出了两种改进措施:(1)利用一个垂直于光轴的反射镜形成四光束干涉的结构光照明模式,获得更高的轴向激发频率,进而通过SIM重建得到~120120160纳米的准各向同性空间分辨率;(2)基于深度学习训练一维超分辨神经网络,再利用旋转-预测-融合的重建策略最终得到~120纳米的各向同性分辨率,同时为延长成像时程,使用RCAN去噪网络和两步去噪法将3D-SIM的成像时程延长至数十个时间点。两种方法从不同的角度入手,相互独立而各有所长:基于光学系统改进的第一种方法带来的轴向分辨率提升由物理原理与适定的解析重建算法保证,成像可信度与保真度更高,然而需要引入反射镜模块和提升轴向采样率,造成更高的光毒性与光漂白;基于深度学习算法的第二种方法可以直接适配在任意一台3D-SIM系统甚至其他光学成像系统上,带来的时空分辨率提升更显著,同时应用成本更低。
总而言之,Hari Shroff研究团队所提出的方法可以十分便捷地应用在实验室自主搭建或商业购买的3D-SIM系统上,带来稳定的轴向分辨率提升,极大地拓展了三维超分辨活体成像技术在生命科学研究中的应用范围,为探索亚细胞结构互作与动态规律等生命科学问题提供了新的技术路径。
李栋(中国科学院生物物理研究所):
近年来,以PALM/STOMR、STED和SIM为代表的超分辨显微成像技术逐渐成为生物医学研究展的重要工具。其中三维结构光照明超分辨显微镜(3D-SIM)兼具超分辨、高成像速度、低光毒性与光学层析能力等优势,成为最适合对活细胞进行三维显微观测的技术手段之一。然而,尽管3D-SIM能够在XYZ三个方向上将分辨率相较于衍射极限提升~2倍,依然无法解决单物镜模式下荧光信号采集在纵向的锥角缺失和不对称所导致的分辨率各向异性问题,即轴向分辨率(~300纳米)远低于横向(~120纳米)。
近日,霍华德休斯医学研究所Hari Shroff研究团队的李雪松博士和吴一聪博士针对3D-SIM分辨率各向异性的难题,从光学系统硬件和深度学习软件两个角度提出两种相辅相成的解决方案。在光学系统角度,通过引入一个紧贴生物样本放置的平面镜,形成“四光束干涉”的照明模式,使激发结构光在样本轴向形成更高的调制频率,再通过传统SIM重建算法完成三维超分辨重建,从而将轴向分辨率提升至160纳米左右。为进一步提升成像时程,该研究团队又提出了一种基于深度学习的多步去噪与各向同性超分辨重建算法,从而实现~120纳米各向同性分辨率的多色超分辨成像性能。
相比于基于双物镜的I5S、3D-iLLS等提升轴向分辨率的技术方法,Hari等人所提出方法无需引入第二个物镜光路,仅需对标注的3D-SIM光路进行微调(引入平面镜)或无需进行调整(使用深度学习方法),即可简便、高效、低成本地实现轴向分辨率的增强,有望成为细胞生物学等领域研究提供能够被广泛应用和普及的技术方案。
https://www.nature.com/articles/s41587-022-01651-1
制版人:十一
1. Gustafsson, M. G. L. et al. Three-Dimensional Resolution Doubling in Wide-Field Fluorescence Microscopy by Structured Illumination. Biophys. J. 94, 4957-4970 (2008).
2. Shao, L. et al. I5S: wide-field light microscopy with 100-nm-scale resolution in three dimensions. Biophys. J. 94, 4971-4983 (2008).
3. Bailey, B., Farkas, D. L., Taylor, D. L. & Lanni, F. Enhancement of axial resolution in fluorescence microscopy by standing-wave excitation. Nature 366, 44–48 (1993).
4. Manton, J. D., Strohl, F., Fiolka, R., Kaminski, C. F. & Rees, E. J. Concepts for structured illumination microscopy with extended axial resolution through mirrored illumination Biomedical Optics Express 11, 2098-2108 (2020).
5. Shao, L., Kner, P., Rego, E. H. & Gustafsson, M. G. L. Super-resolution 3D microscopy of live whole cells using structured illumination. Nat. Methods 8, 1044-1046 (2011).
6. Chen, J. et al. Three-dimensional residual channel attention networks denoise and sharpen fluorescence microscopy image volumes. Nature Methods 18, 678-687 (2021).
7. Wu, Y. et al. Multiview confocal super-resolution microscopy. Nature 600, 279-284 (2021).
8. Weigert, M. et al. Content-aware image restoration: pushing the limits of fluorescence microscopy. Nat Methods 15, 1090-1097 (2018).

【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。
