
在上个世纪,英国剑桥的医学中心分子生物学实验室(MRC-LMB)Sydney Brenner团队耗时14年,在1986年首次解析出秀丽隐杆线虫(C. elegans)的神经连接组,涉及302个神经元和7600个突触,并因此获得2002年的诺贝尔生理学及医学奖。今年3月,LMB的Marta Zlatic团队耗时12年,在Science上发布了迄今为止最完整的果蝇幼虫全脑神经元连接组图谱,涉及3016个神经元和神经元之间的54.8万个突触连接,这已是有史以来所能完成的最复杂的动物大脑图谱。
2023年9月28日,LMB的Jing Ren(任婧)团队联合西安交通大学、北京生命科学研究所,在Nature Methods以长文形式发表文章D-LMBmap: a fully automated deep-learning pipeline for whole-brain profiling of neural circuitry,借助深度学习与图像处理技术,开发了介观尺度下小鼠全脑神经环路的量化解析方法,使得神经环路与连接组的研究从几百到几万神经元的线虫、果蝇更易于进一步扩展至上亿神经元的小鼠中。

随着近些年来显微成像组织、神经元视踪以及组织透明化技术的发展,小鼠介观全脑高通量的成像数据飞速积累。然而由于小鼠神经元分布密集且极度复杂、脑成像方式多样等因素,下游的计算与分析任务正成为解析神经连接组学的主要技术瓶颈,目前仍缺少一套能完整实现全脑神经环路量解析的计算方法与工具。为此,任婧团队开发了一套基于深度学习的算法和工具D-LMBmap (deep-learning pipeline for mouse brain mesoscale automatic profiling),实现了从全脑神经元的自动分割及环路重建、跨模态的脑图像风格转换及脑区分割、到三维全脑配准及神经环路投射的整个流程。该项工作的主要成果包括:
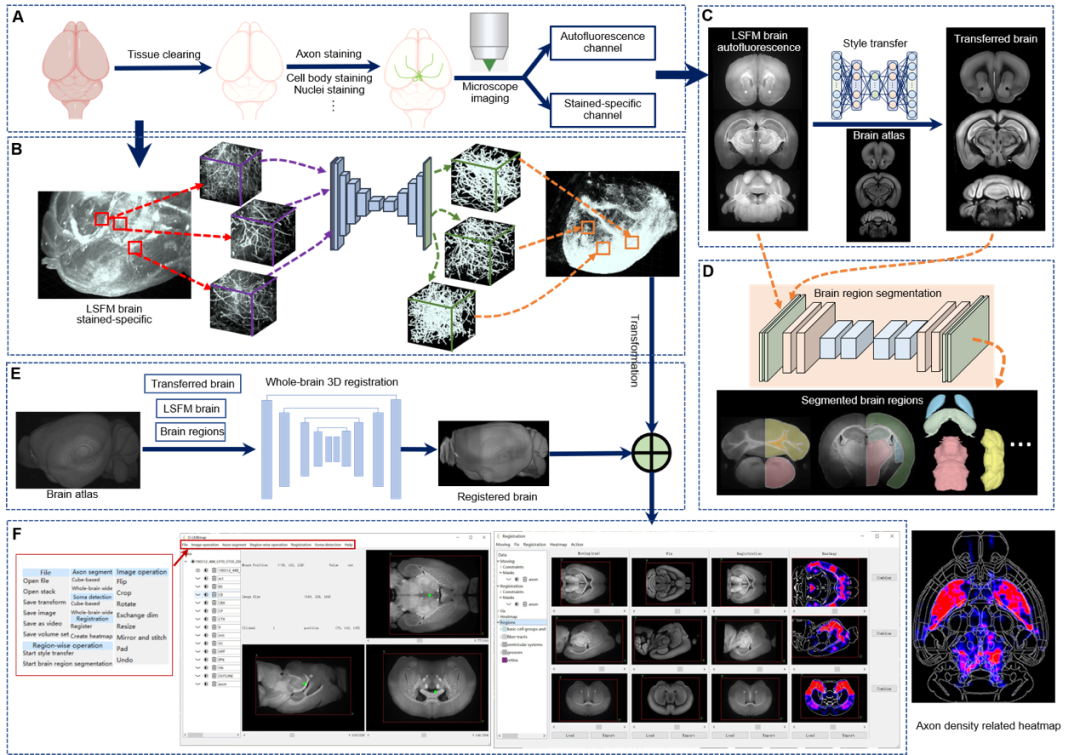
图1.D-LMBmap的主要工作:A.LSFM全脑成像流程,包括自身荧光(488 nm)和染色特异性(647 nm)两个通道;B.全脑神经元分割与环路重建;C.脑图像风格迁移,将LSFM脑图像迁移到Allen图谱的风格;D. 脑区分割;E.全脑的三维配准和环路在各脑区的投射;F.D-LMBmap的软件界面及量化解析结果
1. 全脑神经元的自动分割与环路重建:该团队首次开发了一套神经元的自动标注方法,通过筛选相对干净的神经元前景和背景立方体区域,结合多种数据增强技术,以此训练面向神经元分割的三维深度神经网络模型,极大的避免了繁琐且耗时的人工标注过程。该方法可在数个小时内快速实现全脑数据上(443)对各种类型(5-羟色胺、多巴胺、GABA等)的分割与全脑环路重建。该方法同样适用于由不同视踪方式标记的神经元,无论稀疏或密集都可以被准确。
2. 脑图像的风格转换与脑区分割:考虑到不同成像方式下的脑图像存在模态差异,D-LMBmap将图像的风格迁移技术扩展到了脑图像中,该方法可将光片显微镜成像(LSFM)、核磁共振成像(MRI)等模态的脑图像与目前广泛使用的Allen脑图谱(Allen CCFv3)图像进行风格统一,从而缓解了脑图像的跨模态问题。并以此开发了半监督多视角的脑区分割方法,在无需或仅需少量脑区标注的前提下,即可实现大脑中主要脑区的精确分割(如大脑皮层,纹状体,小脑皮层,脑干等)。
3. 多重约束下的三维全脑配准:这项工作开发了一种基于多重约束的多尺度三维全脑配准方法,将以上获取的具有Allen图谱风格的实验脑、以及自动分割的主要脑区作为配准网络中的约束,设计了一种由低分辨率到高分辨率级联的配准网络模型。该方法可以在几分钟内生成准确性和鲁棒性较高的跨模态配准结果(例如从LSFM脑配准到Allen CCFv3图谱),结合全脑神经环路的重建结果,可精确地量化数百个大脑区域的神经元密度。
4. 提供了用户友好且高效的软件工具:在以上所设计算法的基础上,团队进一步开发了一个用户友好且高效的软件工具,并为其配置了面向LSFM脑图像的深度神经网络模型,用户可以使用其进行三维脑图像的预处理、神经元自动分割、脑图像风格迁移、脑区分割、全脑配准以及连接组在各脑区的投射。D-LMBmap同时提供了软件的详细使用教程,无需计算机编程基础即可直接上手使用。
动态图. 5-羟色胺神经元的全脑分割结果
基于以上方法和所开发的软件工具,团队人员定量的解析出了5-羟色胺、多巴胺、GABA以及小脑中央核团在成年小鼠全脑的神经环路,给出了这些类型的神经元在数百个脑区的投射分布情况。这其中也包括了首个dorsal raphe 5-羟色胺神经系统全脑投射的三维图谱。
综上,D-LMBmap为神经科学研究提供了一个强大的计算分析工具,使得研究人员更好地理解大脑神经环路的结构和功能,有助于推动脑连接图谱、神经疾病机理等问题的进一步研究。作者们已将全部程序开放共享,并配有指导录像(https://github.com/lmbneuron/D-LMBmap)。
英国医学研究中心分子生物学实验室(MRC LMB)的任婧研究员为论文的通讯作者,李钟毓博士为论文第一作者(现为西安交通大学副教授)。西安交通大学的商增谊硕士、刘婧怡硕士、甄昊天硕士、朱恩涛硕士生为的共同第一作者。北京生命科学研究所(NIBS)钟诗琳博士、林睿研究员为共同作者。
MRC LMB位于英国剑桥,是首次破译DNA结构的研究所,迄今为止LMB的科学家们共获得过12次诺贝尔奖。任婧研究员于2020年在LMB成立实验室(https://www2.mrc-lmb.cam.ac.uk/group-leaders/n-to-s/jing-ren/),致力于5-羟色胺(5-HT/serotonin)神经系统在分子、细胞、环路和行为功能各个层面的研究【1,2】。实验室现有博士后职位空缺,诚挚欢迎申请。
原文链接:
https://www.nature.com/articles/s41592-023-01998-6
制版人:十一
1. Ren, J., Friedmann, D., Xiong, J., Liu, C. D., Ferguson, B. R., Weerakkody, T., ... & Luo, L. (2018). Anatomically defined and functionally distinct dorsal raphe serotonin sub-systems. Cell, 175(2), 472-487.
2. Ren, J., Isakova, A., Friedmann, D., Zeng, J., Grutzner, S. M., Pun, A., ... & Luo, L. (2019). Single-cell transcriptomes and whole-brain projections of serotonin neurons in the mouse dorsal and median raphe nuclei. eLife, 8, e49424.

【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。
