空间位置对于理解复杂生物组织结构至关重要。细胞和分子的相对位置在维系组织的生物功能和协同作用方面发挥着关键作用。近年来,空间转录组技术迅速发展,可在捕获多细胞、近似单细胞以至亚细胞分辨率的基因表达,为认识复杂的生物组织结构和细胞微环境提供了新的机遇。张世华课题组于近期分别发布了适应于不同空间转录组技术、不同生物组织的生物组织空间亚结构识别工具-STAGATE(详见BioArt报道:Nat Commun|张世华课题组开发空间转录组生物组织亚结构解析新工具-STAGATE)和生物组织多切片空间转录组数据整合新工具-STAligner(https://www.nature.com/articles/s43588-023-00528-w);并与合作者绘制了地中海涡虫再生过程中的三维空间转录组图谱-STAPR,系统鉴定了多个再生关键调控因子(https://www.nature.com/articles/s41467-023-39016-0)。
空间转录组图谱显示出显著的空间表达模式和空间可变基因(Spatially Variable Genes, SVGs)。常用的空间可变基因识别方法通常采用统计建模的方式。例如,SpatialDE利用高斯过程回归对每个基因的空间表达建模,然后通过似然比假设检验识别空间可变基因。然而,高斯过程回归处理大规模空间转录组数据非常耗时。为了解决该问题,SPARK-X采用非参数模型拟合基因表达协方差和空间协方差之间的依赖关系,利用非参数假设检验识别空间可变基因。然而,现有方法存在两个不足。首先,对每个基因独立进行假设检验,忽视了相关基因的空间表达模式的相互性和互补性。同时,由于空间转录组数据存在缺失和稀疏的特点,单独对每个基因进行假设检验会削弱假设检验的效力。其次,多数方法识别的基因没有关联特定的空间结构。
近日,中国科学院数学与系统科学研究院张世华课题组与合作者陈洛南、合原一幸在Nucleic Acid Research在线发表了题为STAMarker: determining spatial domain-specific variable genes with saliency maps in deep learning的文章,开发了基于深度学习显著图的空间域特异可变基因识别方法 STAMarker(https://github.com/zhanglabtools/STAMarker)。
STAMarker是一个基于集成深度学习并由三阶段组成的方法(图1):(1)训练多个STAGATE图注意力自编码器,学习数据的低维表示,然后基于该表示聚类得到空间域标签。STAMarker采用一致聚类集成多个自编码器的聚类结果,以获得稳健的空间域;(2)STAMarker基于空间域类别标签,训练多个多层感知机(MLP)分类器来建模对应嵌入和空间域之间的映射关系;(3)STAMarker将编码器与对应的MLP堆叠在一起,并通过反向传播计算显著图(saliency map)。STAMarker的核心思想是,显著影响空间域分类的基因(通过显著图度量)对于理解该空间域的生物功能至关重要。
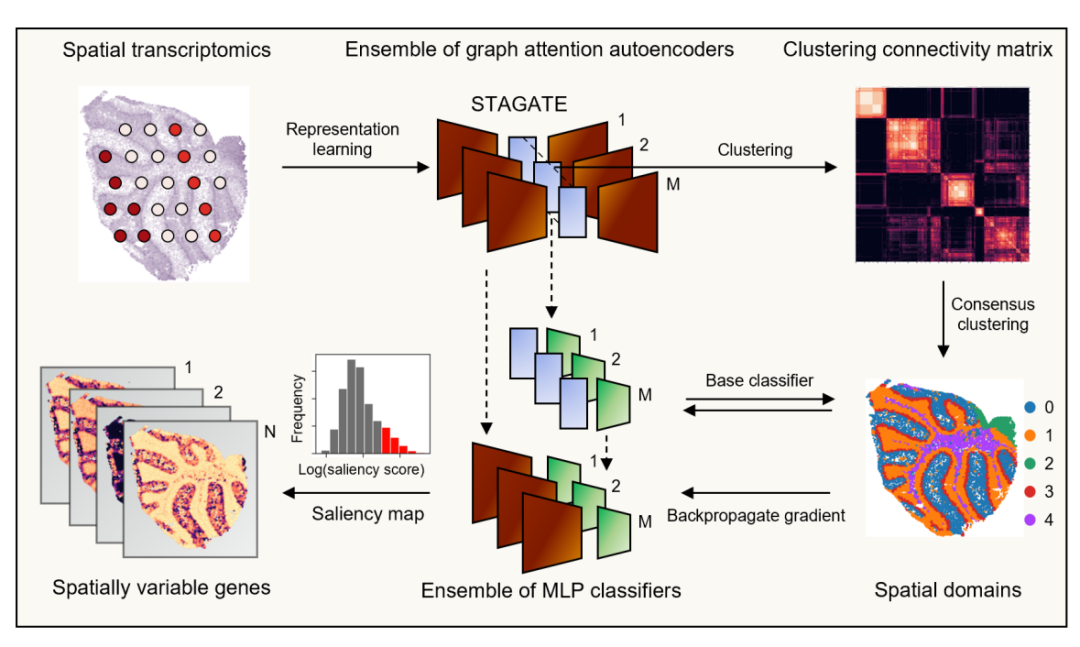
图1. STAMarker工作流程示意图
与SpatialDE和Spark-X等传统方法相比,STAMarker在稀疏数据上取得了更加一致稳健的空间可变基因。与传统方法不同,STAMarker可以识别空间域特异的空间可变基因。STAMarker在阿兹海默小鼠模型的海马体Slide-seq数据中发现了小胶质细胞特异的空间域,并发现了对应该空间域的多个空间域特异可变基因(如P2ry12、Trem2、Hexb和 Cx3cr1等)。小胶质细胞在阿尔茨海默症中通过参与炎症反应和清除异常蛋白质等方式对疾病的发展产生影响,对于理解阿尔茨海默症的进展有重要意义。STAMarker在小鼠嗅球和小脑的多种空间转录组数据,与其他方法相比,STAMarker识别的空间特异基因往往在更多的有意义的生物学过程、通路或功能上富集。这些结果表明,STAMarker在捕捉与特定的空间环境和生物学上下文相关的基因方面具有显著的优势,有助于更全面地理解组织结构和功能之间的关系。
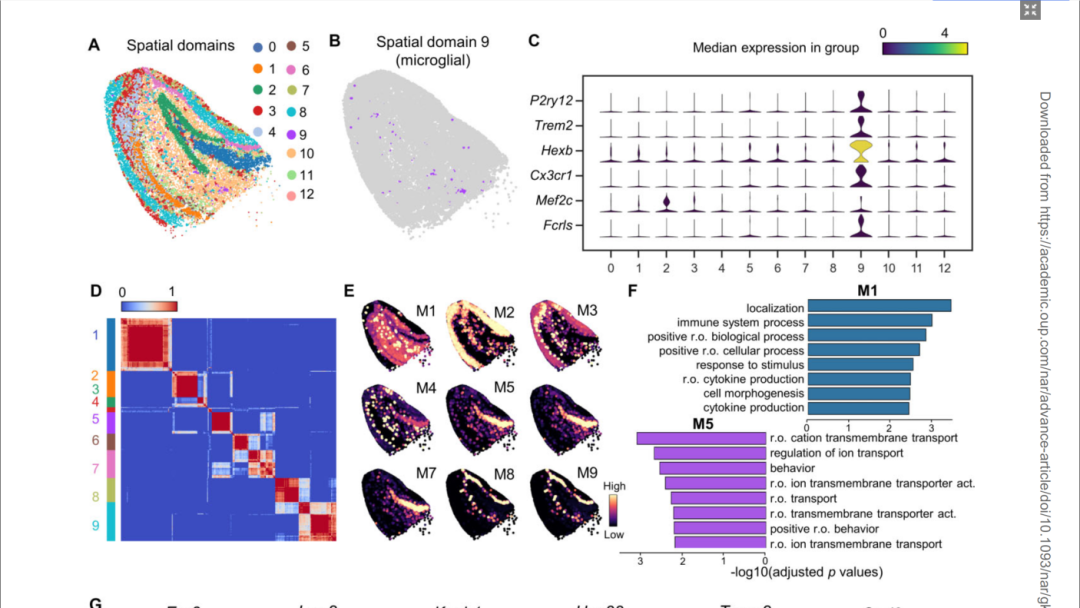
图2. STAMarker揭示小鼠模型海马体阿兹海默症特异的空间可变基因
总的来说,STAmarker 是一种基于集成深度学习的空间特异可变基因识别方法,同时实现了空间域识别和对应的空间可变基因识别,为细粒度分析空间转录组数据提供了一种有效的方法。
中国科学院数学与系统科学院张驰浩助理研究员为该论文第一作者,董康宁博士提供了必要的协助,东京大学国际神经智能国际研究中心合原一幸教授、中国科学院分子细胞科学卓越中心陈洛南研究员和中国科学院中国科学院数学与系统科学研究院张世华研究员为本文的共同通讯作者。
张世华研究员课题组常年招收人工智能、生物信息学与计算生物学方向的博士后、科研助理和(客座)博士研究生,有意向的同学可投递简历。
https://jinshuju.net/f/ZqXwZt或扫描二维码投递简历原文链接:https://doi.org/10.1093/nar/gkad801
软件链接:https://github.com/zhanglabtools/STAMarker
制版人:十一

【非原创文章】本文著作权归文章作者所有,欢迎个人转发分享,未经作者的允许禁止转载,作者拥有所有法定权利,违者必究。
