
全基因组选择(Genomic selection, GS)是利用覆盖全基因组的高密度标记进行选择育种的新方法,简言之就是在全基因组范围内的标记辅助选择。全基因组选择作为新一代的育种技术,通过构建预测模型,根据全基因组估计育种值进行早期个体的预测和选择,从而缩短育种世代间隔,加快育种进程,节约成本,推动现代育种向精准化和高效化方向发展。统计模型作为全基因组选择的核心,极大地影响着全基因组预测的准确度和效率。传统预测方法基于线性回归模型,难以捕捉基因型和表型间的复杂关系。相较于传统模型,非线性模型具备分析复杂非加性效应的能力,机器学习(Machine Learning,ML)和深度学习(Deep Learning,DL)算法为解决大数据分析和高性能并行运算等难题提供了新的契机。
为了响应广大学员在该方向上的学习需求,北京金智研将于6月28~30日举办第三期全基因组选择学习交流会。届时,我们将邀请中国农业科学院作物科学研究所大数据智能设计育种创新团队首席、博士生导师李慧慧研究员为我们系统讲授基于机器学习和深度学习算法的全基因组选择课程。李老师长期从事基于高通量组学数据全基因组选择育种预测算法的研发和模型构建等生物信息学、统计基因组学和数量遗传学相关研究;在Molecular Plant, Nature Plants, Plant Biotechnology Journal, Trends
in Plant Science等期刊发表第一/通讯作者45篇,研究成果获得2023年度海南省十大杰出农业科技成果;获国家基金委优秀青年科学基金项目资助,获中国农学会青年科技奖,并入选中国农科院“农科英才”领军人才;担任中国作物学会智慧农业专业委员会秘书长。
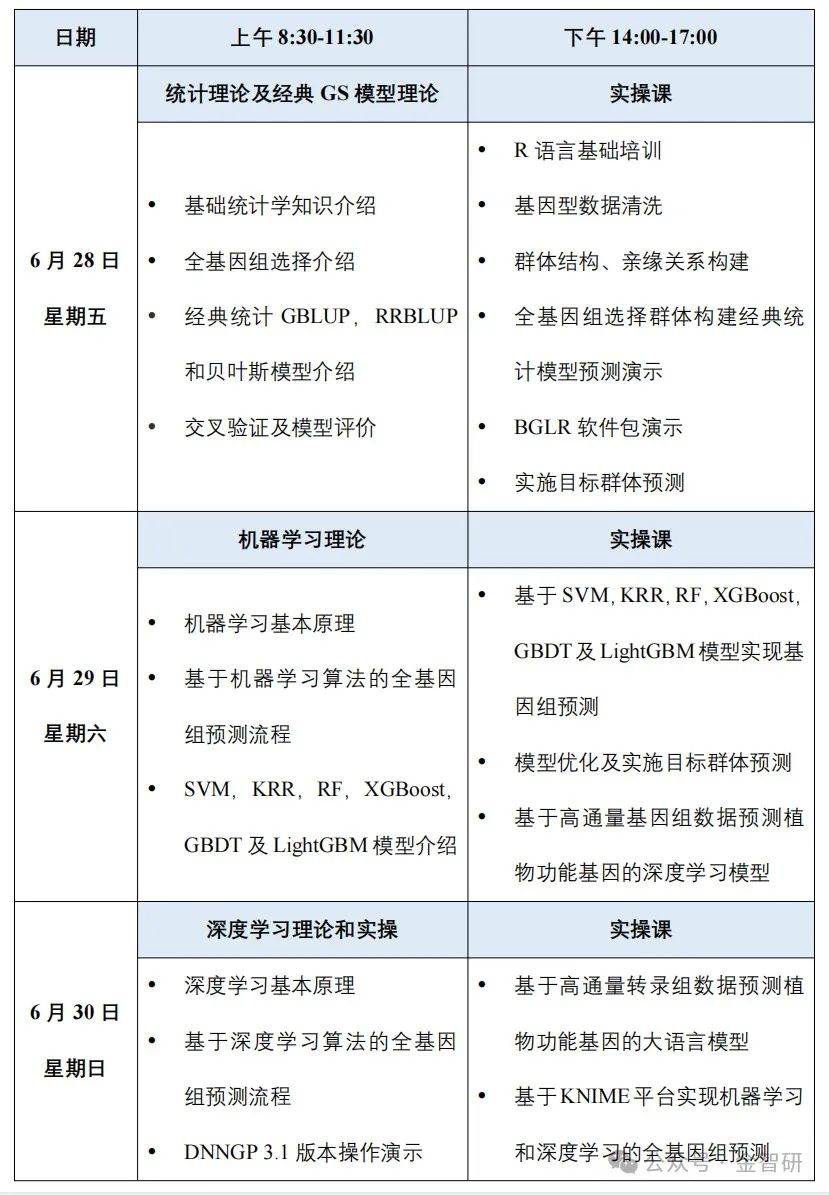
本次课程,李老师做了精心的策划设计,仍然遵循理论学习+实操演练相结合的原则,兼顾身处不同学习阶段学员的需求,将从统计学基础知识、基因组选择概述入手,涵盖了利用经典统计(GBLUP,RRBLUP)和贝叶斯模型、基于机器学习算法的全基因组选择模型(SVM,KRR,RF,XGBoost,GBDT,LightGBM)、基于深度学习算法的全基因组选择模型(DNNGP)实现基因组预测;模型优化及实施目标群体预测;特别是相较于前面两期,新增了基于高通量基因组数据预测功能基因的深度学习模型以及利用高通量转录组数据预测功能基因的大语言模型。在此基础上,李老师团队对每个模型都会做详细的预测演示和交叉验证结果评价并实现可视化,带大家利用KNIME平台构建深度学习网络,最终让我们利用组学大数据基于深度学习算法开展全基因组预测。课时三天,旨在解决我们在实操过程中遇到的痛点、难点,相信将为大家呈现一场全基因组选择的视听盛宴。
形 式:腾讯会议线上直播(课程免费回放三个月)
时 间:2024年6月28~30日
所有从事植物、动物、林木、水产、微生物等的遗传育种、数量遗传学及相关领域研究的在读研究生、科研人员及企业研发人员。
1. 熟练掌握基因组大数据统计分析和群体结构亲缘关系构建;
2.系统学习经典统计GBLUP、RRBLUP和贝叶斯模型,能够独立运用经典统计模型进行全基因组预测,实施目标群体预测;
3.系统学习基于机器学习算法的全基因组预测流程,能够独立运用SVM、KRR、RF、XGBoost、GBDT及LightGBM模型进行全基因组预测,进行交叉验证结果评价,实现预测结果的可视化,模型优化及实施目标群体预测;
4. 系统学习基于高通量基因组数据预测功能基因的深度学习模型以及利用高通量转录组数据预测功能基因的大语言模型;
5.系统学习DNNGP软件与KNIME平台的使用方法,利用KNIME平台构建深度学习网络,实现基于机器学习算法的全基因组预测。
前期准备:
1. 硬件要求
携带配有英伟达GPU的个人电脑
2. 软件安装
1)安装anaconda或miniconda任选其一即可(DNNGP自动脚本必须miniconda)
2)安装R及Rstudio软件平台
3)配置Jupyter Notebook环境
4)安装Pycharm开发平台或Vscode代码编辑器(选择安装)
5)安装KNIME软件
3. 个人要求:
具备一定的数量遗传理论基础。
注:相关软件和预习资料请在报名交费后于会务人员处获取,电话15210096658(微信同号)
李慧慧,研究员,博士生导师,中国农科院作物科学研究所大数据智能设计育种创新团队首席科学家、作物基因与分子设计中心副主任,三亚中国农业科学院国家南繁研究院副院长。自2005年以来一直致力于作物数量遗传学研究,围绕数量性状遗传解析方法及育种应用,研制了系列方法和软件工具,在国内外多个科研及育种单位广泛应用。在Molecular Plant, Nature Plants, Molecular Ecology, Trends in Plant Science等期刊发表第一/通讯作者45篇,其中单篇他引超过500次的论文3篇,单篇他引超过100次的论文9篇。获国家基金委优秀青年科学基金项目资助,获中国农学会青年科技奖,并入选中国农科院“农科英才”领军人才。担任农学国际主流期刊Frontiers in Plant Science分刊主编,Theoretical and Applied Genetics, Journal of Integrative Agriculture (JIA) 和《作物学报》编委,中国作物学会智慧农业专业委员会秘书长。
余廷熙,研究方向为机器学习,智慧育种,在线发表学术论文三篇(其中SCI收录一篇,EI检索两篇),并申请软件著作权一项。
张昊,从事农业育种领域大数据以及深度学习软件工具研发等方向的研究,新型农业基因选择工具DNNGP的主要维护人员。
学时3天,3200
元/人(包含3个月的视频回放)。
优惠1:三人及以上成团,团购价2900元/人。
优惠2:早鸟优惠,6月15日之前报名并交费尊享3100元/人。
(温馨提示:两项优惠任选其一,不可叠加使用。)
扫描或长按识别下方二维码,填写报名信息,提交并支付汇款,即为报名成功,报名截止于2024年6月27日17:00,届时报名通道将自动关闭。

1. 转账汇款
收款单位:北京金智研生物科技有限公司
开户银行:中国工商银行股份有限公司北京中海凯旋支行
账号:0200205009200045825
2. 个人扫码支付
对于不能及时由单位转账的学员,可接受个人垫付的形式,我们在收到报名费后,会及时为您开具发票协助办理报销事宜。扫描下方二维码(支持公务卡、银联卡、信用卡)即可完成支付。如遇问题,请致电或微信:15210096658。
温馨提示:汇款时请备注“姓名+全基因组选择”

以下链接为本期培训班的《会议通知》,请自行下载保存。
请准确填写报名表单的发票信息。在此过程中,您有任何问题可以先行与我们沟通联系,我们都将为您做出详细解答,电话:15210096658(微信同号)。
电话:杨老师:15210096658
徐老师:15001277701,010-88608002
北京金智研生物科技有限公司是中国作物学会单位会员,公司下设“智研在线”学习交流平台,平台立足于生命科学领域的科技传播与分享,专注于研究前沿科技,分享专业实用的科学知识、研究进展、软件使用技巧,现已成功举办了生物信息学、高级生物统计、基因组选择、GWAS实操、科研绘图、表观遗传学、基因编辑、分子互作等一系列精品课程,是为您提供学习、交流、分享、互动的一站式服务平台。