上一期我们带大家入门机器学习的数据预处理实战(二),希望大家从这2期数据预处理的知识分享中有所收获与体会!本期我们将开始针对初步处理的数据进行特征工程学习与实践,让数据更有说服力。
特征工程指的是利用领域知识和现有数据,通过手动或自动方法转为适合模型输入与对结果预测有用的特征的系统工程。机器学习的特征工程类似于临床预测模型的变量筛选,筛选出适合模型的变量,用于模型的构建。
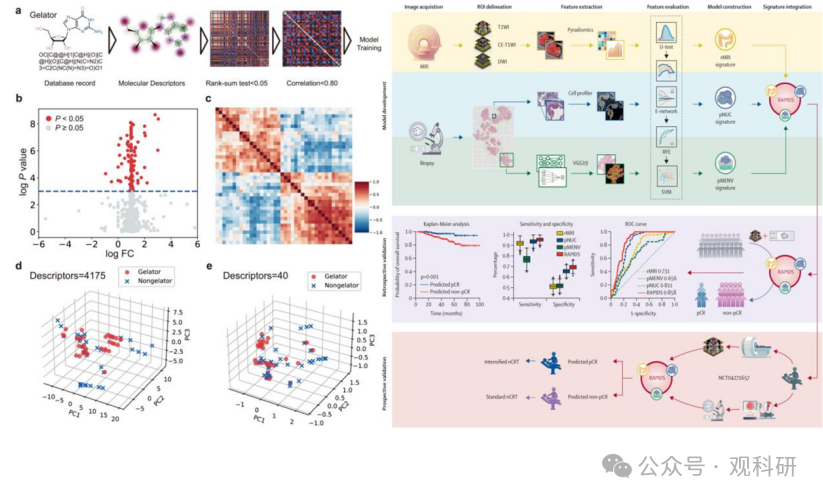
广义上,特征工程包括前两期介绍的数据预处理、特征选择、特征提取、特征构造等,往往互相联系、互有交集。特征工程的好坏能决定模型的上限,改进算法只不过是逼近这个上限而已。模型输入的特征越好,灵活性会越强、构建的模型会越简单、模型的性能会越出色。今天我们将剖析特征工程之特征选择,选择论文中常使用的方法进行R语言实战,包括U检验、递归特征消除、Lasso回归、森林之神Boruta等(一般在训练集开展特征工程,如何划分数据集见第3期推送)。
U检验通常指的是Mann-Whitney U检验,这是一种非参数检验,用于比较两个独立样本的分布是否相同。在特征选择的背景下,Mann-Whitney U检验可以用来评估一个特征在不同类别的样本中是否有显著差异,从而判断该特征是否有助于分类。其R代码如下:
#加载安装包
install.packages("dplyr")
library(dplyr)
#假设data是我们已经加载的数据框,其中Class是类别标签
#data$class是分类变量,其余列是数值型特征
#拆分数据
set.seed(123)
dataing.samples $class%>%
createDataPartition(p = 0.8, list = FALSE) #按8:2比例进行训练集与测试集划分
traintesttable(test$class)
table(train$class)
numeric_vars #训练集中开展特征选择
train[, c(2:ncol(train))] result_gky variable = character(),
p = numeric(),
stringsAsFactors = FALSE
)
for (var in names(numeric_vars)) {
result $class)
p $p.value
result_wx variable = var,
p = p
))
} #对训练数据集的所有特征进行循环的U检验
result_wx$p_adjusted $p, method = "fdr")
result_wx $p_adjusted #获得每个的特征P值后,按P小于0.05筛选出适合模型的特征
result_wx dim(result_wx)
dput(result_wx) #获得最终的特征
递归特征消除(Recursive Feature Elimination,RFE)是一种特征选择方法,用于自动选择对于目标变量预测最重要的特征。它通过递归地构建模型并剔除最不重要的特征来实现这一目标。
install.packages("caret")
install.packages("mlbench") # 用于获取示例数据集
library(caret)
library(mlbench)
data(Sonar) #使用mlbench包的Sonar数据集
set.seed(123)
ctrl functions = rfFuncs, method = "cv", number = 10) #rfFuncs表示我们将使用随机森林作为基础模型,method = "cv"表示我们使用的是交叉验证,number = 10意味着我们使用10折交叉验证。
set.seed(123)
rfe_result #执行递归特征消除,尝试从1个特征到所有特征的所有可能子集,并找到最优的特征集合。
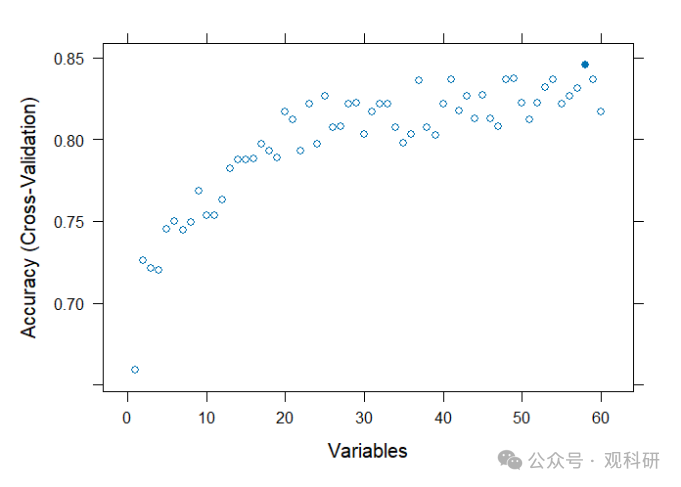
print(rfe_result) #每个子集中特征的数量及其对应的平均预测错误率
plot(rfe_result) #会生成一个图表,显示不同特征数量下的模型性能。
final_features dput(final_features) #获得最终选定的特征
上述代码结果如下所示:

以上代码示例仅为关键部分,实际应用可能要根据数据的需求和代码运行结果做适当调整。特征选择作为机器学习中极其重要的一环,并非一蹴而就,需要在考虑领域知识的基础上,持续优化特征、尝试多种方法结合,以提高模型性能、降低过拟合风险,加速模型预测速度。下一期我们将继续分享机器学习的特征选择,Lasso回归与Boruta的R实战。
今天的分享到这里就结束啦,你学会了吗?大家对于推送内容有任何问题或建议可以在公众号菜单栏“更多--读者的话” 栏目中提出,我们会尽快回复!
参考文献:
1. Li W, Wen Y, Wang K, et al. Developing a machine learning model for accurate nucleoside hydrogels prediction based on descriptors.Nat Commun. 2024;15(1):2603. Published 2024 Mar 23. doi:10.1038/s41467-024-46866-9
2. Feng L, Liu Z, Li C, et al. Development and validation of a radiopathomics model to predict pathological complete response to neoadjuvant chemoradiotherapy in locally advanced rectal cancer: a multicentre observational study.Lancet Digit Health. 2022;4(1):e8-e17. doi:10.1016/S2589-7500(21)00215-6
期待已久~临床科研交流群来啦!

若群满,请添加下方微信,备注:“进临床科研与统计_互助交流群” )
“观科研”(点击进一步了解我们吧)是由一群北京协和医学院(清华大学医学部)的博士开创的公众号,初心是让医学科研有迹可循,帮助一线的医学科研人员更快地成长,希望大家支持与关注!
如果大家对分享医学科研知识感兴趣,特别欢迎加入我们,期待与您的相遇相识相知,也非常欢迎大家自主投稿,如果您有需要分享的内容或对我们有任何建议,可通过后台留言、公众号菜单栏“更多—读者的话”栏目(进一步了解)或发送邮件至mascu_forever@163.com与我们交流并留下个人联系方式,我们会及时与您联系。如果您觉得我们长期的干货推送对您的科研工作有所帮助,可以在合适的机会致谢(包括但不限于SCI论文、毕业论文等),格式如下: The authors thank the support of Skill Learning from Kaixin Doctor and MASCU (Medical Association with Science, Creativity, and Unity), Inc, Shenzhen, China (mascu_forever@163.com).
微信公众号的推送规则发生改变(不再按照时间顺序来显示),如果没有将“观科研”设置为星标,你就可能错失里面的精彩推送。