新项目启动已经过去了一周,我总算是重新找回了写插件的感觉。暂时还达不到分享试用的程度,就随便跟大家聊点什么吧。
首先要说的是,为什么这个项目叫做“MolScaffold”?
其实在最初编写Mol3DStruct的时候,我就在考虑插件名称的问题。想来想去都觉得不合适,最后还是借用的2018年在3dsMax中写的分子插件的名称。
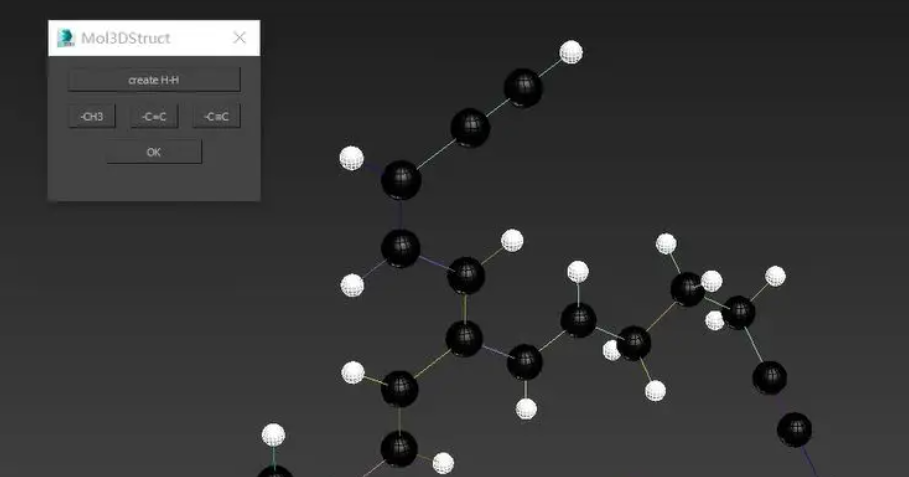
从2018到2024,这七年间我用过多款专业的分子可视化软件,包括Diamond、PyMol和Chimera等。对于分子结构的可视化也算是颇有心得。直到我接触到Blender软件之后,逐渐摸索出一套基于点线骨架+几何节点的可视化方法。
所以在开始这个2.0项目的时候,我毫不犹豫地选择了MolScaffold这个名字。
第二个想说的是如何写代码的问题。
我不是计算机专业,本科的时候只修过一门C语言的课。记得那会儿还是我人生中第一次接触电脑,只得了个B+的成绩。
真正编一点程序是在我研究生毕业前后那几年,主要用的是Fortran和Matlab。基本上属于自娱自乐,和专业人士比起来相差甚远。再之后,就是自学了一点Python基础,包括这个项目主要用到的Blender Python在内。
去年年底到今年上半年,我遇到代码上的问题基本都求助于Google。国外有专业的Blender Python相关论坛,大概能帮我解决70~80%的问题。剩下的靠自己摸索。拼拼凑凑,总算是把Mol3DStruct给整了出来。不少人说很好用,但和我预想的还是有些差距。如果打分的话,我给自己打65分。
其实,写插件这事儿,代码的问题还是其次。首先要考虑的是底层架构和用户体验,也就是说,我还得兼任产品经理的角色。
虽然Blender的几何节点很好用,但对于分子可视化这个庞大复杂的系统,节点数量是惊人的,节点网络是复杂的。我可不指望用户能搞明白其中的原理,于是我就想办法让节点看上去更加简洁有条理。
本来我是想用节点组的方式,但折腾了大半个月都没有用代码实现。最后,退而求其次,弄了下面这么个玩意儿。看上去是简洁了一些,但不多。
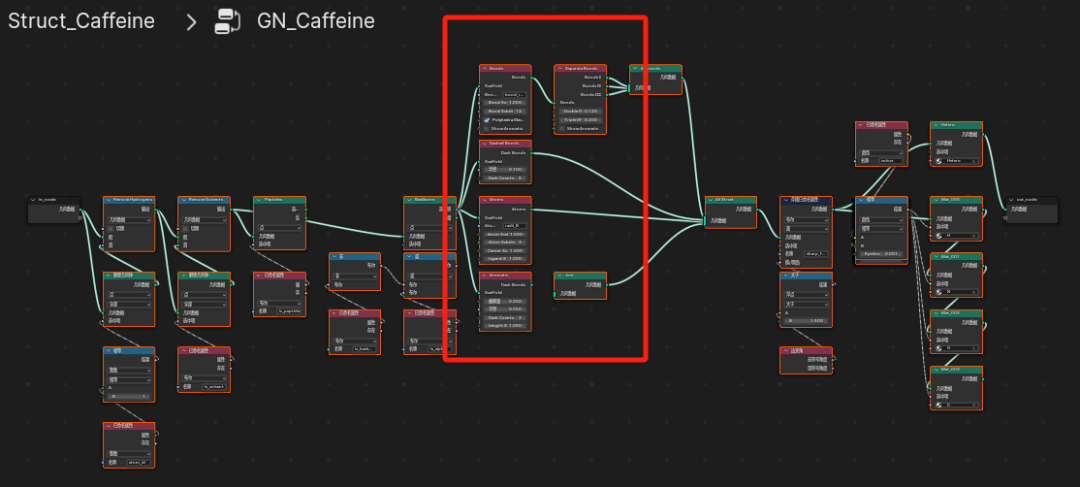
好在这次我尝试用ChatGPT辅助,这是我要说的第三个问题。
由于有了项目1.0的经验,在插件架构上我的思路很清晰,先生成分子骨架,然后筛选结构,选择可视化模式(包括结构和材质类型),最后一键渲染。遇到代码上的问题时,我就打开ChatGPT,比如当我问道:
如何用rdkit读取SMILES文本?
ChatGPT给我的回答是:
from rdkit import Chem
smiles = 'CCO'
mol = Chem.MolFromSmiles(smiles)
print(mol)
print(Chem.MolToSmiles(mol))
虽然这些代码在rdkit的API Reference中也能找到,但直接用ChatGPT可以节省大量的搜索时间。
于是,我花了半天时间,用ChatGPT搞定了之前一直困扰我的自定义几何节点组的问题。在MolScaffold中,你看到的节点基本是下面这样,简直不要太清爽!之后我会把一些必要的参数设置做成端口显示,方便用户调节。

最后,跟大家汇报下项目进展:
本次开发我是在Blender 4.2.1 LTS版本中进行的,应该会向上兼容至4.0版本。插件位置还是在View_3D窗口的右侧边栏。
这次除了分子文件外,我还考虑了分子名称、SMILES、结构式图片智能识别等多种读取方式。此外,还可以从PubChem和RCSB PDB数据库直接通过编号导入分子。
例如,Aspirin在PubChem网站的CID编号是2244。只需要在输入栏输入“2244”,然后点击“创建骨架”按钮,就可以导入Aspirin分子的点线骨架啦。
当然,你要是能写出Aspirin分子的SMILES符号,也可以直接创建。
今天就先跟大家聊到这里吧,我得继续工作去了。