新兴的空间转录组学(ST)技术能够在保存组织空间背景信息的同时捕捉基因表达,提升了我们对不同器官结构和细胞组成的理解。基于下一代测序(NGS)的ST方法,在测序前结合空间位置信息从组织中捕获转录本,能够高通量和无偏覆盖检测整个转录组的基因表达,且更容易获得商业化产品。但是测序技术仍然限制了创建精细分辨率空间细胞类型图谱的能力。
近日,中国国家生物信息中心杨运桂团队、中国科学院数学与系统科学研究院张世华团队合作开发了一种基于深度学习的空间转录组细胞注释工具STASCAN,通过整合基因表达谱和组织学图像的细胞特征学习来预测组织切片未知区域的细胞类型,并对捕获区域内的细胞进行细分注释,从而大大提高空间细胞分辨率。STASCAN还成功地仅从相邻切片的组织学图像中生成细胞分布图,从而以更低的实验成本构建更详细的器官3D细胞图谱。此外,STASCAN适用于来自不同ST技术的不同数据集,并在破译高分辨率细胞分布和解决增强的组织结构方面显示出显著优势。该成果发表在Genome Biology上,文章题为“STASCAN deciphers fine-resolution cell distribution maps in spatial transcriptomics by deep learning”
STASCAN采用了基于ST技术提供的空间基因表达图谱以及相应的组织学图像的深度学习模型。利用这些多模态数据,STASCAN通过生成捕获区域和未知区域中的spots或细分spots的细胞类型注释,描绘组织中精细分辨率的细胞分布图(图1a)。首先,STASCAN基于位置信息从切片图像中提取spots图像,并在预注释过程中使用去卷积方法推断出每个spot的高置信度细胞标签。然后,STASCAN构建了一个基础卷积神经网络(CNN)模型(VGG16 架构),并使用已标记的细胞类型spots图像对其进行训练。此外,STASCAN还提供了可选的针对特定切片的训练,通过迁移学习对基础CNN模型进行微调,以提高对特定单个切片的预测准确性。经过充分训练后,STASCAN可以仅通过组织学图像准确预测细胞类型(图1a)。STASCAN设计为三个应用模块:1)对未知区域内嵌入的未见spots进行细胞注释,实现超分辨细胞分布(图1b);2)对细分spots进行细胞注释,获得亚分辨率细胞分布(图1c);3)对未知切片进行细胞注释,构建3D细胞模型(图1d)。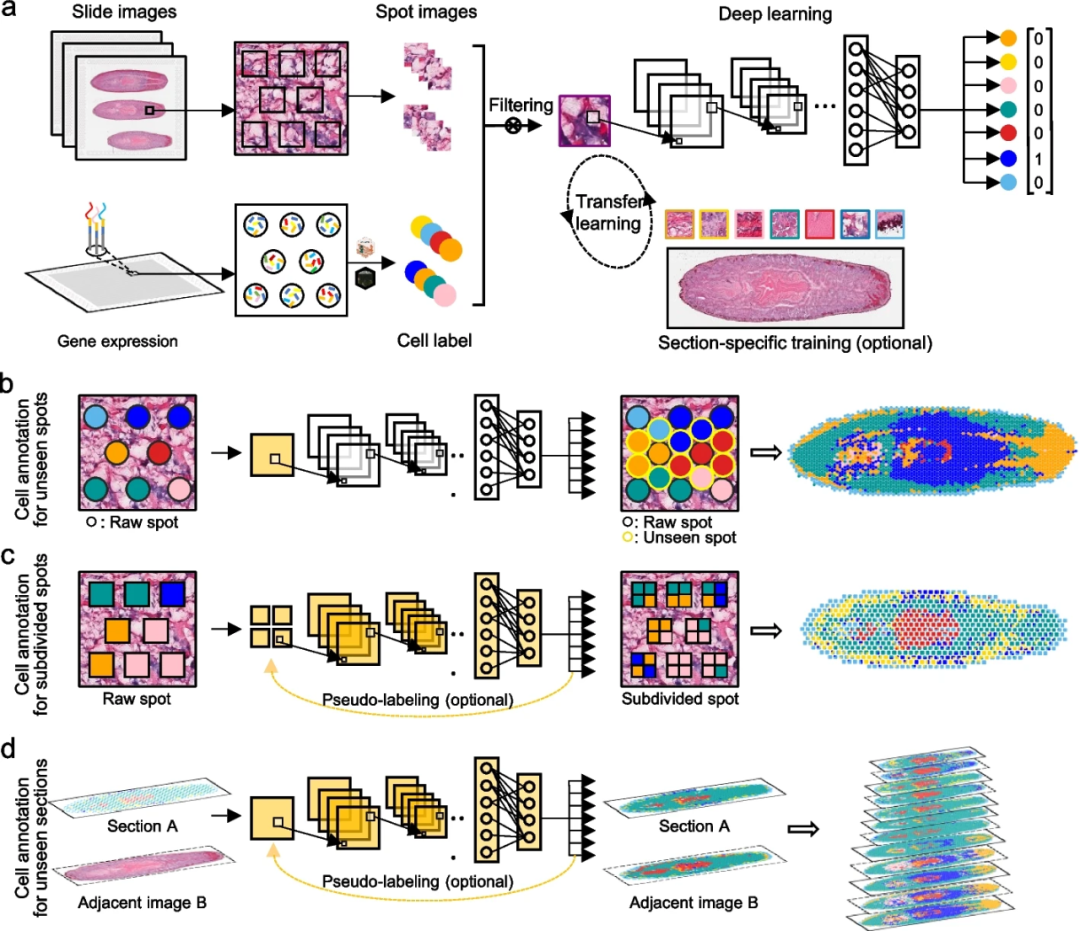
图1.STASCAN概述
为了定量评估STASCAN的性能,研究团队首先将其应用于由10× Visium技术生成的数据集,从10个已收集切片中提取了1829个spots图像,构建了基础模型,以学习通过测序信息鉴定出的7种主要细胞类型的特征,包括表皮、肠道、肌肉、干细胞、神经元、间质细胞和分泌细胞,大多数细胞类型的召回率均超过78%(图2a)。该学习模型在预测细胞类型方面表现出极高的准确性,ROC曲线下面积(AUC)高达0.936-0.996(图2b)。考虑到不同ST切片之间可能存在的批次效应,研究人员基于基础模型进行了切片特定训练。切片特定模型显示出比基础模型更高的准确性,AUC值显著提高,表明切片特定训练有助于整个框架的预测性能提升(图2c)。
进一步将STASCAN在原始spots上预测主要细胞类型的性能与其他方法进行比较,如Cell2location、Seurat和RCTD。STASCAN与手动注释的结果高度一致,且显著优于其他方法(图2d, e)。同时,STASCAN与其他结合形态学特征和转录特征的方法进行比较,发现STASCAN在预测中的表现更为精确,能够准确定位7种主要细胞类型的空间分布,并与它们已知的生物功能一致(图2d)。
相较于现有方法,STASCAN另一个重大进展是其能够仅基于相应的spots图像准确预测细胞类型,实现了与同时提供图像和基因表达数据时一致的精确细胞注释预测。
图2.STASCAN在10 × Visium数据集中的评价
接下来,研究团队使用扁形动物数据集评估了STASCAN在不同应用模块中的能力。在带有可靠标签的原始spots作为训练模型的优先spots后,STASCAN基于图像实现了可靠的细胞类型注释,并描绘了超分辨率的细胞分布图(图3a,b)。
此外,STASCAN准确定位了细胞混合物的组成及其在亚分辨率下的具体位置,有效区分了每个细分spots的细胞类型,并显示了更详细的细胞分布(图3c)。STASCAN还在亚分辨率下识别出位于咽和肠交界处的肌肉细胞;预测了肠细胞的增强亚分辨率分布,获得了肠细胞的细粒度分布,并重现了扁形动物肠道的经典分支结构(图3d)。以上结果表明,STASCAN显著增强了亚分辨率下的细胞粒度,有助于细粒度细胞描绘重要的亚结构。
最后,STASCAN利用相邻ST切片的学习特征,实现了仅通过H&E图像来预测未知切片中的细胞分布(图3e)。研究人员选择了两张相邻的ST切片作为测试数据(第 21 切片)和真实数据(第23切片),分别训练了两个STASCAN模型(一个基于第21切片,另一个基于第23切片),并使用这些模型仅基于H&E染色图像预测第23切片的细胞分布。结果显示,从第21切片训练的模型可以预测第23切片中的细胞分布,并与真实数据和直接通过第23切片训练的模型的预测结果高度相关。以上结果进一步验证了STASCAN在未知切片细胞注释方面的可靠性。此外,研究团队从ST切片和相邻图像中生成了原始spots和未知spots,应用STASCAN预测其细胞类型,并为不同结构重建了带有细胞模式的3D模型(图3g)。该模型展示了三维细胞分布,具有改进的空间细胞分辨率,促进了不经ST测序的染色图像的利用。
图3.STASCAN为10× Visium数据集提供全面和多维的细胞注释此外,为进一步评估STASCAN对不同组织结构ST数据集的识别性能,研究团队将其应用于人类肠组织、肺组织、心脏组织、小鼠大脑组织进行分析。结果显示, STASCAN识别出人类肠组织不同细胞层的清晰边界,绘制了人类肠组织中细胞亚型的空间分布图;STASCAN显示了人类肺组织更精确的细胞和结构模式,并敏感地识别位于气管壁旁的平滑肌束,该结构与H&E染色图像高度一致。同时,STASCAN 扩展了生态位分布的应用,并提供了更好理解细胞微环境相互作用的见解。在胚胎小鼠脑组织中,STASCAN揭示了发育阶段的主要解剖组织,在大脑发育过程中,大脑结构的复杂性逐渐增加。

图4.STASCAN显示了10× Visium人肺数据中的特殊结构
计算算法的发展对空间转录组数据的分析至关重要。该研究团队开发的STASCAN是一种通用的、精确的细胞类型预测方法,整合了组织学图像和空间基因表达信息,以增强的空间分辨率确定组织中细胞的复杂空间分布。STASCAN在不同组织样本中的应用已经证明了其在提高空间分辨率和发现新结构方面的卓越性能,为解决不同类型组织在不同发育、再生和疾病阶段的细胞类型分布提供了潜力。此外,STASCAN充分利用了各种生物条件下更容易获得的组织图像数据集,可以潜在地用于推断病理细胞的亚型,并通过广泛的数据训练进一步将空间细胞分布与疾病诊断联系起来,从而消除了对测序的需要。论文原文:
Wu, Y., Zhou, JY., Yao, B.et al. STASCAN deciphers fine-resolution cell distribution maps in spatial transcriptomics by deep learning. Genome Biol 25, 278 (2024). https://doi.org/10.1186/s13059-024-03421-5
·END·

快点亮"在看”吧