
随着人工智能的快速发展,深度神经网络能够从蛋白质结构或序列数据中学习高阶特征,深入掌握蛋白质序列与结构的关系,从而实现功能蛋白的高效设计。然而,大多数深度学习模型仍主要用于单链骨架的设计,难以充分应对更复杂的蛋白-蛋白相互作用(protein-protein interaction, PPI)设计挑战。为此,复旦大学药学院戚逸飞/王任小团队开发了专门用于PPI界面序列设计的模型——ProBID-Net。该模型能够根据给定的受体蛋白结构,预测配体蛋白界面上的氨基酸序列,并在多项测试中展现了优良的性能。近日,该项研究成果已在Chemical Science期刊在线发表【1】。ProBID-Net首先对目标残基及其周围相邻链残基主链原子在三维网格中的密度分布进行提取,随后利用20种天然氨基酸的原子密度信息编码,存储在不同的通道中,作为界面相互作用特征的输入,最后输出各位置氨基酸的概率。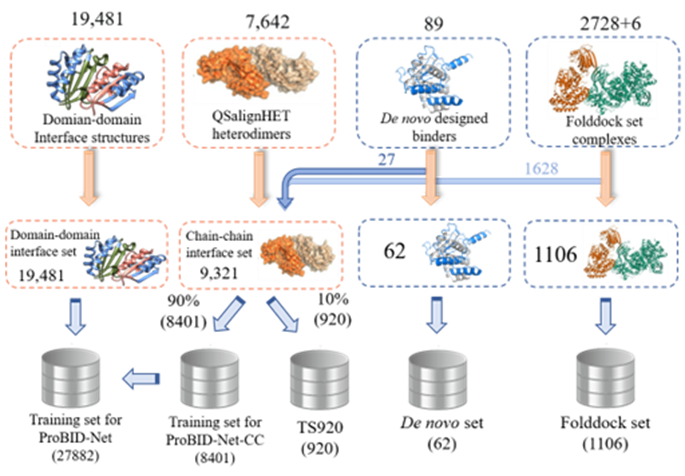
图1:ProBID-Net模型训练与测试结构数据处理流程图
ProBID-Net使用QSalignHET标记的PDB异源二聚体界面结构作为训练集,补充结构域-结构域界面作为补充训练数据,并收集了近年从头设计的蛋白-蛋白复合物结构以及来自Folddock【2】的数据作为测试集。训练集和测试集按设计链序列一致性小于40%的冗余度阈值重新聚类划分(图1)。界面序列平均恢复率和困惑度用于评估序列设计模型对PPI界面氨基酸残基的识别和设计能力。结果显示,结构域-结构域界面结构数据的加入提升了模型对界面残基的预测准确性。ProBID-Net和2024年诺贝尔化学奖得主David Baker教授研究小组开发的ProteinMPNN【3】在三个测试集上的界面残基恢复率相当(图2左上),但ProBID-Net具有更低的困惑度(图2右上),预测置信度更高。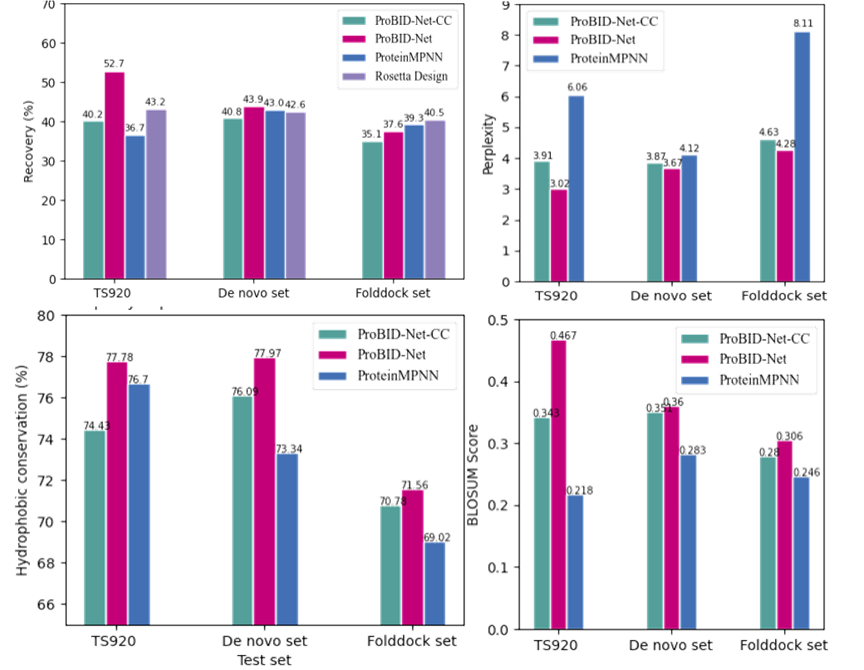
图2:ProBID-Net与ProteinMPNN在三个测试集与多项测试指标对比
界面残基的疏水保守性测试结果表明,ProBID-Net的疏水保守性优于ProteinMPNN(图2左下)。结构域-结构域界面数据的引入增强了模型对界面残基疏水位点的保守能力。ProBID-Net的BLOSUM分数最高(图2右下),更符合天然蛋白中氨基酸的分布和替换规律。我们将De novo测试集中配体链的界面残基替换为ProBID-Net最高概率预测的氨基酸,并使用AlphaFold2 multimer进行结构预测。图3显示,ProBID-Net设计的序列能够良好地折叠为原复合物结构,且链间
PAE较低,表明ProBID-Net在结合界面设计方面优于ProteinMPNN。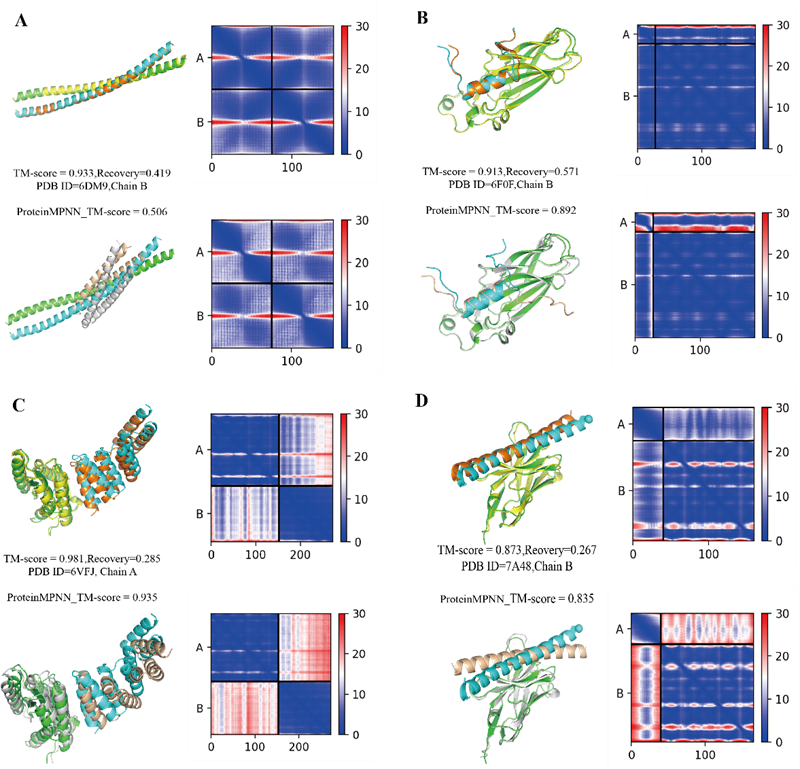
图3:ProBID-Net(上)和ProteinMPNN(下)设计序列预测结构叠合比对与AlphaFold-Multimer预测链间PAE热图
蛋白-蛋白结合亲合力零样本测试表明,ProBID-Net在未经过突变样本训练的情况下,依然可以凭借泛化能力预测蛋白复合物的结合亲合力变化(图4)。ProBID-Net预测的界面残基上有益突变,可用于增强蛋白-蛋白复合物的结合亲合力和整体稳定性。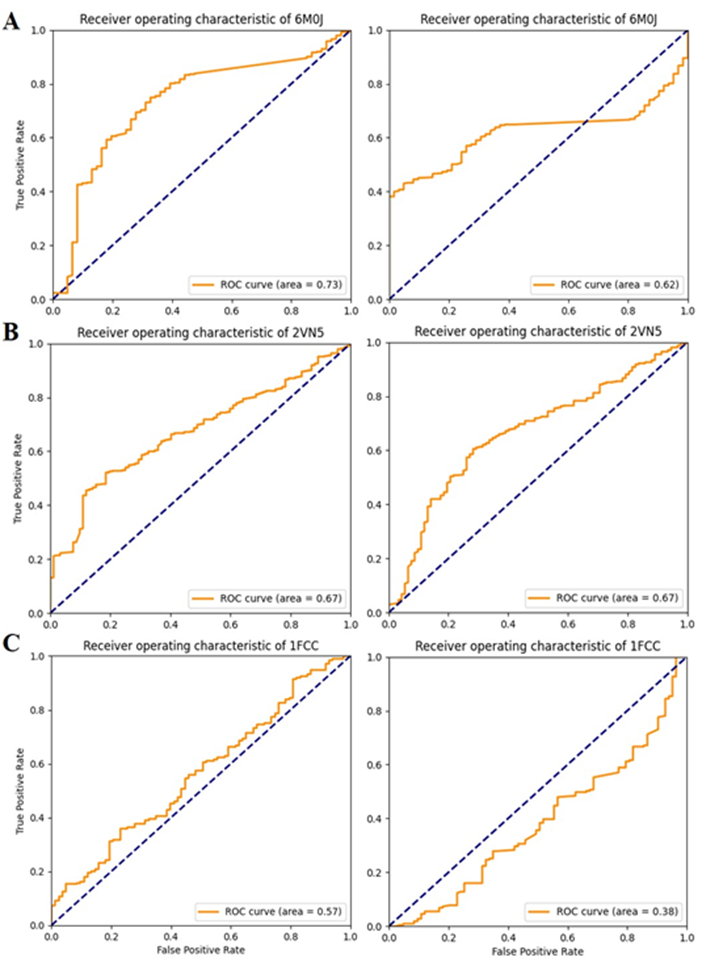
图4:ProBID-Net(左)和ProteinMPNN(右)预测与各复合物中ΔΔG的ROC曲线对比。A: C. thermocellum粘连素片段(PDB ID: 2VN5);B: IgG和IgG-Fc复合物(PDB ID: 1FCC);C: SARS-CoV-2受体结合域和ACE2复合物(PDB ID: 6M0J)
小结:在该项工作中,我们开发了一种基于深度学习的蛋白界面设计方法ProBID-Net。该方法以蛋白-蛋白界面结构为输入,通过理解蛋白-蛋白相互作用,实现界面序列的准确设计。结构域-结构域界面数据的加入增强了模型的泛化能力。在各项测试中,ProBID-Net在界面残基恢复率、困惑度、界面疏水保守性和折叠能力等方面表现出优异的性能,为蛋白-蛋白相互作用的设计提供了有效工具。ProBID-Net模型目前已开源,源代码可从https://github.com/ ComputArtCMCG/ProBID-NET获取。参考文献
【1】 ZH Chen, et
al. ProBID-Net: A Deep Learning Model for Protein-Protein Binding Interface
Design. Chemical Science, 2024. https://pubs.rsc.org/en/Content/ArticleLanding/2024/SC/D4SC02233【2】 Bryant,
P. et al. A. Improved prediction of protein-protein interactions using
AlphaFold2. Nat Commun 13, 1265 (2022). https://doi.org/10.1038/s41467-022-28865-w【3】 J.
Dauparas et al. Robust deep learning–based protein sequence design using
ProteinMPNN. Science 378,49-56(2022). https://doi.org/10.1126/science.add2187