随着“单细胞/空间转录组”技术的推广,生物/医学基础研究进入到更微观、更立体、更精准的层次。那聪明的小伙伴肯定也会想,既然有“单细胞/空间转录组”,那以此类推,是否应该有“单细胞/空间蛋白组”?嗯嗯,猜对了,这个技术确实有的。
但是,借助于“PCR”这种天才级的核酸体外扩增技术,由此衍生出的DNA/RNA相关的测序就变得迅速且高效,包括“单细胞/空间转录组”;相比于PCR技术,以“质谱”为基本逻辑的蛋白测序,就显得“低效且成本高昂”,特别是想要获取目标组织(比如1cm圆形组织)的高空间分辨率(100um级别)蛋白数据,需要分析8000个样本。这些技术瓶颈限制了“空间蛋白组”的进一步应用。
那么,由蛋白质组学国家重点实验室贺福初院士与秦伟捷研究员共同通讯,在Cell Discovery上发表的研究,利用深度学习技术,开发了一种利用计算辅助图像重建方法的空间蛋白质组学(S4P)稀疏采样策略,能将样本数量降低数千至数万倍,能极大的推广空间蛋白组的应用。
贺福初,在2001年,仅39岁当选科学院院士,40岁被授予少将军衔。主要从事蛋白质组学、精准医学和系统生物学研究。率先提出人类蛋白质组计划的科学目标与技术路线,倡导并领衔了人类第一个组织、器官的国际“肝脏蛋白质组计划”。率先提出并实践“蛋白质组学驱动的精准医学”新范式。曾获国家科技进步奖一等奖1项、二等奖3项、国家自然科学奖二等奖2项等。

下面,就和生信塔一起来看看贺福初院士和秦伟捷研究员所开发的取样策略是如何实现的!
单细胞/空间组学,更有助于我们筛选靶标分子、确定目的细胞、探讨胞间通讯等!假如小伙伴们对课题创新性要求很高,那么这些方法可以事半功倍。如果你又限于经费和样本,那么可以滴滴生信塔,从公共数据中获取原始测序数据进行二次分析,显著提高性价比!

l题目:通过深度学习促进的稀疏采样策略进行深入和高通量的空间蛋白质组学用于整个组织切片分析
研究背景
哺乳动物的器官和组织由异质性分布的细胞组成,这些细胞在空间上与细胞外基质相互作用。因此,空间解析基因表达对于确定这些细胞的功能和表型至关重要。然而,与转录组学相比,空间蛋白质组学的发展相对滞后,这主要是由于蛋白质的非可扩增特性和质谱技术的灵敏度限制。
研究思路
对厘米级小鼠脑组织样本,使用稀疏采样策略(S4P)结合深度学习模型(DeepS4P),进行空间蛋白质组学分析。首先,将组织切片成连续的10微米厚切片,并进一步微切割成平行条带,记录其空间位置。然后,通过液相色谱-质谱(LC-MS)分析每个条带。利用DeepS4P对多角度平行条带投影数据进行空间重构,生成蛋白质的空间分布图。该方法可显著减少所需样本量,从而降低质谱分析时间,实现高空间分辨率的全组织切片蛋白质组学分析。
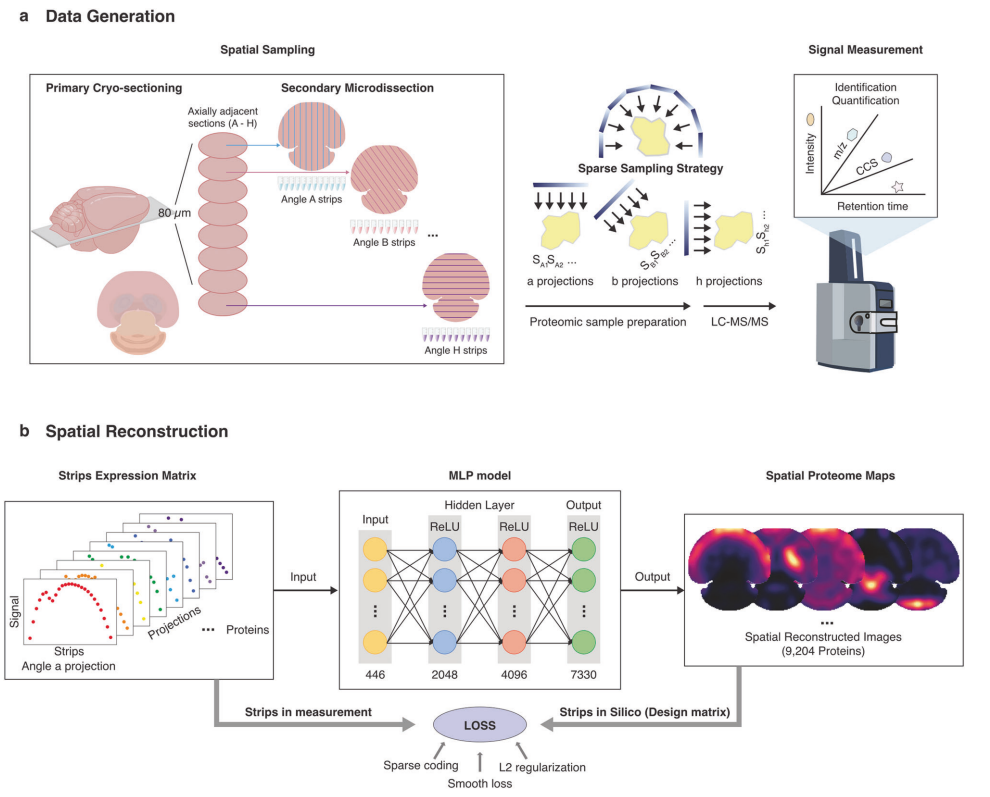
主要结果
1、 S4P策略的验证
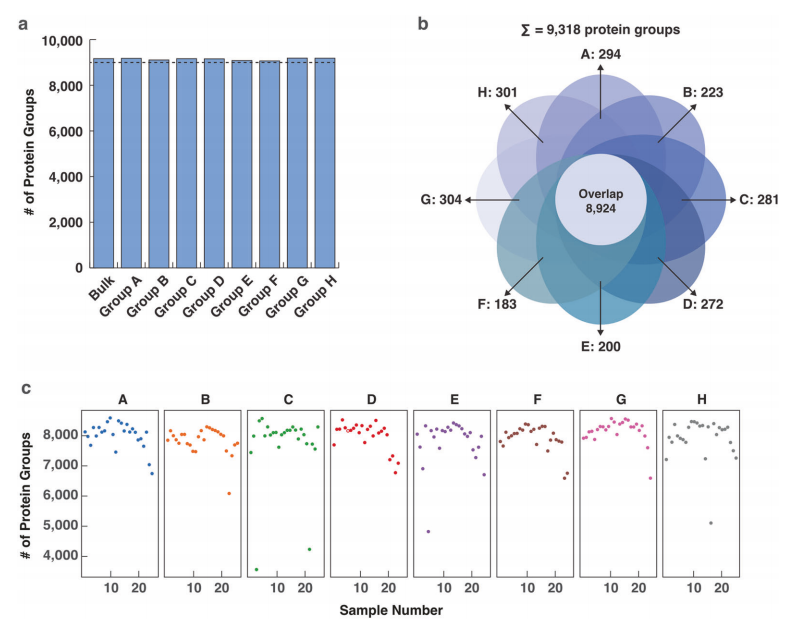
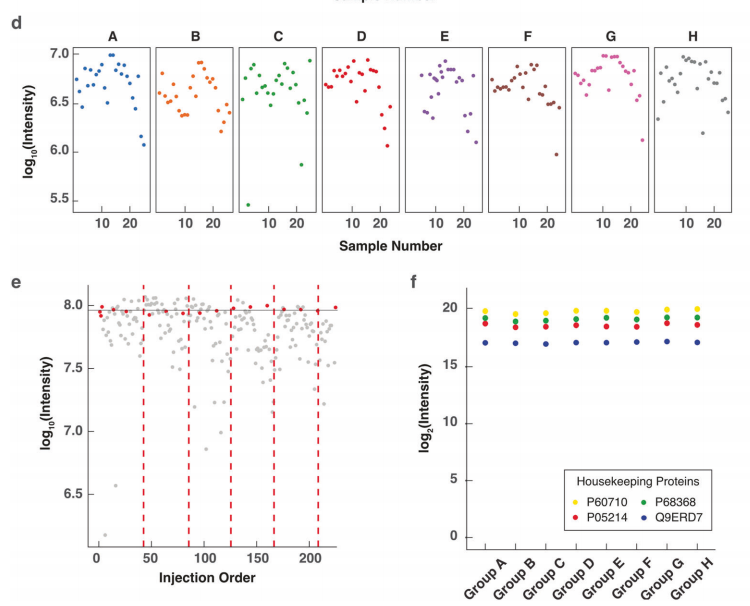
从小鼠脑中获取8个相邻的10微米厚组织切片,每个切片被微切割成平行条带,条带宽度为300微米,切割方向间隔22.5度。通过液相色谱-质谱(LC-MS)分析每个条带的蛋白质组。对同一轴向位置的条带进行蛋白质组学比较,结果显示高一致性,中位数变异系数(CV)< 0.13。通过主成分分析(PCA)和批量效应评估,验证了数据的可靠性和稳定性。在约200小时的质谱时间内,成功鉴定了234,768个肽段和9,318种独特蛋白质。每个切片鉴定的蛋白质数量高度一致(9,107–9,228),且超过96%的蛋白质在不同切片中共享。
2、小鼠脑空间蛋白组图谱的重建与评估
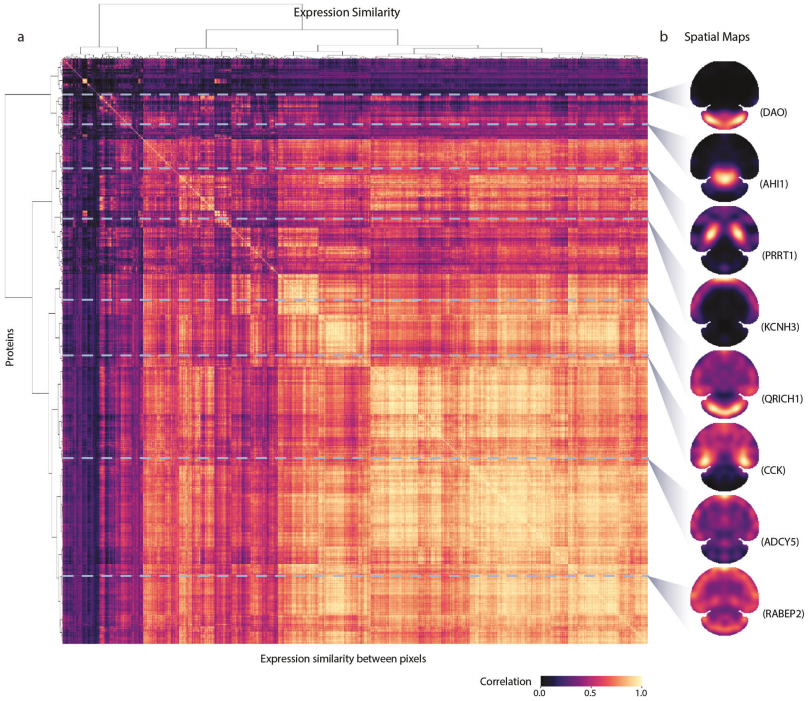
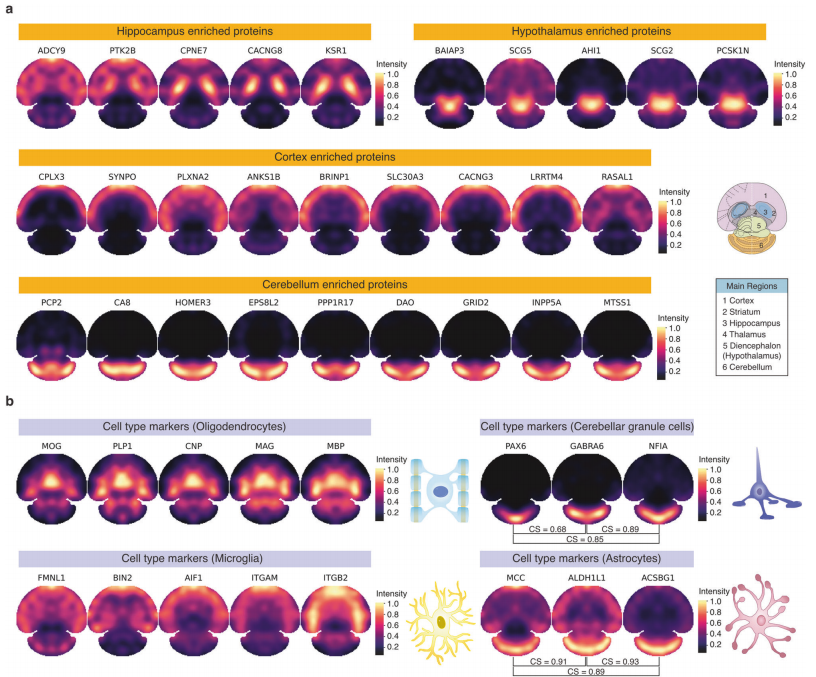
作者开发了一个前馈神经网络模型DeepS4P,用于从多角度平行条带投影数据中重建蛋白质的空间分布。模型通过最小化损失函数(包括稀疏编码、平滑损失和L2正则化)进行训练。成功重建了小鼠脑全组织切片的空间蛋白组图谱,包含9,204种蛋白质,空间分辨率为525微米。通过与免疫组化(IHC)结果对比,验证了S4P重建结果的准确性。发现了一些潜在的新脑区或细胞类型标记蛋白,如NRP1、PRRT1(海马)、LBHD2、HAP1(下丘脑)、KCTD16、SORBS2(皮层)和GRM1、ARHGEF33(小脑)。
3、小鼠脑蛋白质空间分布特征
S4P方法揭示了小鼠脑中关键生物学过程(如脑形态发生、神经递质摄取、转运和分泌、神经元-神经元突触传递等)的蛋白质空间分布。发现同一生物过程中的蛋白质在空间分布上具有一定的区域相似性,但也存在差异,这可能归因于它们在其他脑区的不同功能。例如,SV2B和SLC17A7在胶质母细胞瘤中的分布模式相似,CS值高达0.99。
4、S4P与空间转录组学(ST)的关联分析
对小鼠脑组织切片进行空间转录组学(ST)分析,并与S4P结果进行对比。共检测到8,691对基因表达产物。作者计算了转录本-蛋白质对的空间表达Pearson相关系数,发现不同分子对的相关性差异很大,范围从-0.47到0.62。正相关对主要富集在突触调节和神经元连接相关的过程;非相关对富集在细胞代谢和蛋白质翻译过程;负相关对富集在神经调节和信号传导过程。这些结果进一步证明了直接映射蛋白质数量和位置的必要性。

文章小结
综上,作者开发出的S4P方法在表征蛋白质的数量和位置方面具有显著优势,特别是在研究RNA和蛋白质空间分布不一致的复杂组织(如大脑)时。这一策略的提出,很可能会对空间蛋白组学测序有极大推动作用!如小伙伴们对课题创新性要求很高,那么这些方法可以事半功倍。如果你又限于经费和样本,那么可以滴滴生信塔,从公共数据中获取原始测序数据进行二次分析,显著提高发文性价比!
生信塔公众号持续为大家带来最新生信思路,更多创新性分析思路请点击往期推荐,快来查看吧!想复现这种思路或者定制更多创新性思路欢迎直接call生信塔,我们团队竭诚为您的科研助力!