
Basic Information
- 英文标题:A deep learning strategy to identify cell types across species from high-density extracellular recordings
- 中文标题:一种通过高密度细胞外记录识别跨物种细胞类型的深度学习策略
- 文章作者:Maxime Beau | Javier F. Medina
- 文章链接:https://www.sciencedirect.com/science/article/pii/S0092867425001102
Highlights
Para_01
Summary
Para_01
- 高密度探针允许同时从整个大脑回路中的许多神经元进行电生理记录,但无法揭示细胞类型。
- 在这里,我们开发了一种策略,可以从清醒动物的细胞外记录中识别细胞类型,并揭示具有不同功能、分子和解剖学特性的神经元的计算作用。
- 我们结合了光遗传学和药理学,以小脑作为实验模型,生成了一个精心策划的静止真值库,其中包含了浦肯野细胞、分子层中间神经元、高尔基细胞和苔藓纤维的电生理特性。
- 我们训练了一个半监督的深度学习分类器,该分类器基于记录神经元的波形、放电统计数据和层次结构,能够以大于95%的准确性预测细胞类型。
- 分类器的预测与使用不同探针、不同实验室、来自功能不同的小脑区域以及跨物种的记录上的专家分类一致。
- 我们的分类器通过揭示行为期间同时记录的细胞类型的独特贡献,扩展了现代动力系统分析的能力。
Graphical abstract
Keywords
- cerebellum; cerebellar cortex; cell-type identification; circuit mapping; Neuropixels; variational autoencoder; machine learning; classification
Introduction
Para_01
- 神经系统包含许多细胞类型,这些细胞类型由它们的分子、解剖学、形态学和生理学特性定义。
- 强大的现代分子技术已经揭示了即使在已知的解剖学细胞类别中也存在多种亚型。
- 在多个层面上识别细胞类型对于理解大脑如何工作以及开发针对脑功能障碍的选择性和靶向性疗法至关重要。
- 因此,制定策略来确定细胞类型并在分析的不同层面上交叉引用细胞类型是至关重要的。
Para_02
- 在行为过程中对神经元的动作电位进行细胞外记录是理解神经回路如何产生行为的一个重要方法。
- 因此,在特定且量化的行为期间,基于细胞外记录特征实现可靠的细胞类型识别是必要的。
- 具体而言,使用高密度多接触记录探针进行大规模的同时电生理记录16,17,结合体内细胞类型识别,将有助于表征驱动行为的电路级处理。
Para_03
- 识别细胞类型对于仅提供尖峰波形和放电统计数据的细胞外记录技术来说是一个特别困难的挑战。
- 通过测量记录的具体特征来尝试以前进行细胞外神经元识别的方法,在不同实验室中并未证明是可靠的。
- 虽然光遗传学方法在细胞类型识别方面提供了一个可行的解决方案,可以创建一个细胞外特性的库,但直接使用光遗传学进行识别无法达到足够的规模:目前光遗传学识别只在小鼠中常规使用,并且一次只能针对一种或两种细胞类型。
- 总的来说,由于必需的技术与在清醒动物中同时对多种细胞类型进行电极记录不兼容,因此通过揭示神经元的转录或解剖特性来解决细胞类型识别的挑战是不可行的。
Para_04
- 我们的目标是仅通过体外记录在清醒动物中的记录来实现细胞类型识别。
-
为此我们开发了一种可以跨实验室、探针和物种扩展的策略。
- 小脑具有晶体状结构,神经元连接定义明确,并且存在少量解剖学上定义的细胞类型,这些细胞类型在进化过程中得到了保留。
- 它拥有从最小和最密集分布(颗粒细胞)到大脑中最大的(浦肯野细胞)等多种大小的神经元,这使我们能够测试我们的记录方法的分辨率。
- 它包含许多自发放电的神经元,其中一些具有较高的自发放电率,这使我们能够严格地表征它们的电生理特性。
- 对于小脑中的所有主要细胞类型,都有可用的转基因小鼠Cre品系。
- 这使我们能够利用光遗传学策略进行基准细胞类型识别。
- 最后,小脑有着长期的神经生理学记录历史,这使我们能够将我们的测量结果和自动化的细胞类型分类与人类专家的宝贵经验进行对比。
- 在这样一个测试平台中解决细胞类型识别挑战的策略应该能为应用于其他结构(包括大脑皮层、海马和基底神经节)提供路线图。
Para_05
- 我们通过创建一个已识别的小脑细胞类型的真实库,该库记录了清醒小鼠中的小脑细胞类型,并开发了一种半监督深度学习分类器,该分类器基于波形、放电统计数据和记录的解剖层,准确预测真实库中小脑细胞类型。
- 该分类器在行为小鼠和猴子的大量由专家标记的小脑记录中,以高置信度识别细胞类型。
- 动态系统分析通过揭示在小鼠和猴子进行复杂行为时同时记录的不同实验室的已识别细胞类型的独特时间动态,提供了生物学见解。
Results
General approach
一般方法
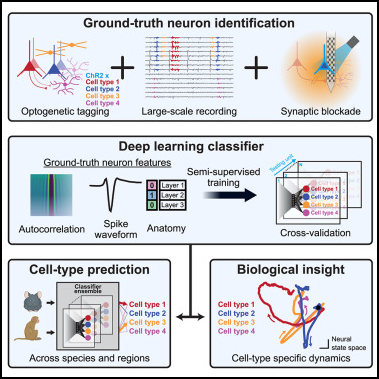
Para_01
- 我们部署了图1中概述的多步策略。(1)我们基于使用突触阻断的光遗传激活创建了一个基于基因定义的神经元的真实细胞类型库,以确认神经元被直接激活。
- (2)我们确定了可以用于在真实细胞类型库上训练半监督深度学习分类器的电生理记录特征。
- (3)我们通过预测来自小鼠和猴子的专家分类记录的独立数据集中的细胞类型来测试分类器的普适性。
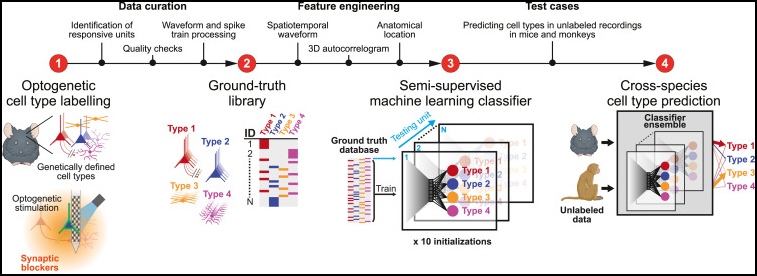
图片说明
◉ 图1。从神经回路的细胞外记录中识别细胞类型的策略。详见正文中。
Multi-contact probe recordings and data curation
多点接触探针记录和数据整理
Para_01
- 在小脑皮层中,形态各异的细胞类型分布在不同的层(图2A)。
- 分子层中间神经元(MLI)分布在整个分子层中,包括投射到浦肯野细胞胞体的篮状细胞和投射到浦肯野细胞树突的星形细胞。
- 颗粒细胞层包含苔藓纤维终末、高尔基细胞和颗粒细胞。
- 其他较少见的细胞类型存在于不同的层中,但因为有可用的Cre品系可以在那些细胞类型中表达感光蛋白,我们专注于小脑回路的主要细胞类型(图2A)。
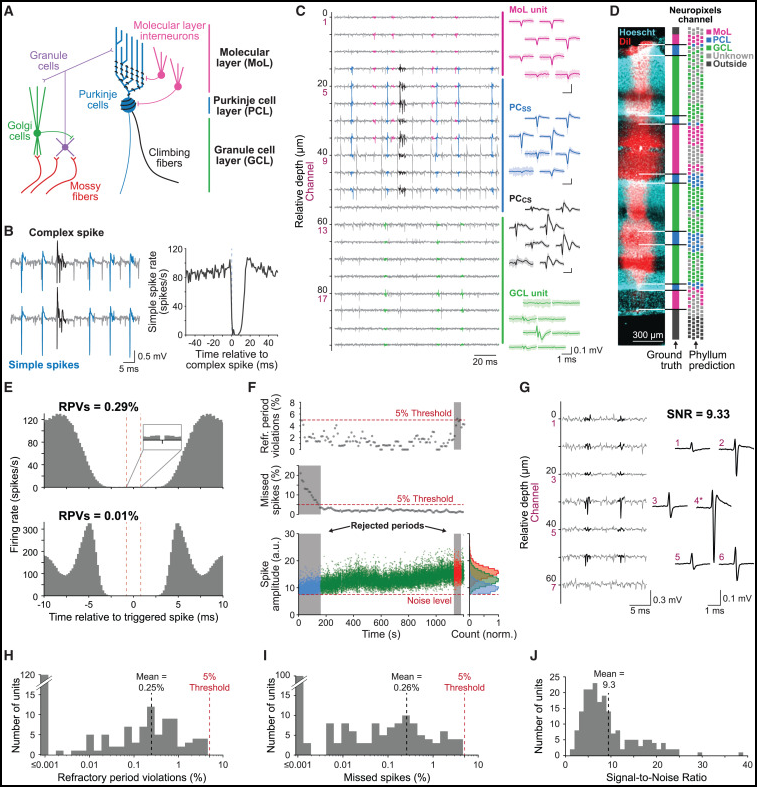
图片说明
◉ 图2。小鼠小脑皮层Neuropixels记录的整理(A) 典型的小脑电路示意图。(B) 浦肯野细胞中的简单尖峰(浅蓝色)和复杂尖峰(黑色)。右侧的交叉相关图记录了复杂尖峰触发的简单尖峰暂停。(C) 来自Neuropixels探针20个通道的示例记录。轨迹突出显示分子层中记录的单个单元(洋红色),浦肯野细胞的简单尖峰(蓝色),同一浦肯野细胞的复杂尖峰(黑色),以及颗粒层中记录的单元(绿色)。(D) 使用DiI和Hoechst标记的示例组织学与Phyllum根据电记录预测的层次结构进行比较。Neuropixels示意图上的不同颜色表示Phyllum预测的层次。(E) 两个单元的典型自相关图,具有很少的不应期违规(RPVs)。(F) 在记录过程中隔离质量随时间变化的分析。从上到下,图表显示了不应期违规百分比、估计的丢失尖峰百分比和尖峰幅度。水平虚线表示接受阈值。灰色区域表示被排除在分析之外的时段。符号颜色表示来自间隔的尖峰,该间隔丢失的尖峰过多(蓝色),隔离可接受(绿色),不应期违规过多(红色)。右侧边缘直方图显示了尖峰幅度分布,以证明在蓝色直方图中噪声水平处的截断,这将导致拒绝时间间隔。(G) 信号与噪声比(SNR)为9.33的代表性记录示例的记录轨迹和空间足迹。波形编号根据其通道。星号(∗)表示具有最大峰-谷幅度的通道,用于计算SNR。(H) 被接受到真实库中的神经元的不应期违规百分比分布。(I) 被接受到真实库中的神经元的估计丢失尖峰百分比分布。(J) 被接受到真实库中的神经元的具有最大振幅波形的通道上的SNR分布。另见图S1。◉ ,
Para_01
- 浦肯野细胞仅通过其细胞外电生理信号就可以进行真实标签的识别(图2B,左)。
- 它们具有‘简单尖峰’,以高频率发射,并且具有‘复合尖峰’,由其攀爬纤维输入驱动,在大约1 Hz的频率下发生,并触发简单尖峰中一个特征性的10-50毫秒暂停。
- 因此,如果浦肯野细胞在其简单尖峰发放的复合尖峰触发直方图中显示出暂停,则可以将其纳入真实标签库(图2B,右)。
Para_02
- 带有 Neuropixels 探针的记录在 384 个通道中的许多通道上检测到神经活动,并且聚类分析产生了许多单元。
- 图2C中的品红色波形来自分子层中的一个神经元,该神经元可能是一个MLI候选者。
- 绿色波形来自颗粒细胞层中记录的一个神经元,可能是苔藓纤维、高尔基细胞或颗粒细胞。
- 蓝色和黑色波形是识别出的浦肯野细胞的简单和复杂尖峰。
- 我们在图2B和2C所示的记录中突出显示了振幅最大的单元,但我们还对较小振幅的单元进行了聚类,并将其纳入我们的分析和筛选流程,详情如下。
Para_03
- 我们分析流程的第一步是对每个通道所在的层进行客观识别。
-
如图2D所示,典型的记录轨迹通过了3个分子层、5个浦肯野细胞层和3个颗粒细胞层。
- 我们使用Phyllum(一种用于分析探针上各通道记录的Phy插件)来确定每个通道对应的层(参见STAR方法)。
- Phyllum推断出的层结构与基于探针的DiI标记轨迹与核染色之间对应关系的组织学层识别结果吻合良好(图2D)。
- 我们在21次经组织学确认的穿透实验中验证了Phyllum的准确性,在分子层、浦肯野细胞层和颗粒细胞层的776、367和1140个记录位点中,其结论分别与组织学结果一致的比例分别为99%、95%和98%。
我们分析了每个孤立神经元假设的0.8毫秒不应期,以评估来自其他神经元或噪声的潜在污染程度。图2E中的示例自相关图显示出较少的不应期违规现象,分别代表了数据集中的平均值(0.25%)和中位数(0.01%)。我们拒绝了超过5%不应期违规的单元(图2F,红色符号和直方图),并且几乎所有被接受的神经元的不应期违规率都低于1%(图2H)。结合平均放电率,不应期违规可以用来估计一个神经元的脉冲中可能受到污染的比例,我们在论文稍后部分在识别出细胞类型之后提供了这一分析。
我们通过用高斯函数拟合尖峰幅度分布,并量化曲线下面积被噪声阈值裁剪的部分来估计遗漏的尖峰数量(图2F)。在图2F中,我们排除了大约前150秒的记录,因为我们估计超过5%的尖峰被遗漏了(蓝色符号和直方图)。在我们接受的记录中,遗漏的尖峰平均占比0.26%,几乎所有神经元的遗漏尖峰比例都低于1%(图2I)。
Para_04
- 作为细胞类型识别的重要输入,严格的准则对于确保可靠的细胞类型识别至关重要。
- 除了上述描述的质量标准外,我们采取了措施以确保外部数据的一致性和适当预处理(图S1),并优化了个别动作电位的时间对齐(见STAR方法)。
- 少数违反不应期要求和少量未检测到的动作电位的要求确保了我们接受的单元具有较高的信噪比。
- 在信号幅度最大的通道上,我们接受样本的平均信噪比为9.3(几乎与图2G中的示例记录相同)。
- 我们的标准旨在明确排除任何可能来自不同神经元、噪声或代表两个或多个神经元叠加的动作电位。
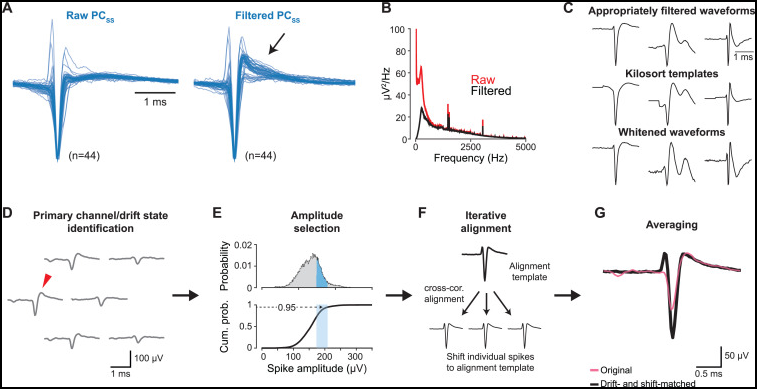
图片说明
◉ 图 S1. 与图 2 相关的神经波形预处理分析流程◉ 当我们比较了不同实验室的记录,并理解了不同的分析流程的独特之处后,我们了解到需要在不同准备之间协调数据,并确保我们的流程输出能够提供对实际波形的最佳估计。◉ 仔细的预处理可以减少每个细胞类型内部的波形变化,可能还提高了分类器区分细胞类型的性能。◉ 我们在这里详细说明了那些程序——一致过滤和漂移匹配。◉ (A) 来自 44 个通过复杂峰引起的简单峰放电暂停确定的浦肯野细胞的简单峰波形。◉ 左侧的轨迹显示了使用硬件滤波器禁用的 Neuropixels 记录主要通道的归一化电压对时间轨迹。◉ 右侧的轨迹展示了在应用了一个等同于 Neuropixels 探针上的板载硬件滤波器(单极 300 Hz 高通巴特沃斯)的软件滤波器后的相同波形。◉ 黑色箭头指出了滤波前后波形形状的主要差异。◉ 通过应用一个软件版本的硬件滤波器来统一跨数据集波形的程序是至关重要的。◉ (B) 表示从 (A) 中的原始(红色)和滤波(黑色)波形在主要通道上低频带功率的平均。◉ 这验证了板载硬件滤波器的影响。◉ (C) 显示三个小脑神经元的波形对比,展示标准工具如何导致 Neuropixels 探针使用的波形出现偏差。◉ 顶部一行展示了使用我们研究开发的分析流程得到的 3 个单元的最佳波形重建。◉ 中间一行展示了 Kilosort 创建的模板,这些模板相对于我们流程识别的最佳波形可能是相当扭曲的。◉ 底部一行展示了 Kilosort 的零相位分量分析(ZCA)白化结果,这是一种与执行尖峰排序相关的处理过程,但由于 ZCA 白化中邻近通道活动的作用,这种过程可能会严重扭曲波形。◉ 标准分析流程导致的一些波形失真突显了我们改进的效果,从而提供了高质量分类器输入。◉ (D) 使用漂移匹配算法进行高质量神经波形识别的预处理流程的第一步:Z 漂移匹配——通过最大峰峰值幅度(红箭头)识别主通道。◉ (E) 第二步,X-Y 漂移匹配:在识别的主通道上选择神经波形子集,顶部图表显示峰峰值幅度分布,底部图表描绘累积概率分布。◉ 选择了 N(用户可配置)个峰峰值幅度低于第 95 百分位数的动作电位波形(蓝色阴影区域),消除了第 95 至 100 百分位数之间的尖峰,以减轻潜在的大振幅伪影。
◉ (F) 第三步,位移匹配:通过交叉相关峰值与对齐模板(根据振幅选择的最大峰峰值波形的平均值)连续/迭代地对齐小批次波形。◉ (G) 主通道上对齐波形的最终平均值。◉ 黑曲线表示经过完整的漂移位移管道后的波形模板,而红线表示从 (D) 复制的原始平均波形。◉ 红线和黑线之间的差异展示了我们分析流程的影响。
Combination of optogenetics and pharmacology for ground-truth cell-type identification
光遗传学与药理学结合用于真实细胞类型识别
Para_01
- 为了通过光刺激识别细胞类型,我们在表达感光蛋白(通常是通道视紫红质-2,ChR2,在一些抑制性感光蛋白用于GABA能中间神经元;详见STAR方法)的特定细胞类型小鼠小脑中插入了Neuropixels探针。
- 我们在多个阶段进行了光遗传学刺激,包括在药理学应用突触阻断剂之前、期间和之后(图3A):基线、对照、灌注和阻断(详情见STAR方法)。
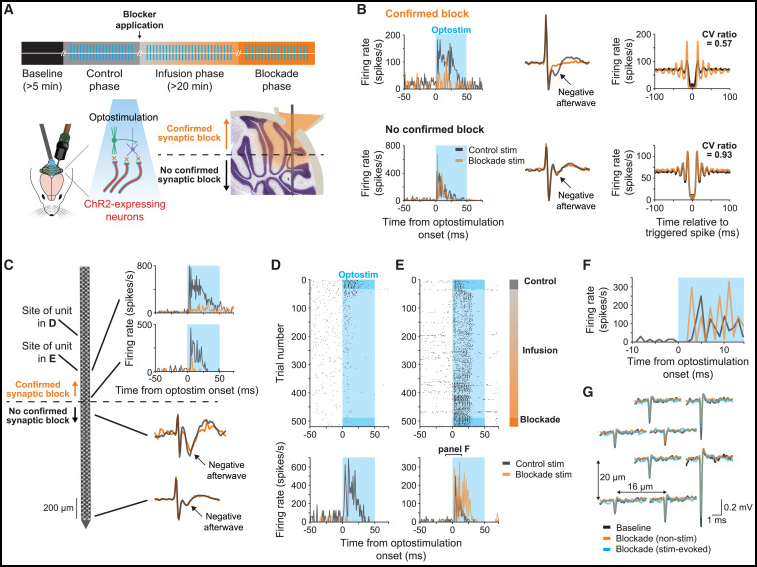
图片说明
◉ 图3 细胞类型真实性的识别策略(A) 显示了在突触阻断存在的情况下测试光遗传激活实验的各个阶段的示意图。(B) 来自6个不同单元的例子,展示了确定有效突触阻断区域所使用的标志。从左到右,每行中的示例单元展示了阻断对光遗传刺激反应的影响、苔藓纤维波形的负后波,以及由自相关图和CV值定义的放电统计。(C) 展示我们如何确定记录是否在突触阻断区域内。沿着Neuropixels探针,右侧顶部的直方图显示了位于阻断区域内的位点,因为在那里记录的单元失去了对光遗传刺激的响应,而较低的波形显示了位于阻断区域下方的苔藓纤维,因为它们保留了其负后波。(D) 在突触阻断期间失去对光遗传刺激反应的神经元的散点图和刺激后时间直方图。黑色与橙色直方图分别显示了突触阻断前后的反应。蓝色阴影表示光刺激的时间。(E) 与(D)相同,但展示了一个在突触阻断期间保持对光遗传刺激反应的神经元。(F) 快速时间尺度记录展示了(E)中神经元在控制阶段和阻断阶段对光遗传刺激的短潜伏期反应。(G) (D)中神经元的空间足迹。黑色、橙色和蓝色轨迹显示了在基线期、无光遗传刺激的突触阻断期和有光遗传刺激的突触阻断期记录的波形。波形根据其接触的相对位置进行排列,两个双头箭头指示了Neuropixels探针上接触点的水平和垂直间距。另见图S2和S3。
Para_01
- 我们只接受那些在光刺激下被直接激活的神经元,前提是它们位于突触阻断区域之内,并且能够对光产生可靠且潜伏期短(反应潜伏期<10 ms,图S2A)的响应。
- 相反,如果无法确定事实真相,例如因为神经元对光遗传学刺激的反应潜伏期较长(≥10 ms),或者在存在突触阻断剂的情况下失去了响应,或者我们无法确认在记录该神经元的位置上突触阻断是有效的,那么我们会排除这些神经元。
- 在特定记录深度上认为突触阻断有效,只有当我们能够记录到在这个深度或更深的神经元显示出图3B顶部一行所示的三种迹象之一:
- 在控制阶段存在的光遗传学刺激响应,在阻断阶段消失(图3B,顶部一行,左侧)。
- 假定的苔藓纤维在阻断阶段负后波消失(图3B,顶部一行,中间)。先前证据表明,负后波是颗粒细胞的突触后反应。45,46
- 神经元自相关图或变异系数(CV)发生显著变化,通常是由于从由突触和内在驱动的放电转变为纯粹的内在生成放电而导致的规律性增加(图3B,顶部一行,右侧)。
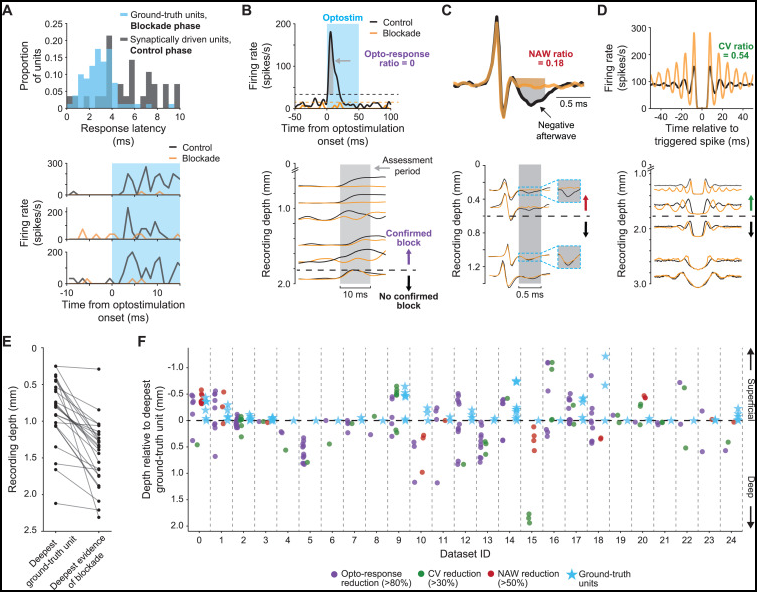
图片说明
◉ 图 S2。突触阻断的定量评估,与图 3 相关。对于每个至少记录到一个真实神经元的记录,我们使用三种定量标准分析了所有记录到的神经元,以确定确认的突触阻断区域。◉ 标准包括突触阻断对(i)光遗传刺激响应大小的影响(B),(ii)苔藓纤维波形上的负后波大小的影响(C),以及(iii)放电率变异系数的影响(D)。如果在 Neuropixels 探针上任意位置发现了三种有效突触阻断迹象中的至少两种,则认为该记录可能包含真实神经元。◉ 分析证实,真实神经元库中的每个神经元都在或高于确认突触阻断的最深深度记录位点(E 和 F)。我们得出结论,真实神经元库中的所有神经元很可能被光遗传刺激直接激活,而不是通过突触输入间接激活。(A)光遗传反应潜伏期分布。◉ 蓝色和灰色条形表示真实神经元库中神经元的潜伏期与由于在突触阻断期间失去对光遗传刺激的响应而被认为通过突触激活的神经元的潜伏期。◉ (B)上部面板显示了一个示例神经元的响应,说明我们如何量化突触阻断对光遗传刺激响应的影响。黑色和橙色轨迹显示控制阶段与阻断阶段的数据,蓝色阴影表示光遗传刺激的时间,灰色阴影表示用于测量曲线下面积的间隔。"光遗传响应比"定义为阻断后测量间隔内的曲线下面积除以前阻断的曲线下面积。水平虚线表示光遗传响应的统计学确定基线。◉ 下部面板显示了单个 Neuropixels 探针记录的多个神经元的响应,说明了许多在确认突触阻断区域内失去对光遗传刺激响应的神经元。水平虚线标记了突触阻断的最深证据线,在这里放置在记录到一个在阻断阶段失去对光遗传刺激响应的神经元的最深处。◉ 我们将光遗传响应比小于等于 0.2 视为突触阻断的证据。(C)上部面板显示了突触阻断如何改变一个示例神经元的自相关图和变异系数。黑色和橙色自相关图显示了控制阶段与阻断阶段的结果。"变异系数比"定义为阻断后的变异系数除以前阻断的变异系数。◉ 下部面板显示了突触阻断对位于水平虚线之上或之下的多个神经元自相关图的影响。水平虚线指示了确认突触阻断的最深部位。记录在虚线上方的神经元在突触阻断后表现出放电率的规律化,而记录在虚线下方的神经元的自相关图几乎没有变化。◉ 我们将变异系数比小于等于 0.7 视为突触阻断的证据。(D)上部面板显示了突触阻断如何改变一个示例神经元的自相关图和变异系数。黑色和橙色自相关图显示了控制阶段与阻断阶段的结果。"变异系数比"定义为阻断后的变异系数除以前阻断的变异系数。◉ 下部面板显示了突触阻断对位于水平虚线之上或之下的多个神经元自相关图的影响。水平虚线指示了确认突触阻断的最深部位。记录在虚线上方的神经元在突触阻断后表现出放电率的规律化,而记录在虚线下方的神经元的自相关图几乎没有变化。◉ 我们将变异系数比小于等于 0.7 视为突触阻断的证据。(E)图表绘制了所有 24 个至少记录到一个真实神经元的记录的最深真实神经元深度和最深突触阻断证据。◉ 每条线对应一次单独的记录。所有线的负斜率或零斜率表明所有真实神经元都在定量验证的突触阻断区域内。◉ (F)散点图显示了满足有效突触阻断标准之一的每个神经元的深度,对于所有 24 个至少记录到一个真实神经元的记录。青色星号表示真实神经元,红色符号表示突触阻断导致负后波幅度减少至少 50% 的苔藓纤维,绿色符号表示突触阻断导致变异系数减少至少 30% 的神经元,紫色符号表示突触阻断导致光遗传刺激响应大小减少至少 80% 的神经元。◉ 所有深度相对于实验中记录到的最深真实神经元的深度绘制。在所有情况下,至少有一个神经元在最深真实神经元的深度或更深的深度满足突触阻断的标准。
Para_01
- 为了说明我们的策略,我们展示了一个转基因小鼠实验的例子,该小鼠品系(Thy1-ChR2品系18)中的苔藓纤维表达了ChR2蛋白。
- 图3C显示了六个神经元的探针位置,因为它们在对照阶段被光激活且潜伏期较短,所以被认为是真正的苔藓纤维的良好候选者。
- 由于在阻断阶段失去了对光遗传刺激的反应,我们从真实的苔藓纤维库中排除了其中三个神经元(图3C和3D)。
- 其中两个神经元具有潜在的苔藓纤维波形(图3C),但是因为无法在其位置确认突触阻断,我们也排除了它们:它们在阻断阶段保留了负后波。
- 只有这六个神经元中的一个满足了所有必要的条件,可以被分类为真实的苔藓纤维:它在对照阶段和阻断阶段都对光遗传刺激产生了短暂潜伏期的响应(图3E和3F),
- 并且它位于有效突触阻断区域,这一点通过在同一位置或其下方记录的两个神经元在阻断阶段失去了光刺激响应得到证实(图3C)。
- 该神经元在整个实验过程中外细胞波形保持不变(图3G),表明记录稳定。
- 我们在图S2B-S2F中展示了对所有记录的突触阻断的定量分析。
Para_02
- 我们使用的一些小鼠品系不如Thy1-ChR2品系在图3C-3G例子中那么特异,这种复杂性可能导致多个细胞类型的直接光激活,并导致一些记录到的神经元误识别。
- 图S3展示了我们如何结合基于Phyllum的记录层鉴定和光遗传学激活来明确确定细胞类型,即使是在我们选择用于标记高尔基细胞的GlyT2-Cre品系中,这个问题尤为突出,该品系存在非目标表达的问题。
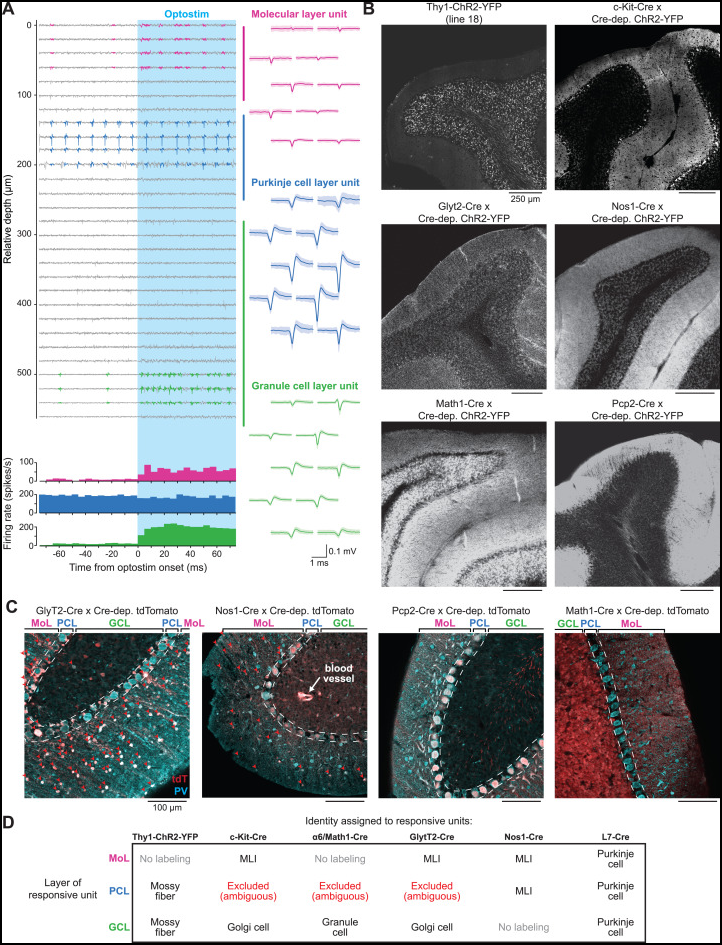
图片说明
◉ 图 S3。量化我们研究中使用的鼠系潜在脱靶感光蛋白表达及策略来减轻其影响,与图 3 相关 脱靶标记是我们使用的一些鼠系的一个重要属性。◉ 因此,我们不能简单地假定一个通过光遗传学激活的神经元是该系之前描述的标记细胞类型。◉ 此图描述了一种基于(i)对我们使用的所有鼠系进行细致的组织学评估和(ii)通过Phyllum识别记录层的策略来管理脱靶标记问题的方法。◉ 脱靶表达的问题在之前用于成像高尔基细胞活动的GlyT2-Cre系中最为明显。◉ (C)显示GlyT2-Cre系在分子层中间神经元中有显著的脱靶表达,并且非常偶尔在浦肯野细胞中有脱靶表达;分子层中间神经元的相对密度高于高尔基细胞(79比20%)。◉ 因此,我们在颗粒层和分子层都记录了对光遗传学刺激直接响应的神经元(A底部的PSTH)。
◉ 我们使用Phyllum来识别记录层和颗粒层中被光遗传学刺激直接激活的标记单元作为高尔基细胞,以及分子层中的标记激活单元作为分子层中间神经元。◉ 此图中剩余的组织学显示,其他鼠系也表现出一些但不那么明显的脱靶表达。◉ 例如,用于标记颗粒细胞的Math1-Cre系总体上是特异性的,但偶尔标记浦肯野细胞。◉ c-kit-Cre系也标记少量的高尔基细胞,Nos1-Cre系除了标记分子层中间神经元外,还偶尔标记非神经元细胞。◉ 相比之下,我们使用的其他系更为干净,如用于标记苔藓纤维的Thy1-ChR2-YFP系18和用于标记浦肯野细胞的Pcp2-Cre系。◉ 至关重要的是,在我们的任何一条系中,我们都没有观察到单个小脑层内的多个标记细胞类型。◉ 因此,已识别层与直接光遗传学激活的结合使我们能够在所有实验中区分细胞类型。◉ (A) GlyT2-Cre系中一个示例光标签实验,显示Neuropixels探针通道上的原始电压轨迹。◉ 右侧洋红色、蓝色和绿色波形显示了分子层(MoL)、浦肯野细胞层(PCL)和颗粒层(GCL)中神经元的空间足迹。◉ 电压轨迹下方的直方图显示,分子层和颗粒层的神经元都被光遗传学刺激激活(青色阴影)。◉ (B) 组织学显示ChR2-YFP融合蛋白在我们鼠系中的定位。◉ 第一行显示,在用于识别苔藓纤维的Thy1-ChR2系和用于识别分子层中间神经元的一个实验室的c-kit-Cre系中,脱靶表达最少。◉ 第二行显示,在用于识别高尔基细胞的一个实验室的GlyT2-Cre系中,分子层中间神经元有显著的脱靶表达,以及Nos1系在MoL中的密集标记,我们用它来识别分子层中间神经元。◉ 第三行显示,在用于尝试识别颗粒细胞的Math1-Cre系和用于识别一些真值浦肯野细胞的L7-Cre系中,脱靶表达很少。◉ (C) 来自Cre系的小脑切片,与tdTomato报告基因小鼠杂交,免疫染色以标记Cre阳性细胞(红色)和parvalbumin(PV;青色),一种标记浦肯野细胞和分子层中间神经元的标志物。◉ 左图表明,GlyT2-Cre系驱动在GCL中的高尔基细胞和MoL中的分子层中间神经元中的表达。◉ 红色箭头表示双重标记的细胞。◉ 剩余的图显示了Nos1-Cre、Pcp2-Cre和Math1-Cre系的代表性切片。◉ (D) 表格概述了我们如何利用层信息来克服某些Cre系中的脱靶表达问题。◉ 即使对于脱靶表达最少的Cre系,如c-kit-Cre系,我们也使用层信息作为合理性检查。
The ground-truth library
真实数据图书馆
Para_01
- 在两个实验室的188次Neuropixels记录中,我们总共记录了3,652个通过尖峰排序和整理管道存活的神经元(图4A)。
- 大多数未能达到纳入地面真值库的标准(详情见图例):只有97个在突触阻断存在的情况下保留了反应(例如,图3E,在图3F中的快速时间基记录;延迟分布见图S2A)。
- 另外28个神经元在突触阻断期间失去了反应,因此被间接的、突触激活驱动,具有较短的潜伏期(<10毫秒)(例如,图3B顶部:5毫秒,图3C中的两个神经元:3毫秒,图3D中的两个神经元:2毫秒;延迟分布见图S2A)。
- 在97个直接激活的神经元基础上,我们增加了由简单尖峰触发的暂停识别出的62个浦肯野细胞的简单尖峰和复杂尖峰,并去除了5个未通过最终客观质量检查的单元以及14个层分配不明确的单元。
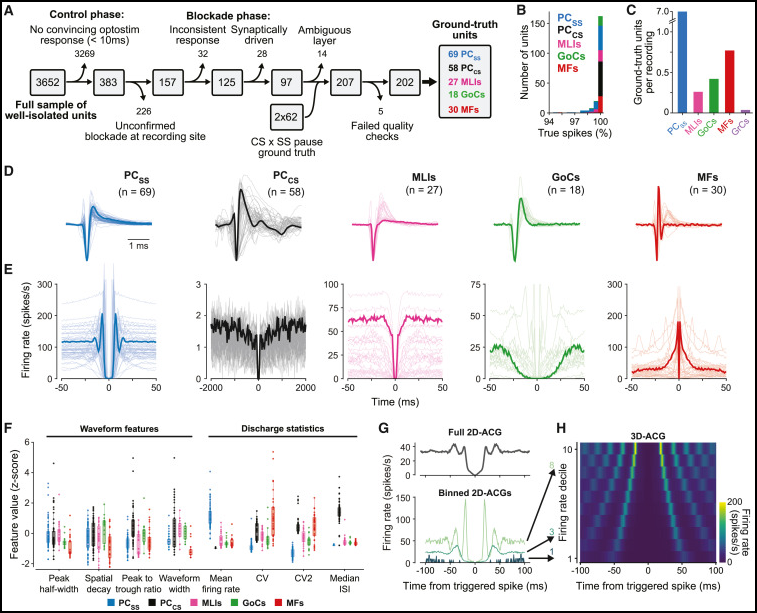
图片说明
◉ 图4 皮质细胞类型真实库的选择标准和特性 (A) 创建真实库的整理过程,包括每个整理阶段保留或删除的数量。◉ (B) 根据颜色按细胞类型分隔的实际属于给定神经元的尖峰百分比估计分布。有关尖峰百分比真实性的计算,请参阅STAR方法。缩写词为PCSS:浦肯野细胞简单尖峰;PCCS:浦肯野细胞复杂尖峰;MLIs:分子层中间神经元;GoCs:高尔基细胞;MFs:苔藓纤维;GrCs:颗粒细胞。◉ (C) 显示每种细胞类型的地面实况单元数量的直方图,该数量归一化为记录次数。缩写词同(B)。◉ (D) 地面实况库中每种细胞类型的叠加波形。缩写词同(B)。粗线表示图S7C中的3D-ACG示例中的神经元。波形幅度归一化并翻转,以确保最大的峰值为负。注意翻转导致一些苔藓纤维(MFs)具有正向偏移的负后波(如图3B所示)。其他MFs根本不显示负后波,可能是因为探头接触点与MF之间的相对位置变化。通常,负后波只出现在一个接触点上,这可能是由于认为负后波反映了突触后颗粒细胞场电位,因此应该只出现在更靠近glomerulus的通道上。另外请注意,分子层中间神经元有两种不同的波形。◉ (E) 同(D),但显示地面实况神经元的自相关图。◉ (F) 传统波形或放电统计测量无法区分细胞类型。每个符号显示来自单个神经元的不同特征的Z得分值,不同颜色表示不同的细胞类型,见图底部的图例。Z得分是针对每个特征单独计算的,但在每个特征内跨细胞类型计算。缩写词同(B)。◉ (G) 不考虑发射率的二维ACG(上图)和根据瞬时发射率分离尖峰的二维ACG(下图)。不考虑发射率创建的二维ACG(上图)在不规则间隔处有多肩部,而为不同平均发射率创建的三个ACG(下图)则更规则。◉ (H) 从G中的二维ACG创建的三维ACG,用于归一化发射率变化。箭头显示每个二维ACG的目的地。三维ACG绘制了包含10个不同均发射率十分位数的二维ACG的10行。另见图S4。
Para_01
- 神经元位于真实情况库中(图4A右侧),它们都被很好地分离。
- 它们超过95%的峰值未受污染(图4B),并且几乎所有的峰值都接近100%,这是STAR方法中详细说明的指标。
- 为了与之前的报告进行比较19,49,50,51,52,53,表S1使用一系列指标报告了我们真实情况库中不同细胞类型的电生理特性。
Para_02
- 神经元真实库中单位的比例偏向于浦肯野细胞(图4C),我们比较了该分布与表S2中更好的记录概率估计。
- 即使我们在表达颗粒细胞光敏蛋白的小鼠中进行了82次记录,我们从颗粒细胞中进行常规记录的成功率也非常低(参见STAR方法)。
- 确实,我们记录到了多个可能的颗粒细胞单元,这些单元在确认的突触阻断区域对光刺激有响应,但几乎所有单元都未能满足我们的好隔离标准中的一个或多个。
- 经过筛选后,我们从82次记录中保留了3个单元(每次记录约0.04个颗粒细胞)。
- 我们的样本量太小,无法将它们纳入接下来我们要开发的分类器中。
- 颗粒细胞难以常规记录可能是多种因素共同作用的结果:它们相对较小的尺寸,54,55它们细胞外电位的空间限制性封闭场,以及Neuropixels16(150 kOhm)的低电极阻抗49。
Para_03
- 我们使用了一个真实数据集,接下来基于跨细胞类型的电生理特征的一致差异,开发了一种准确的分类方法。
Waveform
波形
Para_04
-
体外的膜片钳记录证实了我们的预期,即不同细胞类型的不同的生物物理特性和形态会导致不同的波形(图S4)。
- 然而,在体外膜片钳记录中(图S4C),标准化后的波形在体内的跨细胞记录(图4D)中具有更加多变的形状。
- 我们认为体内外记录的波形变化更大是因为体外记录是在高度均匀的条件下进行的,电极直接接触细胞膜。
- 相比之下,体内记录中的距离从记录接触点到神经元、电场相对于探针的方向以及背景噪声都有所不同。
- 因此,基于体外记录波形训练的细胞类型分类器无法对体内真实情况下的神经元进行分类(图S4F)。
- 这意味着我们不能用体外记录的波形来补充我们的真实情况库。
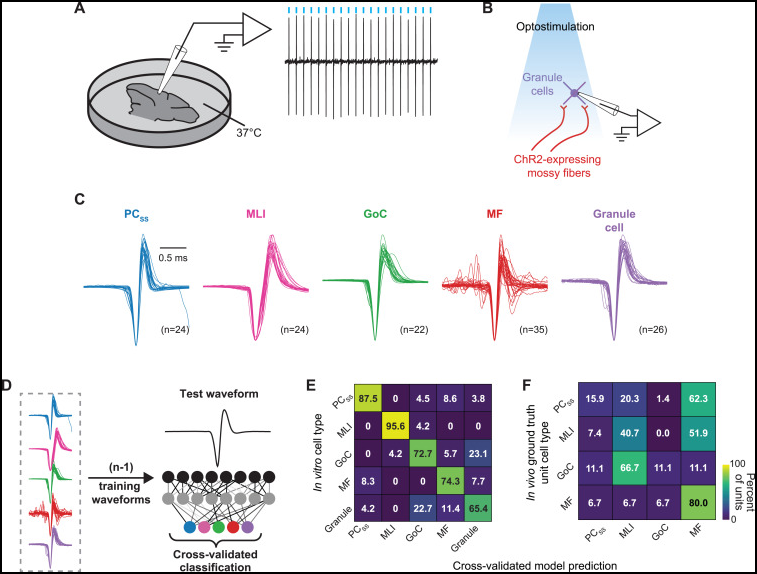
图片说明
◉ 图S4。通过体外记录证明波形是细胞类型的信息指标,与图4相关。◉ 我们受到图4D的印象,即从体外记录中提取的波形是细胞类型的一个有用指标,因此进行了更多的实验来测试波形在不同类型的基准数据中的信息性。◉ 我们在37°C下对小鼠小脑切片进行了细胞附着膜片钳记录。◉ 我们使用Thy1-ChR2系18的光遗传学方法识别苔藓纤维,并在记录过程中通过显微镜可视化观察其他细胞类型。◉ 动作电位相关的电流在细胞类型内部非常均匀,并且在细胞类型之间明显不同。◉ 与多接触探针在体内用多点探针记录的细胞外场相比,在体外记录的波形更均匀并不令人惊讶。◉ 许多未受控制的因素会影响精确的细胞外记录波形。◉ 此外,记录技术和准备方式的不同使得无法将体外记录的波形与图4D中的波形进行比较,但不同的细胞类型具有可区分的波形这一原理仍然成立。◉ 我们通过创建一个深度学习分类器并通过"留一法"策略验证其性能,验证了体外数据波形的信息性。◉ 分类器的整体准确率为78%,而随机性能预期为20%。◉ 我们得出结论,我们可以使用细胞外波形作为主要特征之一来分类细胞类型是有生理机制原因的。◉ (F)显示了根据体外波形训练的分类器在识别体内基准神经元波形的细胞类型方面的表现不佳。◉ 我们得出结论,我们必须使用体内细胞外记录的波形来创建一个基于细胞外记录的体内细胞类型分类器。◉ (A)切片记录示意图。◉ (B)在细胞附着膜片钳记录期间光遗传学刺激苔藓纤维的示意图。◉ (C)来自已识别细胞类型的叠加波形,不同颜色表示不同细胞类型:PCSS,浦肯野细胞简单尖峰;MLI,分子层中间神经元;GoC,高尔基细胞;MF,苔藓纤维。◉ (D)我们训练了一个机器学习分类器,该分类器基于波形预测细胞类型。◉ (E)混淆矩阵显示了分类器在左出测试细胞类型上的表现。矩阵条目中的数字表示给定基准类型(y轴)的细胞作为分类器在x轴上预测的函数的比例。对角线上的百分比最高,意味着分类器准确:分类器的整体准确率为78%,而随机性能预期为20%。◉ (F)混淆矩阵显示了根据体外波形训练的分类器在识别我们基准库中波形的细胞类型方面的不良表现,证实了体外数据的波形不能用于从多点探针记录的体内细胞外记录中识别细胞类型。
Discharge statistics
discharge 统计数据
Para_04
- 在真实情况库中,通过自相关图估计的放电统计数据在细胞类型之间有所差异(图4E)。
- 我们没有包括在麻醉动物或体外记录的神经元的放电统计数据,因为它们不能代表清醒状态。
Cell-type identification from a semi-supervised deep learning classifier
从半监督深度学习分类器中进行细胞类型识别
Para_01
- 图4F和表S1揭示了很难猜测哪些特定的波形和放电统计量测度将最有助于成功区分清醒动物中的细胞类型。
- 相反,原始数据(1)包含更丰富的信息,(2)为细胞类型的识别提供了无偏输入,并且(3)可能在不同区域、任务和物种之间具有普适性。
- 此外,我们使用了"三维自相关图"(3D-ACGs,图4G和4H,详细信息见图例)来量化放电统计量,这种方法可以对记录过程中和不同物种之间的放电率变化进行归一化处理。
Para_02
- 我们的分类器(图5A)由三部分组成:(1)一个多头、归一化的输入层,该层接受来自两个预训练变分自编码器的10维输出,其中一个用于3D-ACGs,另一个用于最优对齐和振幅归一化波形的平均全时间序列(参见STAR方法),这些输出来自信号最强的通道,以及一个单元的小脑层的"一位热"3位二进制码;(2)一个处理这三个输入的隐藏层;(3)一个输出层,每个细胞类型有一个输出单元。输出单元的值总和为1,因此分类器的输出是给定输入集属于五个细胞类型的概率。
- 我们使用梯度下降法和留一交叉验证法,在真实库的数据上训练了分类器中的权重。
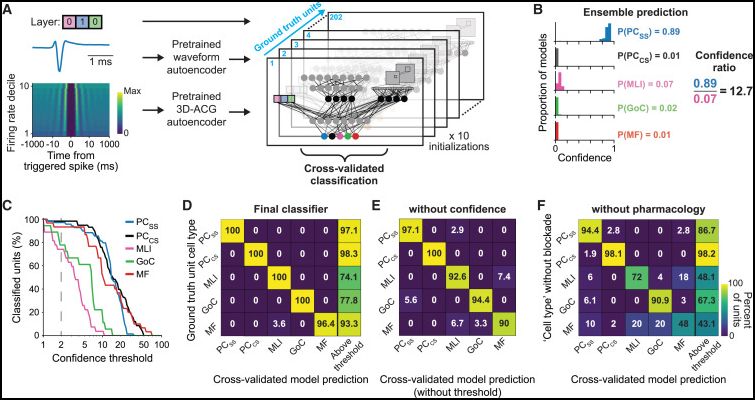
图片说明
◉ 图5。深度学习分类器在细胞类型识别方面的表现(A)分类器架构。我们使用202个地面实况单元中的每一个进行了10次不同的初始化训练,这些单元由分类器中的202页象征性表示。(B)直方图显示了分类器对一个留出神经元的10种不同初始条件下的预测。(C)根据我们选择的置信阈值比率,分类为不同单元的比例。不同颜色的数据代表不同地面实况细胞类型的统计数据。(D)混淆矩阵显示了分类器对该单元预测的结果与该单元的真实细胞类型之间的关系。每个单元格内的数字表示每种真实细胞类型的细胞类型预测百分比。最右侧的列显示了接收超过2的置信度的真实神经元的百分比,这些神经元被纳入混淆矩阵。(E)与(D)相同,但置信阈值= 0。(F)与(D)相同,但分类器是在所有光遗传激活单元上训练的,不需要突触阻断。另见图S5和S6。◉ 图5。深度学习分类器在细胞类型识别方面的表现(A)分类器架构。我们使用202个地面实况单元中的每一个进行了10次不同的初始化训练,这些单元由分类器中的202页象征性表示。(B)直方图显示了分类器对一个留出神经元的10种不同初始条件下的预测。(C)根据我们选择的置信阈值比率,分类为不同单元的比例。不同颜色的数据代表不同地面实况细胞类型的统计数据。(D)混淆矩阵显示了分类器对该单元预测的结果与该单元的真实细胞类型之间的关系。每个单元格内的数字表示每种真实细胞类型的细胞类型预测百分比。最右侧的列显示了接收超过2的置信度的真实神经元的百分比,这些神经元被纳入混淆矩阵。(E)与(D)相同,但置信阈值= 0。(F)与(D)相同,但分类器是在所有光遗传激活单元上训练的,不需要突触阻断。另见图S5和S6。◉ 图5。深度学习分类器在细胞类型识别方面的表现(A)分类器架构。我们使用202个地面实况单元中的每一个进行了10次不同的初始化训练,这些单元由分类器中的202页象征性表示。(B)直方图显示了分类器对一个留出神经元的10种不同初始条件下的预测。(C)根据我们选择的置信阈值比率,分类为不同单元的比例。不同颜色的数据代表不同地面实况细胞类型的统计数据。(D)混淆矩阵显示了分类器对该单元预测的结果与该单元的真实细胞类型之间的关系。每个单元格内的数字表示每种真实细胞类型的细胞类型预测百分比。最右侧的列显示了接收超过2的置信度的真实神经元的百分比,这些神经元被纳入混淆矩阵。(E)与(D)相同,但置信阈值= 0。(F)与(D)相同,但分类器是在所有光遗传激活单元上训练的,不需要突触阻断。另见图S5和S6。◉ 图5。深度学习分类器在细胞类型识别方面的表现(A)分类器架构。我们使用202个地面实况单元中的每一个进行了10次不同的初始化训练,这些单元由分类器中的202页象征性表示。(B)直方图显示了分类器对一个留出神经元的10种不同初始条件下的预测。(C)根据我们选择的置信阈值比率,分类为不同单元的比例。不同颜色的数据代表不同地面实况细胞类型的统计数据。(D)混淆矩阵显示了分类器对该单元预测的结果与该单元的真实细胞类型之间的关系。每个单元格内的数字表示每种真实细胞类型的细胞类型预测百分比。最右侧的列显示了接收超过2的置信度的真实神经元的百分比,这些神经元被纳入混淆矩阵。(E)与(D)相同,但置信阈值= 0。(F)与(D)相同,但分类器是在所有光遗传激活单元上训练的,不需要突触阻断。另见图S5和S6。◉ 图5。深度学习分类器在细胞类型识别方面的表现(A)分类器架构。我们使用202个地面实况单元中的每一个进行了10次不同的初始化训练,这些单元由分类器中的202页象征性表示。(B)直方图显示了分类器对一个留出神经元的10种不同初始条件下的预测。(C)根据我们选择的置信阈值比率,分类为不同单元的比例。不同颜色的数据代表不同地面实况细胞类型的统计数据。(D)混淆矩阵显示了分类器对该单元预测的结果与该单元的真实细胞类型之间的关系。每个单元格内的数字表示每种真实细胞类型的细胞类型预测百分比。最右侧的列显示了接收超过2的置信度的真实神经元的百分比,这些神经元被纳入混淆矩阵。(E)与(D)相同,但置信阈值= 0。(F)与(D)相同,但分类器是在所有光遗传激活单元上训练的,不需要突触阻断。另见图S5和S6。◉ 图5。深度学习分类器在细胞类型识别方面的表现(A)分类器架构。我们使用202个地面实况单元中的每一个进行了10次不同的初始化训练,这些单元由分类器中的202页象征性表示。(B)直方图显示了分类器对一个留出神经元的10种不同初始条件下的预测。(C)根据我们选择的置信阈值比率,分类为不同单元的比例。不同颜色的数据代表不同地面实况细胞类型的统计数据。(D)混淆矩阵显示了分类器对该单元预测的结果与该单元的真实细胞类型之间的关系。每个单元格内的数字表示每种真实细胞类型的细胞类型预测百分比。最右侧的列显示了接收超过2的置信度的真实神经元的百分比,这些神经元被纳入混淆矩阵。(E)与(D)相同,但置信阈值= 0。(F)与(D)相同,但分类器是在所有光遗传激活单元上训练的,不需要突触阻断。另见图S5和S6。
Para_02
-
在优化分类器之前,我们在大量未标记的数据样本上训练了变分自编码器,以将波形和3D-ACG的高维度降低到10维。
- 这一步和其他步骤(参见图S5和STAR方法)有助于缓解过拟合问题,因为真实库中的训练样本数量相对较少。
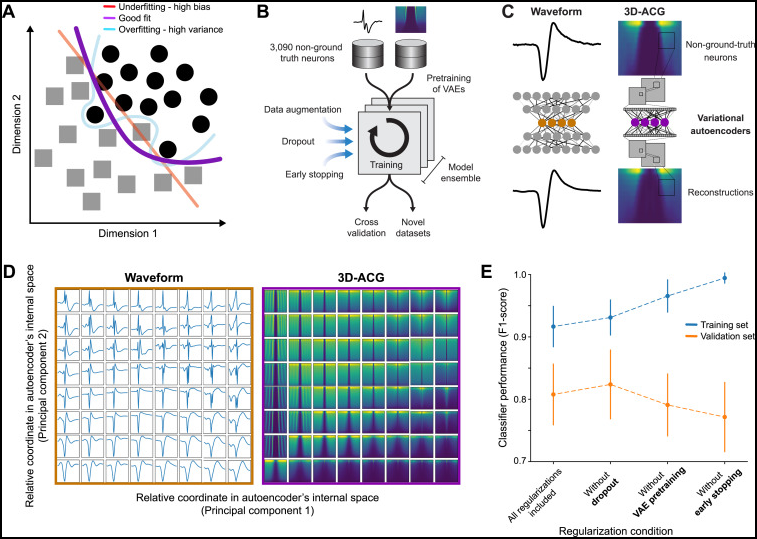
图片说明
◉ 图S5。包含在分类器策略中的正则化方法,以减轻过拟合,与图5相关。◉ 我们意识到深度学习分类器容易过拟合,尤其是在训练数据集规模较小的情况下。◉ 过拟合会导致对真实标签数据集进行分类时表现优异,但对新数据集(包括论文后期测试的专家标记的数据集)的泛化能力较差。◉ 在这里,我们概述了几项旨在避免过拟合的措施,并提供了一些证据表明这些措施是成功的。◉ (A) 过拟合问题的示意图。两组符号显示了可能用于区分两种不同细胞类型的特征分布。◉ 浅蓝色、橙色和品红色曲线分别展示了过拟合、欠拟合和正确分类可能导致的分类结果。◉ (B) 展示我们使用的正则化策略的示意图,包括通过输入变异自动编码器在未标记的数据集中进行降维、数据增强、丢弃、提前停止、模型集成以及留一交叉验证(详情见STAR方法)。◉ 我们还在独立的("新")专家识别的数据集上测试了分类器性能,如图6所示。◉ (C) 设计了两个变异自动编码器,一个用于波形,另一个用于3D-ACG的卷积变异自动编码器,旨在将各自的输入维度降低到一个10元素向量(彩色圆圈),随后可以作为最终分类器的输入(参见图5A)。◉ 每个变异自动编码器是一个人工神经网络,通过在编码和解码网络之间放置一个信息瓶颈("潜在空间")来减少输入维度。◉ 我们使用梯度下降法(见STAR方法)训练自动编码器的权重,使用的输入数据要么是波形(左图),要么是3D-ACG(右图),这些数据来自一组未标记的小脑神经元。◉ 训练过程中最小化提供的输入和编码-解码输出之间的差异。◉ (D) 两个变异自动编码器执行的降维捕获了我们在真实标签数据集和专家标记数据集中存在的波形和3D-ACG统计多样性(见STAR方法)。◉ 这里,我们展示了训练于小鼠神经元上的自动编码器捕获的波形和3D-ACG多样性,这些神经元与真实标签数据集无关。◉ 热图矩阵显示了波形和3D-ACG在自动编码器潜在空间中的表示方式。◉ 大多数低维表示位于体波形的变化中,这些变化确实是数据集中最常见的。◉ 然而,所有其他典型的脉冲形状也被表示出来,包括树突波形、双相和三相轴突波形,以及带有突触后去极化的波形。◉ 重建的3D-ACG也是如此,它们捕捉到了对应于爆发、振荡以及高和低放电率的活动模式。◉ 请注意,图中一些看起来不像是生物信号的痕迹不应被解释为模型失败,而应被视为用于可视化重建的插值过程的一个副产品。◉ (E) 系统地逐个去除正则化预防措施后的后验效应。◉ 作为正则化程序减轻过拟合的证据,该图显示,逐个去除这些预防措施会增加过拟合。◉ 它显示了在训练集上的性能逐渐提高,在验证单元上的性能逐渐下降,通过10次5折交叉验证进行了量化。◉ 这里,分类器性能通过"F1分数"来量化,定义为分类器的精确度和召回率的调和平均值。
Para_02
- 我们不仅评估了分类器输出的准确性,还评估了其"置信度"。
- 对于每次留一法样本(n = 202个真实单位),我们在一个由10个具有随机初始条件的模型组成的集合中平均了细胞类型概率。
- 在图5B中,分类器反复预测被保留的单元是浦肯野细胞简单放电(平均概率为0.89)。
- 我们通过"置信比"量化了分类器的置信度,计算方法是最有可能的细胞类型平均概率与第二可能的细胞类型平均概率的比率。
- 能够根据所选置信阈值作为置信度进行分类的真实单位百分比随着置信比的增加而减少(图5C)。
- 分类器的置信度对于浦肯野细胞简单和复杂放电以及苔藓纤维来说更高,相比于高尔基细胞或MLI。
- 在我们的其余分析中,我们选择了置信阈值为2,因为它在尽可能多地分类神经元和准确分类之间提供了折衷。
Para_03
- 对于每个超过置信阈值的保留神经元,我们将其分配给具有最高平均概率的细胞类型,跨越了10次分类器运行。
- 对于74%和78%的真实MLI和Golgi细胞,置信度超过了阈值,分类器正确地将它们归类,即混淆矩阵的对角线上值为100%(图5D)。
- 超过90%的苔藓纤维、浦肯野细胞简单尖峰和复合尖峰超过了置信阈值,并且几乎所有都被正确识别。
- 没有置信阈值的情况下,分类器的准确性下降,但在所有细胞类型上的准确性仍然超过了90%(图5E)。
- 如果没有层信息,分类器的表现较差,主要是因为层信息使它能够更好地区分Golgi细胞和某些MLI(图S6)。
- 最后,如果我们在训练分类器时不对成功突触阻断进行考虑,而是使用所有光遗传激活的单元,分类器的性能会下降(图5F)。
- 因此,在真实库中只包括那些在确认突触阻断期间持续光遗传激活的细胞是很重要的。
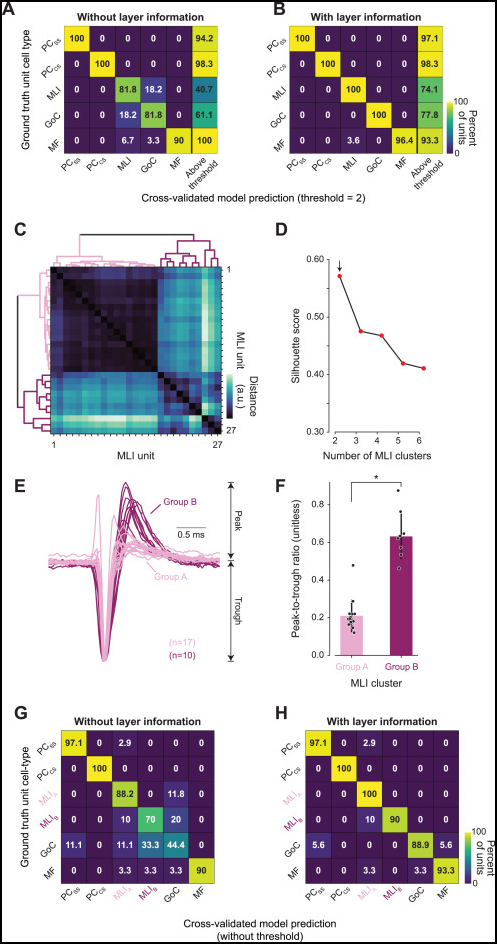
图片说明
◉ 图 S6. 在不将层信息作为输入的情况下分类器混淆情况以及基于存在两组分子层中间神经元且具有不同波形形状对混淆情况的解释,与图 5 相关。◉ 当我们将层信息作为输入时,分类器的表现更好,尤其是在区分分子层中间神经元和高尔基细胞方面(A 和 B)。◉ 在进一步研究中,我们发现真值库中的整个分子层中间神经元样本的波形暗示了双峰分布。◉ 在这里,我们定量地描述这两组,并展示了分类器能够区分这两组分子层中间神经元。◉ 由于一组分子层中间神经元(MLIB)的波形与位于颗粒细胞层中的高尔基细胞非常相似,因此分类器在有层信息输入时表现优于没有层信息输入的情况(G 和 H)。◉ 分子层中间神经元中的两种不同的波形可能与解剖结构或功能差异有关,但其他人的工作表明,仅凭波形无法区分两种已知类型的分子层中间神经元。◉ (A) 当未将层作为输入提供给分类器时,混淆矩阵显示了分类器交叉验证性能。◉ 这里,矩阵中的每个条目值表示 y 轴上的真实细胞类型中有多少比例被分类器预测为 x 轴上的细胞类型。◉ 注意,在没有层信息的情况下,分类器在分子层中间神经元和高尔基细胞之间存在混淆。◉ (B) 与 (A) 相同,但显示了当我们包含层作为分类器输入时的改进。◉ (C) 地真值库中整个 27 个分子层中间神经元样本归一化波形之间的成对距离矩阵。◉ 左侧和顶部的线显示通过层次聚类获得的树状图。◉ (D) 基于分子层中间神经元聚类的轮廓分数函数。◉ 高分表示样本正确匹配到其所属的簇并且与其他相邻簇分离。◉ 最大化轮廓分数提供了对潜在簇数量的无偏估计。◉ 在这里,假设两个簇的最大化轮廓分数。◉ 最大的轮廓分数是 +1,最小的是 -1。◉ (E) 根据 (C) 中的成对距离矩阵和树状图,将分子层中间神经元的波形分为 A 组(n = 17)和 B 组(n = 10),用不同颜色表示。◉ (F) 比较两个分子层中间神经元簇的峰谷比。◉ (G) 当未将层作为输入提供给分类器时,带有 A 组和 B 组分子层中间神经元单独标签的混淆矩阵显示分类器交叉验证性能。◉ 这里,矩阵中的每个条目值表示 y 轴上的真实细胞类型中有多少比例被分类器预测为 x 轴上的细胞类型。◉ 注意,没有层信息时,分类器特别混淆了分子层中间神经元 B 组神经元和高尔基细胞。◉ (H) 与 (G) 相同,但有层信息。
Classifier validation of expert-labeled datasets
专家标注数据集的分类器验证
Para_01
- 我们接下来评估了真实分类器的泛化能力,通过预测从老鼠和猴子身上记录到的专家分类的非真实分类神经元的细胞类型。
Para_02
- 我们利用了在2020年版本的分类器,该分类器通过对202个真实标签单元进行每单元10次分类器实例化训练而产生。
- 我们将每个单元在专家分类的数据集中2020个分类器的概率平均,并绘制了分类器分配给各个细胞类型的概率作为函数的图形(图6A和6B)。
- 单元格出现在恰好一个由五个不同图形中的一个中,即被分类器赋予最高概率的细胞类型所对应的图。
- 例如,最左边的图表报告了所有被分类为最有可能是浦肯野细胞简单尖峰的单元的概率与细胞类型的关系。
- 真实标签分类器与人类专家在老鼠和猴子中几乎所有高于置信阈值的单元的细胞类型上达成了共识(图6C和6D)。
- 此外,它正确地从包含简单尖峰触发复杂尖峰暂停记录中识别出了老鼠和猴子的浦肯野细胞简单尖峰和复杂尖峰(图6E)。
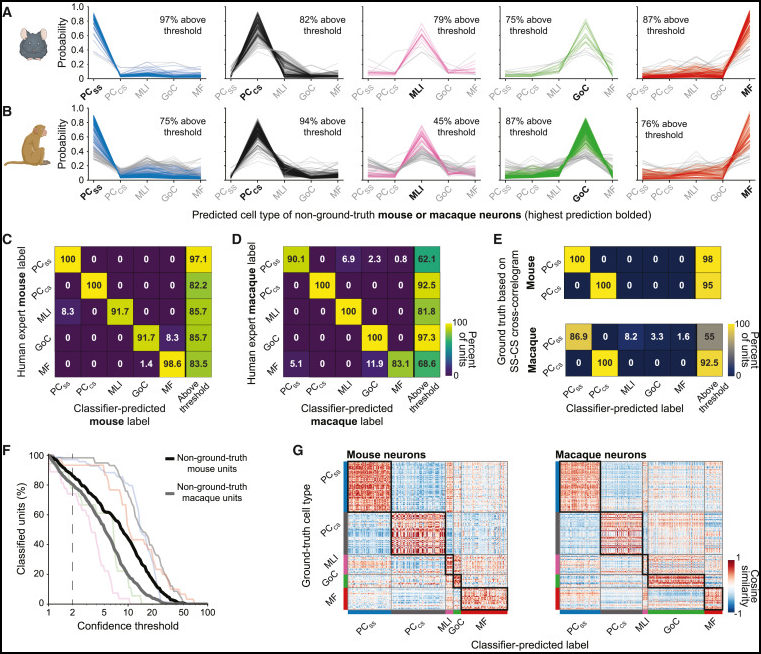
图片说明
◉ 图6。基于专家分类的数据集的小鼠和猴子的真实分类器性能(A)分类器分配的概率作为小鼠专家分类神经元细胞类型的函数,每个细胞类型由分类器赋予最高概率都有单独的图表。从左到右,最高概率的细胞类型依次是浦肯野细胞简单放电(PCss)、浦肯野细胞复杂放电(PCcs)、分子层中间神经元(MLI)、高尔基细胞(GoC)和苔藓纤维(MF)。彩色与灰色轨迹代表超过或未达到置信阈值2的神经元。(B)同(A),但适用于小脑绒球复合体的猴子专家分类神经元。(C)比较分类器预测与记录中小鼠专家标记的细胞类型的实际对应矩阵。每个单元格中的数字表示y轴上专家分类的细胞类型占x轴上分类器预测的细胞类型的百分比。最右侧列显示了从小分类器获得的置信度大于2的专家分类神经元的百分比。(D)同(C),适用于来自猴子绒球复合体的专家分类神经元。(E)混淆矩阵显示了分类器输出与真实识别之间的良好一致性,对于小鼠和猴子,基于简单放电中存在复杂放电触发的暂停,可以识别浦肯野细胞的简单和复杂放电。(F)根据置信阈值对3种准备情况下的分类单元百分比进行比较。淡色轨迹显示了来自图5C的真实库的相同曲线。粗黑线和灰线分别显示了小鼠和猴子非真实库单元的结果。(G)自编码器输出的一致性,用于真实分类与跨准备情况的专家分类神经元。每行对应一个通过真实分类识别的神经元。每列对应一个来自小鼠(左侧)或猴子(右侧)的分类器识别的神经元。行和列交叉处的颜色表示自编码器输出的波形和自相关图的余弦相似度,红色越深表示相似度越高。另见图S7。
Similar properties within cell types across species and cerebellar regions
跨物种和小脑区域内的相似细胞类型属性
Para_01
- 三个额外的分析表明,基于地面真实分类器在专家分类数据上的成功是由于每个细胞类型在各个数据集中的波形和放电统计的统计相似性。
- 首先,随着置信阈值的增加,两个专家分类单元样本和地面真实数据集的具有高置信度的单元百分比同样减少(图6F,粗灰线和黑线与彩色线相比)。
- 其次,对分类器自动编码器输出的分析显示,专家分类数据和地面真实数据的降维表示之间有很好的一致性(图6G)。
- 第三,检查波形、二维自相关图和三维自相关图揭示了地面真实数据、非地面真实小鼠数据以及猴子记录之间的惊人相似性(图S7)。

图片说明
◉ 图 S7。与图 6 相关的不同细胞类型在真实库和来自小鼠和猴子的专家标注数据中的波形相似性和静息放电统计数据。◉ 这里,我们只包括那些置信度大于 2 的神经元。◉ (A) 不同细胞类型的波形跨实验室和物种。◉ 在第一行,波形根据小鼠的真实细胞类型进行划分。◉ 在第二行和第三行,细胞类型根据对小鼠和猴子记录的非真实细胞类型的分类器预测进行划分。◉ 回想一下,所有波形都被翻转,使得最大的偏转为负。◉ 细胞类型的缩写是:PCSS,浦肯野细胞简单尖峰;PCCS,浦肯野细胞复合尖峰;MLIs,分子层中间神经元;GoCs,高尔基细胞;MFs,苔藓纤维。◉ (B) 与 (A) 类似,但显示二维自相关图。◉ 注意,自相关图中的脉冲计数已通过箱宽归一化,因此 y 轴单位为脉冲/秒。◉ (C) 来自小鼠的真实库以及来自小鼠和猴子的非真实记录的 5 种细胞类型的三维自相关图(3D-ACGs)示例。◉ 对于非真实记录,我们选择了分类器预测与专家识别细胞类型一致的例子。
Functional dissection of cerebellar circuits enabled by cell-type identification
小脑电路的功能解剖得益于细胞类型的识别
Para_01
- 不同类型的神经元在行为过程中通常表现出不同的时间反应动力学。
- 我们的四个实验室记录了小脑中四个已知参与各自行为的不同区域(图7A):小鼠外侧小脑中的奖赏条件反射,64小鼠副蚓部HV/HVI最深层的眨眼条件反射,65,66小鼠简单叶中的运动,67以及猴子绒球复合体中的平滑跟踪眼动。
- 每种细胞类型的传统单次试验平均刺激时间直方图显示了不同细胞类型之间的不同时间反应(图7B)。
- 大多数细胞类型在个体单元之间显示出相对较小的变化(图7B中的误差带),表明细胞类型内部的功能同质性。
- 同时,在某些区域的一些细胞类型中展示了多种功能放电模式(例如,在眨眼条件反射期间的浦肯野细胞或在平滑跟踪期间的MLI)(图7C)。
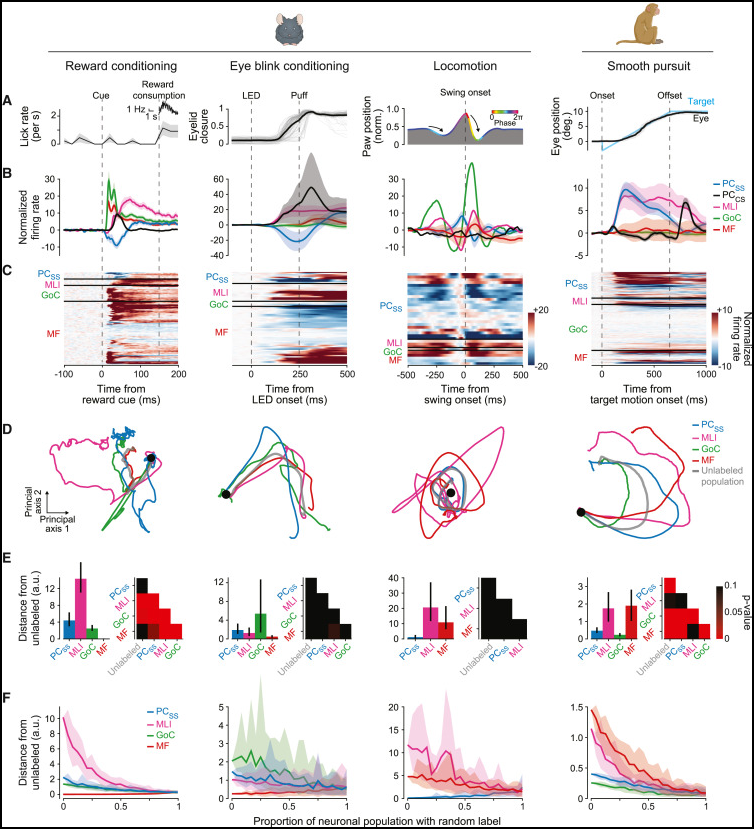
图片说明
◉ 图7。不同细胞类型的神经元群体动力学(A)四种行为任务中四个实验室和两个物种的动作反应时间过程。从左到右:当一个电磁阀点击声提示奖励时每秒舔舐次数(小鼠),插图显示了在一个扩展的时间尺度上的舔舐率;在用LED灯和气流配对条件反射后眼睑闭合(小鼠);行走过程中摆动相开始时爪子的位置(小鼠);以及在平滑目标运动追逐中的眼球位置(猴子)。(B)不同细胞类型在相应行为期间的平均放电率。不同的颜色表示不同细胞类型的归一化反应。误差带显示了神经元的均值±标准误。(C)根据细胞类型划分的神经元放电率。每个线的颜色显示了在四种行为中个体神经元的放电率随时间的变化,并归一化使得基线期的标准差为1。(D)群体动力学的二维轨迹。轨迹从黑色实心圆点开始,灰色轨迹显示了整个细胞类型无关的群体,而彩色轨迹则显示了不同的细胞类型。(E)不同细胞类型动态群体轨迹之间的统计分析差异。对于每种行为,直方图显示了每种细胞类型轨迹与细胞类型无关轨迹的距离。半矩阵总结了所有轨迹相互比较的p值。黑色方块表示不具有统计显著性。(F)随机重新标记每种细胞类型群体的不同比例对这种细胞类型动态群体轨迹与细胞类型无关群体轨迹之间距离的影响。
Para_01
- 为了揭示不同细胞类型特异性群体的神经动力学是否可以相互区分,我们在单次实验会话记录的3只小鼠和更大伪群体的多会话记录中对试验平均数据的神经状态空间进行了降维处理(图7D)。
- 我们优化地将为每种细胞类型群体分别计算的主要成分与整个"细胞类型非特异性"群体的主要成分对齐(参见STAR方法)。
Para_02
- 试验平均后的不同细胞类型的神经轨迹在奖赏条件反射和平滑跟踪过程中显示出与细胞类型非特异性群体以及其他细胞类型在动态上的统计学差异,但在眼睑条件反射或运动过程中则没有(图7D)。
- 两种统计分析验证了任何细胞类型之间的动态差异并不是由于不等或有限的群体规模(图7E)或者分类器对细胞类型的大致误识别所导致的(图7F)。
- 我们研究的两种行为中,不同细胞类型的轨迹未能在两个行为中分化,并不一定意味着所有细胞类型的动态是相同的。
- 相反,这可能是由于同一细胞类型内的动态多样性与跨细胞类型的多样性相匹配甚至超过后者,或者是由于每个细胞类型中存在2个或更多亚组。
- 需要进一步基于功能特性在细胞类型内进行聚类以解开功能亚类。
- 综上所述,图7中的分析展示了如何通过细胞类型识别来提供生物学见解,从而了解不同细胞类型之间的相互作用如何使神经回路控制行为。
Discussion
Para_01
- 我们的方法基于光遗传学刺激并在突触阻断剂存在的情况下识别,提供了清醒小鼠小脑细胞类型的高可靠性的电生理特性基准库。
- 该基准库包括神经元的波形、尖峰训练的统计信息以及我们记录每个单元的小脑层。
- 我们的半监督深度学习分类器能够准确地识别基准库中的细胞类型,并同时报告其在每次识别中的置信度。
- 来自小鼠和猴子的记录由分类器预测的细胞类型与专家的评估一致。
- 分类器的成功是一个出乎意料的证据,表明不同小脑细胞类型的特性在物种和小脑区域之间是一致的。
- 我们对分类器的准确性和精确性感到鼓舞,并期望将来有可能将从体外记录获得的细胞类型与通过其他分析水平(包括解剖和分子指纹)获得的细胞类型对齐。
Para_02
- 细胞类型鉴定能够提供关于电路组织、电路动态以及输出动态与行为之间关系的重要生物学见解。
- 对于两种我们研究的行为,不同的细胞类型显示出了非常不同的群体动态,这表明不同细胞类型在小脑处理和电路计算中扮演了不同的角色。
- 而对于另外两种行为,所有不同类型的细胞都共享相同的群体动态,这可能是由于每种细胞类别中高度异质性的神经反应特征导致的,这些特征构成了这些行为的基础。
- 细胞类型鉴定开启了使用多种不同策略获取生物学见解的大门,这些见解将从未来的分析中浮现出来。
- 例如,细胞类型鉴定与同时记录的结合应该可以使用交叉互相关图分析法直接测量连接强度和电路功能,并且可以在单一电路中定位多个学习位点。
- 或者,对不同类型细胞中的动态进行分析,可以揭示与输出神经元的运动行为相关的群体动态,以及特定类型的中间神经元中的上下文相关性。
Para_03
- 过去尝试识别小脑皮层神经元的离散群体19,50,51,52,53,81的努力不如我们成功。
- 先前的研究要么通过体外发现的放电特征进行定性匹配来识别神经元,要么通过在麻醉准备中记录并在解剖学上通过细胞外标记识别神经元来达成一致。
- 19,49,50,51,53,81我们的记录显示,在清醒、行为中的小鼠中,过去尝试用于总结放电活动的离散指标存在很大的变异性,这些指标不仅在真实类别内而且在类别之间都有所不同(图4F;表S1)。
- 因此,依赖于有限数量特定特征的分类方案不太可能很好地推广到其他任务或区域,或者从麻醉准备推广到行为准备中。
原始波形和3D-ACGs:原始特征是一种无偏输入,允许分类器利用波形和放电统计数据中的大量信息。3D-ACGs对放电率的变化进行了归一化处理,并创建了一种可以跨小脑区域、实验任务和物种进行比较的统计量。使用单通道波形(而不是空间足迹)使分类器能够泛化到不同类型的电极。
缓解过拟合:一种半监督的深度学习策略使用相对少量的真实神经元对分类器进行了训练,同时通过使用大规模未标记的数据集来预训练自动编码器,减少了过拟合的可能性(参见STAR方法和图S5)。成功预测的细胞类型与两个专家分类的数据集一致,支持了分类器的泛化能力。
我们特别注意使我们的分类器值得信赖。我们通过在相同数据上训练多个模型来建立信心。通过要求置信度超过给定阈值,我们提高了模型在真实数据以及非真实记录上的准确性。选择置信度阈值可以让用户权衡是否包括所有神经元,即使某些细胞类型分配可能是错误的,或者只包括较少的神经元但对细胞类型分配有更大的确定性。
Para_04
- 当它将层信息作为输入时,分类器能够更准确地识别单元。
- 相反,它创建了一个平台,随着我们在小脑中对其他细胞类型的准确识别(例如,通过改进的记录探针识别颗粒细胞)的实现,这个平台将变得更加有用。
- 此外,由于波形和放电统计对于区分同一层中的细胞是必要的,分类器会对细胞类型做出统计决策,而不仅仅是依赖于层来识别细胞类型。
- 89 层在小脑中以特定的方式定义,1 但我们认为层信息更普遍地作为一种‘局部电特性’的例子。
- 我们设想还有其他方法可以量化这些特性,例如,局部场电位 (LFP) 和电流源密度分析,90,91 这些方法将在没有分层结构的脑区发挥作用。
Para_05
- 我们项目开始时的目标是从小脑皮层的细胞外记录中实现细胞类型识别,并且可以在不同的实验室和物种之间应用。
- 我们认为,任何对电生理数据进行足够严格的管理的小脑记录实验室都可以放心地使用这个分类器。
- 然而,如果缺乏严格的管理,将导致分类器输入的数据噪声大且不必要的变化,从而污染细胞类型的识别结果。
- 利用层信息来提高分类的相关性应该适用于其他具有可测量局部电信号的结构——如大脑皮层、海马体和上丘。
- 我们分类器中使用的策略通过展示如何在减少原始输入数据维度的同时克服训练集中小样本数量的挑战,实现了泛化。
- 此外,光遗传激活与突触阻断的结合增加了创建真实库的严格性。
Limitations of the study
研究的局限性
Para_06
- (1) 光遗传学识别仅限于小脑皮层的主要细胞类型,因为目前还没有可用的Cre品系来让我们将最近突出的一些新细胞类型添加到基准库中。
- (2) 尽管付出了相当大的努力,从颗粒细胞中记录仍然具有挑战性。
- 高阻抗和/或更高密度记录位点的探针技术的进步最终将解决这一局限性。
- (3) 我们预计我们的分类器可以"开箱即用"用于小脑中的细胞类型识别,但我们的策略需要被用来开发其他区域的基准库和成功的分类器。
- 由于不同大脑区域中的神经元的生物物理特性和放电特性从根本上是不同的,因此不可能有一个‘通用’的、不受区域限制的分类器。
- (4) 目前,没有办法将基于细胞外电生理的细胞类型识别与单细胞RNA测序(RNA-seq)、相邻细胞标记或体内记录和单细胞成像相结合的结果进行对齐。
- 例如,基于细胞外电生理的方法无法通过如RNA-seq等方法来标记记录的细胞,以便在事后分析分子身份。
- 尽管存在这些局限性,我们的研究展示了一种策略,该策略允许从行为中的小鼠和猴子的大规模电生理记录中稳健可靠地识别不同的细胞类型。
- 这种策略对于使用高密度硅探针的小脑研究人员群体来说将非常有价值,并为在其他神经回路中应用的原则性的半自动化检测细胞类型提供了模板。
Resource availability
Lead contact
主要联系人
Para_01
- 进一步的信息和资源、数据集、协议的需求应联系主要联系人Javier F. Medina(jfmedina@bcm.edu),并将由其满足。
Materials availability
材料可用性
Para_01
Data and code availability
数据和代码可用性
支持本文发现的数据在发表日期后可通过关键资源表中列出的DOI公开获取。
支持本文发现的原始代码已存放在 GitHub 上,并在发表日期通过关键资源表中列出的 DOI 以及 https://c4-database.com 公开获取。
重新分析本文报道的数据所需的任何附加信息可应请求从主要联系人处获得。
Acknowledgments
Para_01
- 我们要感谢Bonnie Bowell、Soyon Chun、Wenjuan Kong、Margaret Conde Paredes、Caroline Reuter和Stefanie Tokiyama提供的技术支持;
- Rob Campbell支持了定制双光子断层扫描平台的构建;
- Julie Fabre就尖峰排序质量指标提供了建议。
- 资金由NIH赠款R01-NS112917(S.G.L.、J.F.M.和C.H.)、K99-EY030528(D.J.H.)、R01-NS092623(S.G.L.)和R01-MH093727(J.F.M.)提供;
- Wellcome Trust PRF 201225/Z/16/Z和224668/Z/21/Z(M.H.)以及职业发展奖225951/Z/22/Z(D.K.)资助;
- EMBO ALTF 914-2015(D.K.)资助;
- 欧洲联盟的‘地平线2020’研究与创新计划下的Marie Skłodowska-Curie赠款协议编号844318(M.O.)和891774(F.N.)资助;
- 以及SYNCH项目由欧盟委员会根据‘地平线2020’FET前瞻性计划资助的赠款协议ID 824162(D.C.)。
Author contributions
Para_01
- 概念化,M.B.,D.J.H.,B.A.C.,D.C.,S.G.L.,D.K.,C.H.,M.H.和J.F.M.;方法论,M.B.,D.J.H.,F.N.,F.D'A.和A.S-L.;软件,M.B.,D.J.H.,F.N.,F.D'A.,M.G.M.L.和A.L.;正式分析,M.B.,D.J.H.,F.N.,M.E.H.,F.D'A.,M.O.,A.S-L.,D.K.,M.G.M.L.和N.J.H.;调查,M.B.,D.J.H.,M.E.H.,M.O.,A.S-L.,Y.Y.C.,M.M.,H.N.S.,M.Z.,S.O.,D.K.和C.H.;原始草稿撰写,S.G.L.;审查与编辑,M.B.,D.J.H.,S.G.L.,D.K.,C.H.,M.H.和J.F.M.;可视化,M.B.,D.J.H.,M.E.H.,D.K.和J.F.M.;监督,M.B.,S.O.,B.A.C.,D.C.,S.G.L.,D.K.,C.H.,M.H.和J.F.M.;资金获取,B.A.C.,S.G.L.,C.H.,M.H.和J.F.M.。
Declaration of interests
Para_01
STAR★Methods
Key resources table
关键资源表
Experimental model and study participant details
实验模型和研究参与者详情
Ethical statement
伦理声明
Para_01
- 我们在四个实验室进行了实验,并对两种物种进行了研究,分别是小鼠和恒河猴。
- Häusser 实验室中的所有小鼠操作均获得了伦敦大学学院当地动物福利和伦理审查委员会的批准,并且是在英国政府内政部的许可下进行的,遵循了1986年的《动物(科学程序)法案》以及2010/63/EU号欧洲指令,该指令旨在保护用于实验目的的动物。
- Hull 和 Medina 实验室中的小鼠操作分别得到了杜克大学和贝勒医学院的机构动物护理与使用委员会的预先批准,这些批准基于美国国立卫生研究院的指导方针。
- Lisberger 实验室中的猴子操作得到了杜克大学的机构动物护理与使用委员会的预先批准。
- 我们竭尽全力减少所需的动物数量以及它们可能经历的任何可能的痛苦。
Animals
动物
Mouse
Häusser:通道视紫红质-2(ChR2)在各种小脑细胞类型中表达,主要是通过将Cre品系与Cre依赖性ChR2-eYFP报告品系(Ai32,B6.Cg-Gt(ROSA)26Sortm32(CAG-COP4∗H134R / EYFP)Hze / J)交叉,或在一部分实验中,通过注射Cre依赖性ChR2病毒(AAV1.CAGGS.Flex.ChR2-tdTomato [UPenn])。Cre品系包括:BAC-Pcp2-IRES-Cre(B6.Cg-Tg(Pcp2-cre)3555Jdhu / J),旨在标记浦肯野细胞;Nos1-Cre(B6.129-Nos1tm1(cre)Mgmj / J),旨在标记分子层中间神经元;Glyt2-Cre(Tg(Slc6a5-cre)1Uze),旨在标记高尔基细胞;以及Math1-Cre(B6.Cg-Tg(Atoh1-cre)1Bfri / J),旨在标记颗粒细胞。除了转基因交叉和病毒ChR2表达外,我们还使用了Thy1-ChR2品系18(B6.Cg-Tg(Thy1-COP4 / EYFP)18Gfng / J)来在苔藓纤维中表达ChR2。每种策略的记录如下:L7-Cre x Ai32 – 1次记录(1只小鼠);Nos1-Cre x Ai32 – 40次记录(34只小鼠);Nos1-Cre + AAV1.CAGGS.Flex.ChR2-tdTomato – 3次记录(3只小鼠);GlyT2-Cre x Ai32 – 32次记录(31只小鼠);GlyT2-Cre + AAV1.CAGGS.Flex.ChR2-tdTomato – 3次记录(3只小鼠);Math1-Cre x Ai32 – 47次记录(38只小鼠);Math1-Cre + AAV1.CAGGS.Flex.ChR2-tdTomato – 3次记录(3只小鼠);以及Thy1-ChR2品系18 – 26次记录(22只小鼠)。我们进一步调查了Cre转基因交叉在小脑中opsin表达的特异性,方法是将上述Cre品系与Cre依赖性tdTomato报告品系Ai9(B6.Cg-Gt(ROSA)26Sortm9(CAG-tdTomato)Hze / J)交叉,以便我们可以通过细胞质而非膜结合的荧光来评估表达特异性。
-
构建表达ChR2或抑制性GtACR2或ArchT视蛋白的小鼠的方法有:1)将意图标记分子层中间神经元的c-kitIRES-Cre系与Cre依赖性的ArchT-GFP报告系(Ai40 (B6.Cg-Gt(ROSA)26Sortm40.1(CAG-aop3/EGFP)Hze/J))进行杂交,或者2)将意图标记颗粒细胞的BACα6Cre-C系与Cre依赖性的ChR2报告系(Ai32)进行杂交。另外,我们使用相同的Cre系注射了Cre依赖性的病毒:(向c-kitIRES-Cre和BACα6Cre-C注射AAV1.CAGGS.Flex.ChR2-tdTomato,向c-kitIRES-Cre注射AAV1.Ef1a.Flex.GtACR2.eYFP)。此外,我们还使用Thy1-ChR2系18来在苔藓纤维中表达ChR2。每种策略的记录如下:c-kitIRES-Cre + AAV1.CAGGS.Flex.ChR2-tdTomato – 8次记录(2只小鼠),c-kitIRES-Cre x Ai40 – 3次记录(1只小鼠),c-kitIRES-Cre + AAV1.Ef1a.Flex.GtACR2.eYFP – 11次记录(6只小鼠),BACα6Cre-C x Ai32 – 22次记录(12只小鼠),BACα6Cre-C + AAV1.CAGGS.Flex.ChR2-tdTomato – 10次记录(4只小鼠),以及Thy1-Chr2系18 – 13次记录(4只小鼠)。
所有实验均在从杰克逊实验室获得的野生型C57BL/6J小鼠中进行。
Monkey
Para_01
- 记录是在利斯贝格尔实验室进行的,对象是非人灵长类动物,在三只成年雄性恒河猴(Macaca mulatta)身上进行,它们的体重在10-15公斤之间。
Method details
方法细节
Surgery
外科手术
Mouse
Häusser:我们在小脑上安装了一个定制的铝制头板,头板有一个长5毫米宽9毫米的椭圆形内开口。在手术前至少1小时给小鼠注射了一种皮质类固醇消炎药(地塞米松,0.5毫克/千克),随后在手术前立即注射了镇痛非甾体抗炎药(美洛昔康,5毫克/千克)。麻醉诱导和维持分别使用了5%和1-2%的异氟烷。头板定位在左小脑半球的小脑简单叶上,相对于横截面呈约26度角,并用牙科水泥(Super-Bond C&B,Sun-Medical)固定在颅骨上。术后镇痛(卡普里夫,5毫克/千克)持续了3天。经过几天的适应期后,在记录装置上进行了直径1毫米的颅骨切开术和硬脑膜切开术,以便神经像素探针可以进入小脑简单叶(距中线3毫米,位于顶枕裂前方)。在进行颅骨切开术之前,用牙科水泥将一个圆锥形的丁腈橡胶密封圈(型号749-581,RS组件)固定在头板上作为浴槽。暴露的脑组织随后被一层湿润的止血海绵(Surgispon)和硅胶密封剂(Kwik-Cast,WPI)覆盖,直到实验开始(恢复后1-2小时)。在实验开始时,小鼠头部固定,移除硅胶密封剂,并立即应用生理HEPES缓冲盐溶液以保持颅骨切口的湿润。
我们安装了一个钛合金头柱(HE Palmer,32.6x19.4 mm)到颅骨上,并在左小脑上方放置了一个不锈钢地线螺钉(F.S. Tools),两者均用Metabond(Parkell)固定。手术前3-4小时,小鼠接受了地塞米松(3 mg/kg),并在诱导异氟烷麻醉前20分钟注射了酮胺/赛拉嗪(50 mg/kg和5 mg/kg,腹腔内注射)以及卡洛芬(5 mg/kg)。手术过程中,异氟烷的浓度保持在1-2%,以维持适当的呼吸速率并防止脚趾压迫反应,这些参数在整个手术期间都进行了监测。使用加热垫(TC-111 CWE)维持体温。术后48小时内,小鼠每天两次接受布托啡诺和头孢唑林(分别为0.05 mg/kg和50 mg/kg,皮下注射),并连续观察4天。恢复2周后,小鼠在记录前4-24小时接受了地塞米松(3 mg/kg)。在第一次记录当天,在1-2%异氟烷麻醉下,于小脑蚓部或外侧小脑(相对于前囟:AP方向-6.0至-7.0 mm,ML方向1.0至2.8 mm)处打开约0.5-1.5 mm的开颅术,并在两次记录之间用Kwik-Cast(WPI)封闭并覆盖Metabond。后续记录可以在小于30分钟的1-2%异氟烷麻醉下重新打开开颅术。
梅迪娜:术前进行了镇痛处理(5 mg/kg 美洛昔康,0.02 mL 0.5% 布比卡因和 2% 利多卡因),并在无菌条件下进行了手术。小鼠通过异氟烷麻醉(诱导时氧气中体积占比 5%,维持时 1-2%;SurgiVet),并放置在加热垫上以保持体温。颅骨被暴露并调整至与立体定位平面一致后,植入了两个不锈钢螺丝(相对于前囟:AP -0.3 mm,ML ±1.4 mm)以固定整个装置。一个定制的不锈钢头板覆盖在螺丝上,并用 Metabond 水泥(Parkell)将整个装置固定在颅骨上。此外,还进行了开颅手术(相对于前囟:AP -5.5 mm),移除了一块 5x2 mm 的骨头以暴露小脑蚓部及右侧前叶和后叶。然后用 Metabond 建造了一个腔室覆盖开颅处周围的裸露骨头,用一层生物相容性硅胶(Kwik-Cast,WPI)保护硬脑膜,并用硅胶粘合剂(Kwik-Sil,WPI)密封整个腔室。小鼠在术后完全恢复知觉前一直受到监测,并在手术后三天内提供了镇痛治疗。
Monkey
Para_01
- 部分灵长类动物的数据集在此前已发表,并附有详细的方法说明。108
- 猴子们在异氟醚的作用下经历了几个外科手术,为神经生理记录做准备。
- 我们依次进行了以下操作:(i) 将一个头架固定在颅骨上,(ii) 在一只眼睛的巩膜上缝制一个小线圈,使用搜索线圈技术监测眼睛的位置和速度,109 和 (iii) 植入一个瞄准小脑复合体的记录筒。
Extracellular recording procedures
细胞外记录程序
Mouse
Para_01
- 所有的实验室在进行小脑记录时都遵循相同的通用程序。
- 在进行 Neuropixels 记录之前,小鼠逐渐适应头部固定。
- Neuropixels 1.016 探针以每秒 1-4 微米的速度插入小脑皮层,同时监测电生理信号。
- 探针通常插入到距离小脑表面 2-3 毫米的记录深度,并在开始数据采集前至少静置 20 分钟。
- 颅骨切口周围的记录室用 ACSF 浸泡,可能含有或不含有突触阻断剂。
- 在所有三个实验室中,使用 SpikeGLX 获取 Neuropixels 数据,信号以 30 千赫的频率数字化。
Monkey
Para_01
- 每天,我们都会将钨单电极(FHC)或主要数据所用的定制Plexon s-探针敏锐地插入小脑绒球复合体。
- Plexon s-探针包括16个记录接触点(钨,直径7.5微米),分为两列,每列之间相隔50微米。相邻的接触行也相隔50微米。
- 一旦电极到达腹侧小结区,我们会让电极至少静置30分钟。
- 我们使用Plexon Omniplex系统以40千赫的采样率从所有接触点记录连续的宽带数据。
- 我们在数字化前使用截止频率为6千赫的4阶巴特沃斯低通硬件滤波器来消除由磁场产生的电场对记录信号造成的失真,该磁场用于测量眼球运动。
Reconstruction of Neuropixels probe tracts
神经元探针轨迹的重建
Häusser:小鼠用氯胺酮/赛拉嗪深度麻醉,并通过心脏灌注PBS,随后用4%多聚甲醛(溶于PBS)灌注。大脑被解剖并置于4%多聚甲醛中过夜固定,然后嵌入5%琼脂糖中。为了重建电极通路,我们使用定制的双光子断层扫描显微镜(与切片机耦合),110在ScanImage(2017b,Vidrio Technologies)和BakingTray的控制下,对全脑进行了3D堆栈成像。大脑以20 μm的间隔成像,并以40 μm(每片2个光学切片)的厚度切片。图像通过压电装置(PIFOC P-725,Physik Instrumente)安装的尼康16x/0.8NA物镜,在两个通道(绿光通道:500-550 nm,ET525/50;红光通道:580-630 nm,ET605/70;Chroma)中获取。每个切片以512x512像素分辨率在1025 x 1025 μm的瓦片上成像,重叠率为7%。
在最后一次录音日之后,小鼠用氯胺酮/阿扎嗪(350 mg/kg 和 35 mg/kg)深度麻醉,并用PBS灌注后,再用含有4%多聚甲醛的PBS灌注。大脑被取出并在含有4%多聚甲醛的PBS中过夜固定,然后使用振动切片机(Pelco 102)切成100 μm厚的切片。在切片前,一些大脑被封装在2%琼脂块中以保持稳定。切片先用DAPI(DAPI,二盐酸盐,268298,EMD Millipore)染色,然后用封片剂(Fluoromount-G,Southern Biotech)封片;或者直接用含DAPI的封片剂(DAPI Fluoromount-G,Southern Biotech)封片。使用共聚焦显微镜(Leica SP8)观察电极路径。
梅迪娜:在PBS中用4%的PFA灌注后,取出大脑,在相同溶液中至少固定12小时,然后在PBS中的30%蔗糖溶液中保护性冷冻48小时。将大脑对齐,使冠状切片与轨迹角度相匹配,并在低温超薄切片机(Leica CM1950)上切成50微米厚的切片。游离切片在PBS中恢复,并在 Hoechst 溶液中孵育3分钟(Hoechst 33342,在PBS- TritonX 0.25%中,来自赛默飞世尔科技公司,2微克/毫升)。然后在PBS中洗涤切片三次,并使用荧光保护介质(赛默飞世尔科技公司的 ProLong Diamond Antifade)封片。在 Axio Imager Z1 显微镜(蔡司公司)下以10倍放大率获取宽场荧光,并使用显微镜分析软件(蔡司公司的 ZEN 软件)进行轨迹重建和测量。
Optogenetic stimulation and pharmacology
光遗传学刺激和药理学
Häusser:采用1或2个蓝色发光二极管(470 nm,Thorlabs M470F3)进行了光遗传刺激,并在某些实验中使用蓝激光进行表面照明(Stradus 472,Voltran)。表面照明通过将激光或LED耦合到一根光纤(M95L01,Thorlabs)并通过一根套管(CFMXB05,Thorlabs)接触大脑表面附近探针来实现。在某些实验中,插入了第二个光源——一根锥形光纤(Optogenix 0.39NA/200μm),该光纤直接粘附在Neuropixels探头的头部。光纤尖端(表面光纤)和耦合套管(锥形光纤)处的总功率为1-6.9毫瓦。每次记录会话包括:(1)20分钟的自发活动基线期;(2)一组50次光遗传刺激(频率为0.1 Hz:一次250毫秒的刺激或一组五个频率为5 Hz的50毫秒刺激,具体取决于实验);(3)向小脑表面应用突触阻断剂鸡尾酒(Gabazine 0.2-0.8 mM,NBQX 0.8 mM,APV 1.6 mM,MCPG 0-1.3 mM),随后是20分钟的孵育期;(4)在存在突触阻断剂的情况下进行第二组50次光遗传刺激。我们没有使用蓝激光作为光刺激源记录任何神经元。
Hull: 表达ChR2或GtACR2的神经元分别被450纳米激光激活或抑制(MDL-III,OptoEngine),而表达ArchT的神经元则使用532纳米激光抑制(MGL-III,OptoEngine)。激光通过一个400微米的光纤耦合器(FT400 EMT,Thorlabs)耦合,该光纤耦合器距离脑表面4-10毫米。脑表面的功率约为2-30毫瓦,并且针对每次实验进行了校准,以产生最小伪迹的神经元反应。激光刺激持续50或100毫秒,并在20分钟基线期后在整个记录过程中以0.1赫兹的频率施加,期间有短暂暂停以补充ACSF或应用阻断剂(Gabazine 0.2-0.8 mM,NBQX 0.6-1.2 mM,AP-5 0.15-0.6 mM,MCPG 1-2.5 mM)。在除一个记录外的所有记录中,仅使用Gabazine实现突触阻断,该记录未应用阻断剂,3个MLI基于直接抑制GtACR2的短潜伏期(<3毫秒,这种潜伏期似乎太短而不像是由于突触输入而是直接抑制引起的)被接受到真实库中。
Histological assessment of opsin expression
视蛋白表达的组织学评估
Para_01
- 为了评估视蛋白表达的特异性,对不同转基因小鼠品系(Cre品系与tdTomato报告品系交配)的PFA固定脑组织进行了100微米切片,并准备进行免疫组化。
- 切片在室温下用2.5%正常驴血清/2.5%正常山羊血清/0.5%Triton X-100/PBS封闭4-6小时,然后在4℃下用初级抗体孵育4-6天,最后在4℃下用次级抗体孵育过夜。
- 使用的抗体如下:大鼠抗mCherry(1:250,赛默飞M11217),小鼠抗副钙素(1:1000,密理博MAB1572),驴抗大鼠- Alexa 594(1:1000,赛默科技),以及山羊抗小鼠- Alexa 633(1:1000,赛默科技)。
- Neurotrace 435/455(1:250,赛默飞N21479)加入到次级抗体溶液中。
- 切片被安装并在Zeiss LSM 880上成像,使用20倍物镜,以425x425微米的瓷砖大小,在1024x1024像素分辨率下进行成像。
Para_02
- 为了识别表达光遗传操作元件的脑小脑神经元类别,我们确定了荧光神经元所在的层,并检查它们是否表达了帕尔瓦蛋白(PV),所有分子层中间神经元和浦肯野细胞中都应该存在这种蛋白。
- 每个图像中小脑各层的位置是在Neurotrace(荧光尼尔森)通道中确定的。
- 使用Fiji(NIH),手动标记了表达tdTomato(作为Cre表达的替代标志)和PV的神经元胞体位置在灰度图像中。
- 如果神经元胞体位置相距小于5微米,则认为它们同时表达了tdTomato和PV。
- 通过将Neurotrace分层蒙版叠加到细胞位置上来确定每个神经元所在的层。
Quantification and statistical analysis
量化和统计分析
Analysis of extracellular recordings
细胞外记录的分析
Spike sorting and curation
在数据采集之后,我们使用Kilosort 2.017,112进行了自动尖峰排序,并使用Phy进行了初步的人工校正。然后,我们应用了多重质量检查,以确保用于进一步分析的最终选择的簇对应于具有生理波形、良好的隔离特性和很少或没有不应期违规的单个单元。严格的校正对于我们的长时间记录尤为重要,因为这些记录可能包含良好隔离期和漂移或不良单元隔离期交替出现的时期。我们将记录分为重叠片段(每10秒计算30秒片段),并在每个片段中计算‘假阳性’和‘假阴性’率。假阳性被定义为落在单元不应期(从给定尖峰± 0.8 毫秒)内的尖峰,并称为不应期违规(RPVs)。假阳性的比例估计为不应期违规率与平均放电率之比。假阴性被定义为由于落在记录噪声阈值以下而未被检测到的尖峰。它们通过拟合每个单元的尖峰幅度分布的高斯函数43,44并量化在噪声阈值处截断的曲线下的面积比例来估算。如果一个30秒片段的假阳性率和假阴性率低于5%,则认为该片段是可以接受的。可以接受的间隔被连接起来用于后续分类器训练。为了被纳入样本,单元必须在基线期间有3分钟可接受的隔离。对于基准库中的每个神经元,我们还进行了额外的分析,以评估单元隔离的质量。我们使用其他人推导出的方程48,根据不应期违规次数(nv)、总尖峰数(N)、不应期间隔(Tr)和平均放电率(MFR)来估计‘无污染尖峰的比例’(即不是噪声或相邻神经元的尖峰):1−nvN∗MFR∗Tr。除了少量的苔藓纤维外,基准库中的所有神经元在测量触发尖峰+1 毫秒的不应期违规时,无污染尖峰的比例超过0.97(Tr=0.001)。然而,苔藓纤维的不应期较短,可以以高达1,000次/秒的速度爆发。因此,我们重新测量了触发尖峰+0.5 毫秒的不应期违规(Tr=0.0005),并根据这些测量结果估计真实尖峰的比例。
在每次录音环节之后,我们使用半自动化的‘全二进制追踪’分类器将单个动作电位分配给假定的神经单元,该分类器旨在区分来自不同神经元的时间上和空间上重叠的脉冲。经过自动分类后,我们手动校正了我们的数据集,移除了具有显著的间脉冲间隔违规或低信噪比的神经元。我们灵长类动物数据集中大多数单元显著超过了用于自动分类小鼠数据的指标,这可能使我们所选择的灵长类动物单元倾向于那些更容易记录的单元。
Data harmonization
我们减轻了Neuropixels探头上的硬件滤波器带来的影响。有可能禁用该滤波器,我们在一些小鼠记录中确实这样做了。对于猴子记录而言,这并不是一个因素。为了使所有记录保持一致,我们将相似的因果一阶Butterworth高通滤波器(截止频率为300 Hz)应用于猴子的宽频电压记录以及在小鼠中未使用硬件滤波器获得的记录。
我们使用了一个两步程序来为每个单元构建高质量的波形模板:(a) 我们通过在主通道上将具有相似振幅的脉冲波形分组,并因此使它们处于相同的漂移状态(即探针相对于记录的神经元的位置)来对每个神经元的脉冲进行子采样:‘漂移匹配’,(b) 我们通过最大化每个脉冲与高振幅模板的互相关来重新对齐脉冲的时间:‘位移匹配’。对齐后,单个脉冲被平均,从而得到研究中的神经元的最终平均波形。Neuropixels 数据处理(非人工筛选、滤波、漂移位移匹配)使用了 NeuroPyxels 库。
我们通过选择最高振幅通道的平均波形对所有波形模板进行了预处理,将其重采样至30 kHz(如有必要),使其与峰值对齐,并在必要时翻转以确保波形中最大的偏转始终为负。我们知道动作电位波形的极性取决于多种因素,包括记录电极靠近树突、胞体和轴突的距离以及记录接触点和参考点之间的相对取向。我们使用这些协调一致的波形来计算总结统计量(图4;表S1),这些统计量以前被用于分类小脑神经元。
Assignment of layers with Phyllum
Para_01
- 对于小鼠的记录,我们使用Phyllum(一个为curation软件Phy定制设计的插件)将Neuropixels探针每个通道记录到的单元分配到一层。
- Phyllum中的层识别算法首先通过自动设置‘锚点’通道来启动,这些通道所记录的层可以通过普尔金耶细胞单元的存在(简单和复合尖峰)(普尔金耶细胞层锚点),苔藓纤维单元的三相波形(颗粒层锚点),或低1-2 Hz频率宽波形的树突复合尖峰(分子层锚点)来明确识别。
- 然后,Phyllum通过基于(1)靠近最近的普尔金耶细胞锚点的距离和(2)允许的层转换的迭代过程填充剩余通道的层。
- 分配到普尔金耶细胞层的每个通道必须包含至少一个在100 μm内的普尔金耶细胞记录,但该通道也可能包含位于邻近颗粒层或分子层中的其他单元。
- 如果两个连续的普尔金耶细胞锚点之间的所有通道都不包含另一个锚点单元,则它们的层被设置为‘未知’。
- 平均而言,Phyllum将Neuropixels探针上82%的所有通道分配到特定的层。
- 对21条记录轨迹的组织学重建确认了对于分配到特定层的通道,这种分配是非常准确的:分子层通道>99%,颗粒层通道>98%,普尔金耶细胞层通道>95%。
Identification of units directly responsive to optogenetic stimulation
Para_01
- 在光遗传激活实验中,如果记录到的单元在以下条件下对光刺激表现出直接响应:(1)在突触阻断阶段刺激开始后的10毫秒内,它们的放电率相比于预刺激基线增加了超过3.3个标准差(使用0.1毫秒的箱型计算,并用标准差为0.5毫秒的因果高斯滤波器平滑处理),(对于ChR2而言)或减少了超过3.3个标准差(对于GtACR2和ArchT而言);(2)它们被记录在药物阻断确认的深度;以及(3)在突触阻断阶段诱发的尖峰波形与预刺激基线阶段记录的尖峰波形相匹配。
Construction of 3D autocorrelograms
Para_01
- 所有记录都是在清醒的动物中进行的,这些动物要么是头部固定但可以自由移动在轮子上(小鼠),要么是在执行平滑追随试验(灵长类动物),这导致了行为驱动的放电率在整个实验过程中发生变化。
- 为了使放电率变化对放电统计量测量的影响标准化,我们构建了"三维自相关图"(3D-ACGs)。
- 在每个时间点,我们估计神经元的瞬时放电率为逆间期间隔。
- 我们使用箱型滤波器(250毫秒宽)平滑放电率,并测量每次脉冲时平滑后的瞬时放电率时间序列的值。
- 接下来,我们确定了放电率的分布,通过在每次记录中的脉冲时刻进行评估,将放电率分布分层为10个十分位数,并为每个十分位数内的脉冲计算单独的二维自相关图(2D-ACGs)。
- 我们将得到的3D-ACGs可视化为一个表面,其中颜色轴对应于放电概率,y轴分层放电率十分位数,因此每个3D-ACG包含10行,而x轴表示从触发脉冲开始的时间。
- 请注意,自相关图中的脉冲计数已除以箱宽,因此y轴或颜色映射以每秒脉冲(spikes/s)为单位。
- 作为分类器的输入,我们使用相对于t=0的对数分布箱,而不是在图和补充信息中显示的线性间距箱。
Human expert labeling of cerebellar units
我们对梅迪纳实验室收集的一个未标记的小鼠数据集进行了人类专家细胞类型识别。我们使用Phyllum来识别每个记录的层次。大多数浦肯野细胞通过简单尖峰和复合尖峰的存在以及复合尖峰触发的直方图得以识别,这些直方图显示了在复合尖峰之后简单尖峰放电率的特征性暂停。我们通过简单尖峰的存在、位于浦肯野细胞层、以及产生自相关图中特征性‘肩部’的规则放电率来识别潜在的浦肯野细胞。潜在的分子层中间神经元通过它们位于分子层且放电率高于5次/秒得以识别,这与树突复合尖峰的放电特性不兼容。潜在的苔藓纤维位于颗粒细胞层,并且一些由于在glomerulus附近记录到的负后波而显示出特征性的三相形状。潜在的高尔基细胞位于颗粒细胞层,具有宽阔的波形和相对规律的放电率。
猴。我们在Lisberger实验室收集的一个未标记的猴数据集中进行了人类专家细胞类型识别。我们将记录分类为基准真确的浦肯野细胞,如果它们展示了简单尖峰放电中特有的复合尖峰后暂停。表现出已知的浦肯野细胞简单尖峰特征但缺乏复合尖峰的单元被处理为‘假定’浦肯野细胞,并用于比较分类器预测和专家预测的标签。我们只将分子层中间神经元纳入样本,如果它们在短潜伏期内显示出对某个确定的浦肯野细胞简单尖峰的触发抑制,这使一些潜在的分子层中间神经元未能被纳入我们的样本。我们仅将单元作为假定的苔藓纤维包括进来,如果波形显示负后波,这是靠近单个glomerulus记录的特征。我们注意到,我们对苔藓纤维的分类非常保守,很可能使一大部分不在glomerulus附近的苔藓纤维记录未被标记。假定的高尔基细胞通过它们存在于颗粒细胞层、宽波形和高度规律的放电被识别出来,与之前的记录一致。猴中的单元专家标注是在收集和分析小鼠基准单元之前进行的。
Classifier design
分类器设计
Para_01
- 我们通过选择细胞类型分类器的"超参数"开始了设计:传递给模型的特征空间、模型类别以及模型特性,如单元数量和学习率。
- 我们决定独立于真实数据集选择超参数是至关重要的,这有助于通过最小化过拟合来确保泛化能力。
General classifier architecture
Para_02
- 为了生成分类器的输入,我们使用了两个预训练的自动编码器来降低地面真值库中神经元波形和3D-ACG的维度。
- 这两个自动编码器的输出以及每个神经元的层级被用作在地面真值数据集上训练最终分类器的输入。
- 如下面详细说明的那样,模型特征空间或其架构的任何方面都不是基于模型在地面真值数据集上的性能选择的。
- 我们的分类器是一种"半监督"模型,因为它使用了两个经过调整和无监督学习预训练的变分自动编码器,这些编码器是在一组未标记的神经元上进行训练的,而完整的分类器则是在另一组地面真值识别的神经元上进行监督学习训练的。
Feature engineering
Para_02
-
为了构建一个无偏的特征空间来训练模型,我们预先决定模型的输入将是解剖位置、细胞外波形和存在于3D-ACG中的放电统计。
- 我们选择不使用汇总放电统计,因为与我们选择的输入相比,它们提供的信息量较少。
Unsupervised pre-training with variational autoencoders
Para_02
- 我们使用了两个变分自编码器来降低特征空间的维度,并通过利用n=3,090个在创建地面实况库所用实验中记录的但未标记的单元,完全独立于我们的地面实况数据集优化了模型的架构,这些单元没有通过光遗传学激活。
Para_03
- 我们预训练了两个自动编码器来重构我们的未标记单元的波形和对数缩放的三维自相关图。
- 最终,自动编码器将输入数据压缩到两个10维的‘潜在空间’中,分别对应于输入特征:三维自相关图和波形。
- 潜在空间具有高斯先验,这鼓励每个网络单元在未标记的数据集中具有零均值和单位方差的激活值。
- 变分自动编码器的训练目标是经过修改以包含一个β项的‘证据下界’损失,以促进潜在空间的解耦。
- 在训练过程中,我们采用了Kullback-Leibler散度退火程序以增强模型的稳定性和收敛性。
-
两个变分自动编码器通过梯度下降与Adam优化器进行训练,并辅以余弦退火学习率策略和周期性的热重启。
波形变分自编码器由一个具有高斯误差线性单元(GeLU)非线性的2层感知机(2LP)编码器和同样具有GeLU非线性的2层感知机(2LP)解码器组成。它使用η=1e-4,β=5的学习率和批量大小为128进行了60个周期的训练。
该3D-ACG变分自编码器由一个包含卷积层和平均池化、批归一化以及修正线性单元(ReLU)非线性的2层卷积神经网络(CNN)编码器,和一个具有ReLU非线性的2层感知机(LP)解码器组成。它经过了60个周期的训练,学习率为η=5e-4,β=5,小批量大小为32。
Semi-supervised classifier
Para_02
- 完整的分类器模型依次由以下部分组成:(1) 使用未标记数据预训练的波形和3D-ACG变分自编码器,以减少输入特征的维度;(2) 多头输入层,接受波形和3D-ACG变分自编码器的潜在空间,以及神经元小脑层的"一位热编码"的3位二进制代码;(3) 一个具有100个单元的单个全连接隐藏层,处理这3个输入;(4) 输出层,每个细胞类型有一个输出单元。5个输出单元的值通过softmax函数相加至1,使得分类器的输出是给定输入来自5种细胞类型的概率。在输入层和全连接隐藏层之间,我们应用了批次标准化,以使波形、放电统计数据和层次的贡献相等。
- 为了统一波形、放电统计数据和层次的贡献,我们在输入层和全连接隐藏层之间应用了批次标准化(参考文献编号89)。
Supervised training procedure
Para_02
- 我们在地面真实图书馆的数据上使用梯度下降和留一交叉验证策略训练了完整分类器的权重。
- 我们训练模型直到收敛或最多20个周期,以先到者为准,学习率为η=1e-3,小批量大小为128,并使用AdamW优化器。
- 在优化过程中,我们允许预训练变分自编码器中的权重发生变化,以便进行微调,从而在下游分类任务中获得小幅性能提升。
Strategies to mitigate overfitting
我们通过预训练两个变分自编码器(见上文)来减少输入维度,以最小化最终分类器中新训练的参数数量。
我们采用了旨在正则化高度表达性模型(如深度网络)的数据增强策略。我们总共构建了7种自定义数据增强方法,其中2种适用于波形,5种适用于尖峰列车。
我们在分类器中使用了dropout层,以确保模型不会过度依赖任何一个单一特征或神经元。
我们在训练模型时,在训练集上出现最早收敛迹象时就通过早停法中断了训练。
我们的最终模型是一个集成模型,它结合了多个模型的预测结果来生成输出,并有助于减少与单个模型相关的方差。集成方法通过平均各个模型的误差来减轻过拟合。
Para_02
- 分类器在来自猴子和小鼠的独立数据集上与专家分类的一致性良好,这为分类器不过度拟合提供了进一步的证据。
Assessment of autoencoder operation
Para_02
- 为了生成图S5D中的图形,并评估分类器中的变分自动编码器是否能够在潜在空间中表示广泛的输入统计数据并捕捉数据中的方差,我们向每个自动编码器的解码部分提供了新颖的输入,并可视化了由此产生的重构。
- 因为供给解码器的值可以在10个维度上变化(对应于10维的潜在空间),为了可视化重构,我们确定了潜在空间中占训练数据集方差大部分的两个维度。
- 我们利用编码网络将3090个未标记单元的波形和3D-ACG编码到一个潜在空间。
- 在这个空间内,我们进行了主成分分析以识别解释方差最多的两个成分。
- 我们通过这两个主成分的加权和生成了自动编码器解码部分的新颖输入。
Para_03
- 每个重构波形和图S5D中的8x8矩阵中3D-ACG的位置对应于应用于第一主成分(水平轴)和第二主成分(垂直轴)的权重的相对位置。
- 这些权重被选择为均值为零的高斯分布的八分位数,其标准差被选为产生代表通过这些主成分观察到的波形和3D-ACG分布的标准。
- 我们选择高斯分布中的权重,因为通过分配的标准正态先验,鼓励潜在空间中每个单元上的激活分布为零均值高斯分布,但类似范围内的其他权重选择将产生定性上相似的结果。
- 我们推断,如果自动编码器过拟合或未标记数据集中的一组波形和3D-ACG不够多样化,我们将观察到重构输出中的不连续性而不是可以容纳广泛输入统计的平滑过渡。
Evaluation of classifier performance
分类器性能评估
Cell-type classification of ground-truth neurons
为了应对由每种细胞类型中神经元数量不同所导致的‘类别不平衡’问题,我们在将数据拆分为测试集和验证集后,对每个模型都对代表性不足的细胞类型进行了随机过采样。
-
我们通过留一交叉验证评估了所有模型的表现,该方法相较于其他交叉验证方法具有较低的偏差和可比较的方差,并且在过去已被用于评估小数据集的表现。
我们采用了一种策略来防止置信度误校准,即深度神经网络在预测中表现出过度自信的趋势。我们通过在输出层应用最后一层的拉普拉斯近似来纠正每个模型实例的过度自信。
我们通过在模型的10次实例中对每个细胞类型预测的概率取平均来量化分类器的置信度。我们将置信比计算为输入特征中每个细胞的最高预测细胞类型与次高预测细胞类型的概率之比。我们选择2作为置信阈值,但可以应用更高的阈值以增加每个细胞类型预测的置信度。许多真实标签的神经元在模型的10次实例中被分配了相同细胞类型的高概率。然而,这不一定是这种情况:如果给定单元的数据与多个细胞类型兼容,则分类器可能在一个模型实例中将该单元高度概率地分类为细胞类型#1,在另一个实例中高度概率地分类为细胞类型#2:分类器在10次运行中的平均概率可能是相似的,因此对于这两种细胞类型接近0.5,这表明分类器的置信度较低。
Cell-type classification of unlabeled mouse and macaque neurons
Para_01
- 我们使用了一个集成分类器来预测未标记的小鼠(Medina)和猕猴(Lisberger)小脑神经元的细胞类型,这些神经元没有参与分类器的训练过程。该集成分类器利用了所有真实标签的神经元和初始条件(总共2020个模型)。
- 每个模型都做出了独特的预测,因为由于使用了(i)10种不同的初始条件进行模型训练,以及(ii)由于‘留一法’程序而对不同子集的神经元进行训练,每个模型都有所不同。
- 在未标记样本中的每个神经元的预测细胞类型被选择为其在2020个模型中平均预测的最大值。
- 我们在分析真实标签库中的细胞类型分类时应用了置信比和置信阈值的方法。
Analysis of neural dynamics during behavior
行为期间的神经动力学分析
Behavioral paradigms
Häusser:数据是在小鼠进行自主发起的运动时记录的。手术后五天,小鼠开始限制饮水并适应头部固定在跑步轮上的过程。每天大约30分钟,小鼠被固定在轮子上,并且如果它们向前移动则会被奖励几滴水。在记录当天,小鼠通常在大约一小时内跑完150米。运动使用Vision Mako U-130B摄像机以每秒100帧的速度记录,动物同侧前爪使用DeepLabCut131进行追踪。通过在极坐标系中对爪轨迹进行阈值处理(从希尔伯特变换获得相位)来识别前爪摆动的起始点,并用于对齐神经数据。
在实验前至少五天,小鼠被限制饮水,然后在自由移动的轮子上适应头部固定和奖励递送六天。一个用于递送奖励的管子被放置在小鼠前方,并配有红外发光二极管和光电二极管以检测舔食行为。奖励由用糖精(10 mM)甜化的水组成,通过可听电磁阀每23-85秒递送一次,共进行了218次试验。我们将同时记录的神经元反应与提示奖励递送的电磁阀点击声对齐。
Medina:小鼠通过经典条件反射协议进行了训练,在该协议中,LED(条件刺激)的存在预示着220毫秒后角膜会出现气雾(非条件刺激)。实验的具体方案已在先前的研究中描述。65动物已经接受了广泛的训练,并在神经记录开始前产生了可靠的条件反射。我们对来自单次记录中的许多神经元的同时响应进行了对齐,以条件刺激的启动为基准。
Lisberger:我们在平滑追逐目标运动的离散试验中记录了神经数据。动物被安置在阴极射线显像管显示器前,并训练去追逐一个黑色圆点的平滑移动,该圆点以恒定速度朝八个方向之一移动。在这里,我们只包括目标在水平方向向我们进行神经记录的半球一侧移动的试验,并且猴子成功追踪了目标并在目标运动结束后保持注视。数据与目标运动开始对齐,目标运动持续650毫秒。
Neural data trajectory analysis
Para_01
- 我们分析了每个被我们的分类器以超过2的置信比标记的记录神经元,仅分析每只小鼠的三种行为中的一个会话,以及伪群体在n=163个会话中对猴子的行为进行记录,类似于先前对群体动态的分析。
- 我们将脉冲列车转换为放电率,并使用因果核对其进行了时间平滑处理,这些因果核适合于每种行为范式(10-50毫秒平滑时间常数)。
- 由于行为任务的时间尺度不同(例如,在奖赏条件反射期间大约为200毫秒,但在平滑追随期间则超过1000毫秒),需要使用特定任务的平滑方法。
- 然而,我们注意到,我们的总体结论对于广泛的平滑时间常数是稳健的。
- 平滑后,我们通过标准化每个神经元在其100毫秒预试验基线期间活动的标准差来归一化其放电,将离散试验的反应平均以形成每个神经元的潜刺激时间直方图,并从潜刺激时间直方图中减去预试验基线期间的平均放电率。
- 因此,归一化放电率的变化相对于它们的基础活动表示为基线放电率标准差的倍数。
- 图7B中的PSTHs是所有细胞类型的实例的平均值,图7C中的热图显示了每个细胞类型在群体中的单个试验平均PSTH。
Para_02
- 一种更复杂的方法是利用降维技术来研究大规模群体反应的时间结构,以理解存在于群体中各个神经元之间的一致时间特征。
- 为了回答特定细胞类型群体活动的低维表示是否与无细胞类型特异性群体不同,我们开发了一个分析流程,可以比较不同大小群体的神经轨迹在一个公共空间中的表现。
- 在每种情况下,我们首先构建了一个维度为NxT的矩阵X,其中每一行包含了N个神经元中一个神经元的基线归一化和平滑后的刺激时间直方图,共有T个时间点。
- 为了应用主成分分析,我们将X矩阵中每一行的发放率进行了中心化。
- 由于主成分分析按方差顺序识别维度,那些相对于其基线发放率显示出显著差异的神经元可能会主导大部分方差。
- 为了缓解在没有归一化的情况下高度响应神经元过度代表的问题以及在Z-score情况下非响应神经元过度代表的问题,我们利用了一种先前描述的通过‘软’归一化程序对发放率进行预处理的方法。
- 我们通过确保高活性神经元(其调制超过基线标准差两倍以上)的范围接近于一来降低它们的影响。
- 相比之下,非响应神经元的发放率变化范围被减小到接近零的值,因此不会对最大的主成分产生实质性贡献。
Para_05
- 因为不同细胞类型的群体规模不相等,我们使用置换检验来评估每种细胞类型最优旋转和反射轨迹与从总体群体中得出的细胞类型无关轨迹之间的距离的统计显著性。
- 我们检验了零假设,即随机选择相同比例的细胞用于比较的轨迹将显示出相似的欧几里得距离分布。
- 我们进行了1,000次置换,在每次置换中,我们从两个比较的群体中抽取神经元,将这些置换后的轨迹最优旋转和反射到同一空间,然后推导出距离的零分布。
- 从经验上推导出的零分布使我们能够直接评估两个群体轨迹之间距离的显著性。
Para_06
- 我们也测试了当我们在分类器提供的细胞类型标签中引入随机误差时,轨迹分析的性能。
- 对于每个给定的重标记细胞比例,我们进行了1,000次重复实验,随机选择一组细胞接收新的细胞类型标签,这些标签对应于浦肯野细胞简单尖峰、高尔基细胞、苔藓纤维或分子层中间神经元之一,概率相等。
- 然后,我们选择了具有特定细胞类型标签的所有神经元,如上所述进行降维处理,并计算最优旋转和反射后的低维轨迹与不受细胞类型标签影响的轨迹之间的距离。
- 我们使用自助法统计来根据随机分配新细胞类型标签的细胞比例得出95%的置信区间。
Supplemental information
Para_01
- 下载:下载 Acrobat PDF 文件(290KB)