Phytomedicine作为药学领域的知名顶刊,虽然影响因子不是很高,但含金量非常高,发表在这本期刊上的文章质量也是非常高的。
这本期刊上的大部分文章都是干湿结合方向,纯生信目前是很难见到的。
但是,宁夏医科大学团队近期却做到了0实验在该期刊发文,怎么办到的呢?跟馆长一起看看吧~
选题上,该研究聚焦生姜的抗胃癌作用,药食同源主题是当下中医药领域的热门研究方向,该研究为生姜的临床应用和靶向药物开发提供了全新方向。
研究思路方面,结合了网络药理学、WGCNA和多种机器学习算法(如LASSO、SVM和随机森林),多种方法整合能够提高筛选生物标志物的准确性和效率。除此之外,针对筛选到的核心基因作者进一步进行了临床相关性、遗传相关性、免疫相关性等分析,并通过分子对接进行验证,内容丰富,逻辑严谨。
通过这篇文章也可以看出来,选题选对了就相当于成功了一半。对于掌握了生信基础分析技能的朋友可以确定好选题模仿这个思路来一篇。不会选题、生信分析有困难或者没有服务器进行分子对接分析的朋友直接滴滴馆长即可!

该研究旨在通过网络药理学、WGCNA和机器算法筛选生姜抗胃癌(GC)的生物标志物。寻找构成信号轴的上游转录因子和下游信号蛋白,预测生姜抗胃癌可能的作用机制。
设计思路
1.生姜活性成分及靶点预测
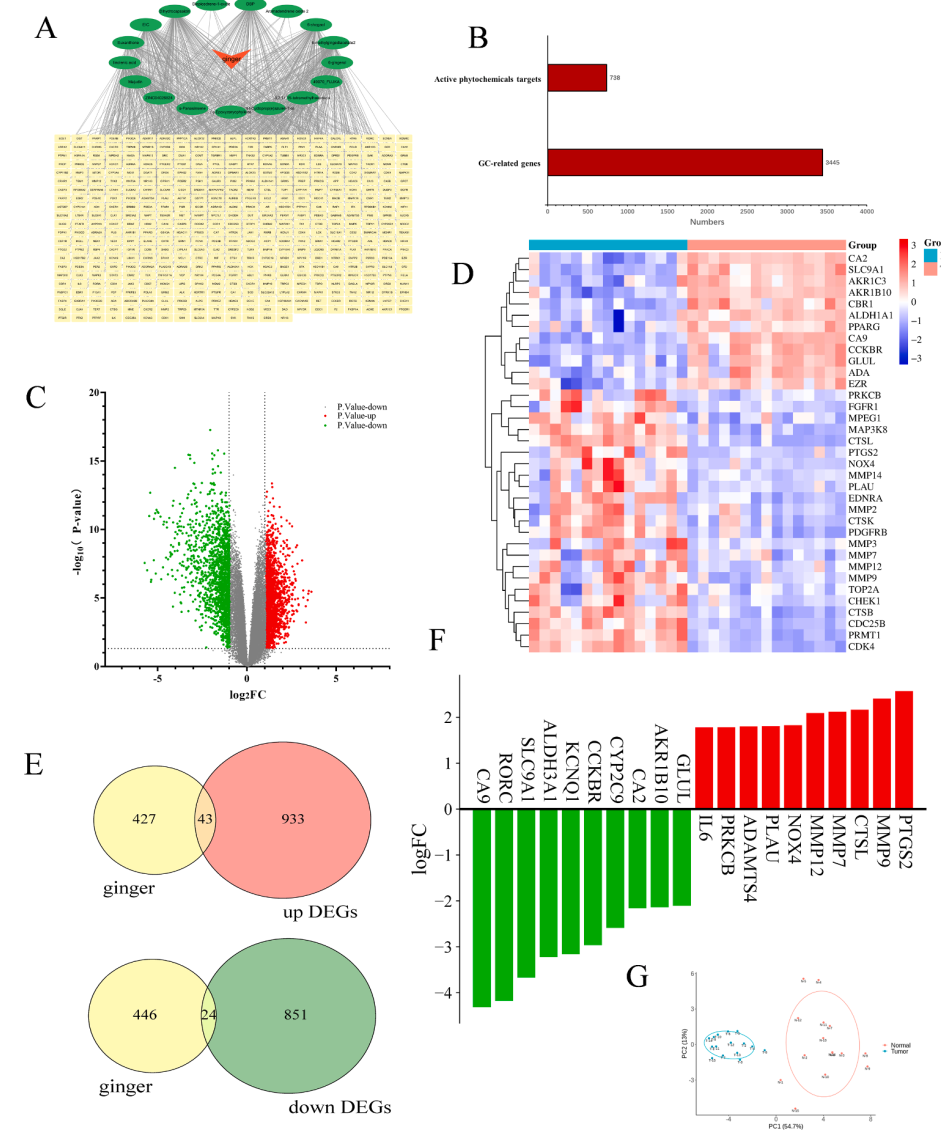
利用TCMSP 数据库筛选活性成分,通过Swiss Target Prediction和SEA 数据库预测潜在靶点。
2.胃癌相关基因筛选
1) 从Genecards、OMIM、Disgenet 数据库获取胃癌靶点;
2) 结合GEO 数据库的GSE118916 数据集,筛选差异基因。
3.核心靶点筛选
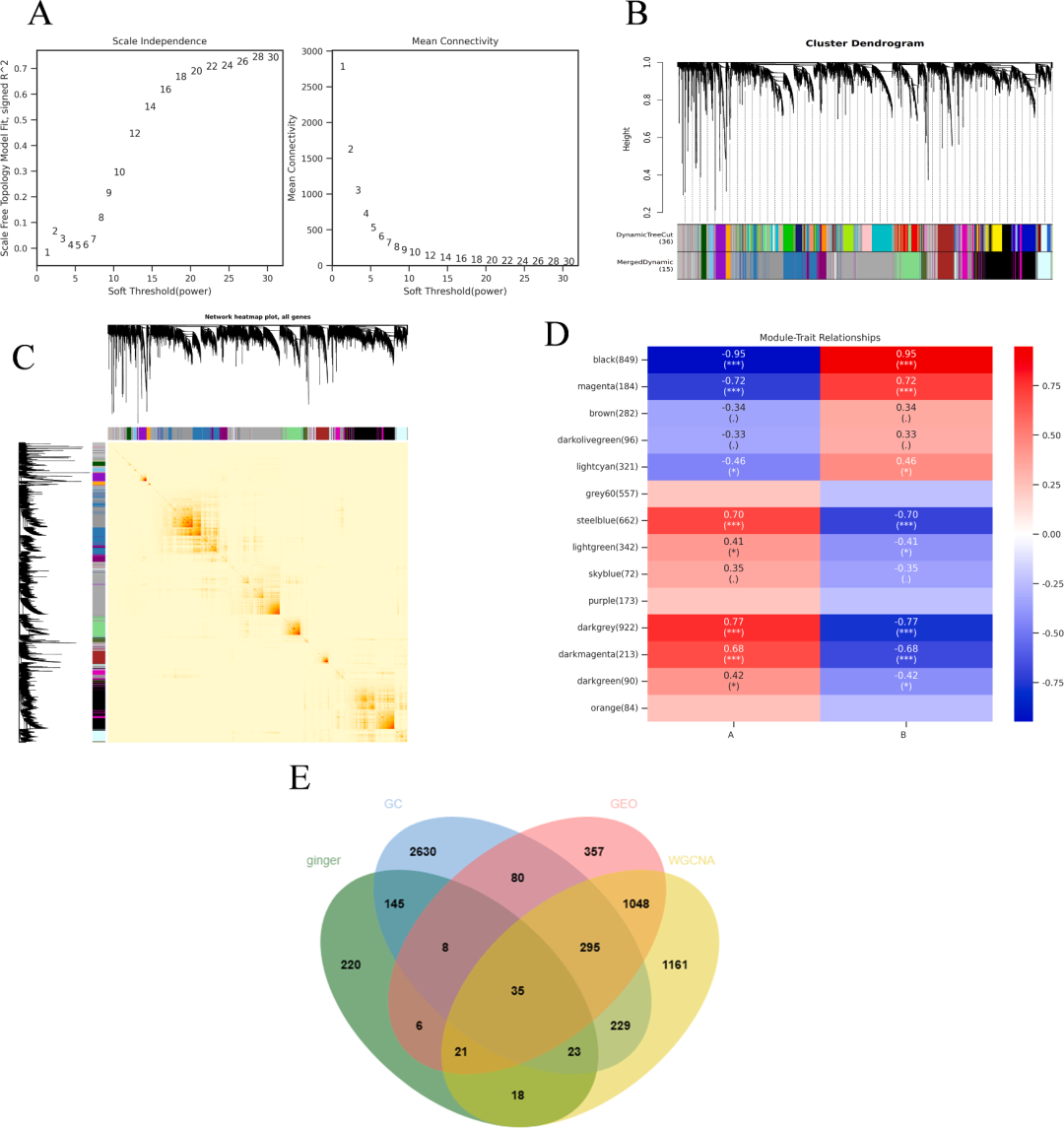
1)基于 GEO 差异基因进行WGCNA分析,筛选关键基因
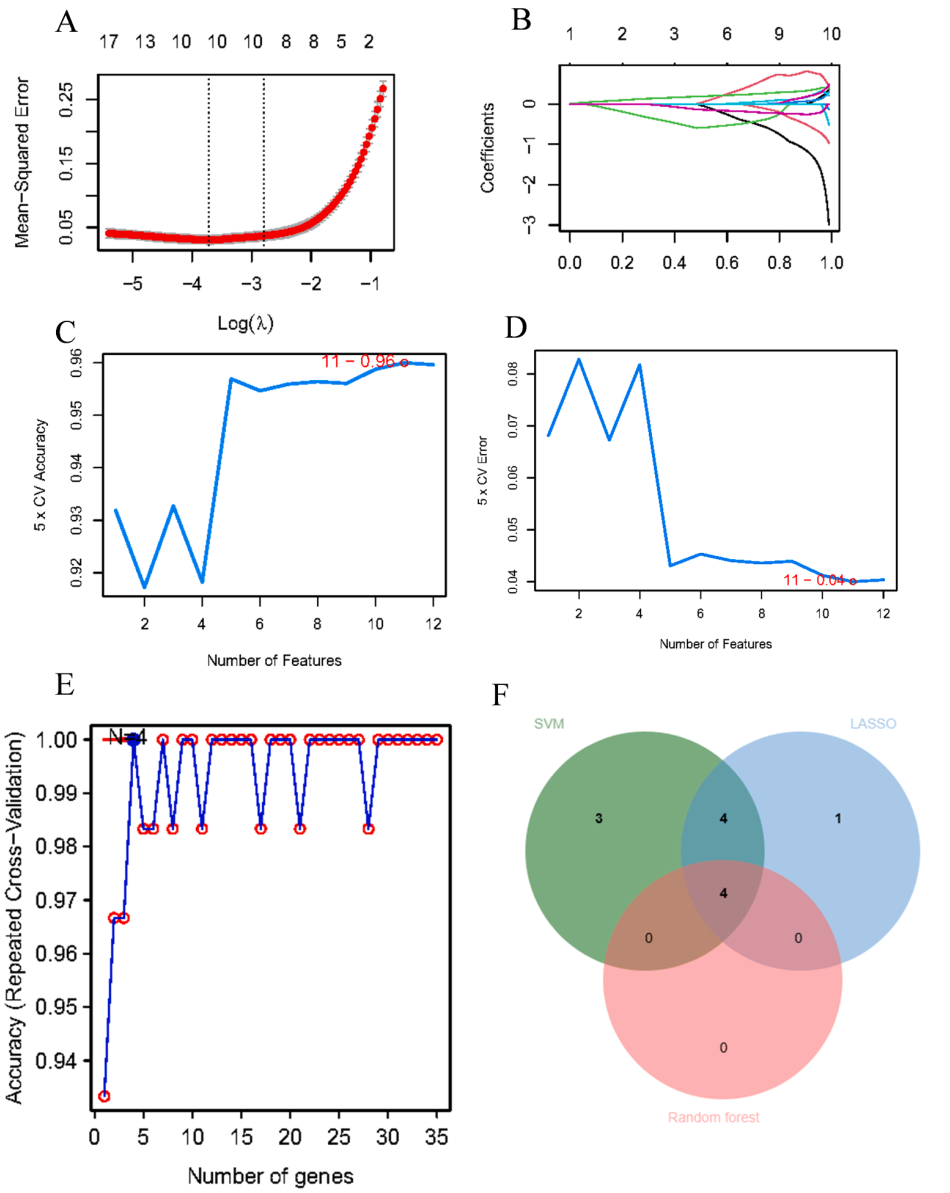
2)结合随机森林、LASSO、SVM-RFE进一步筛选,最终结合PPI 网络确定关键靶点。
4. 临床相关性分析
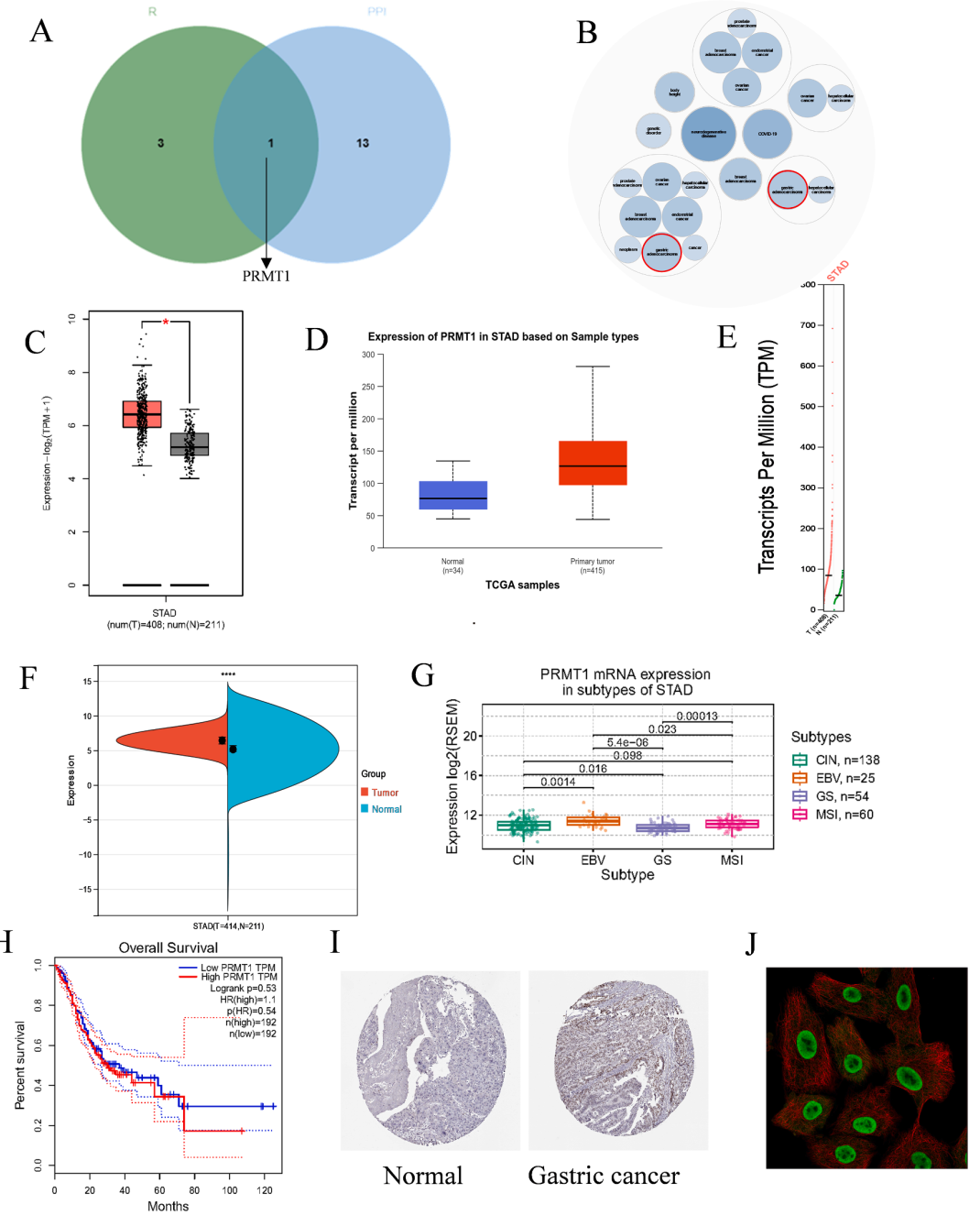
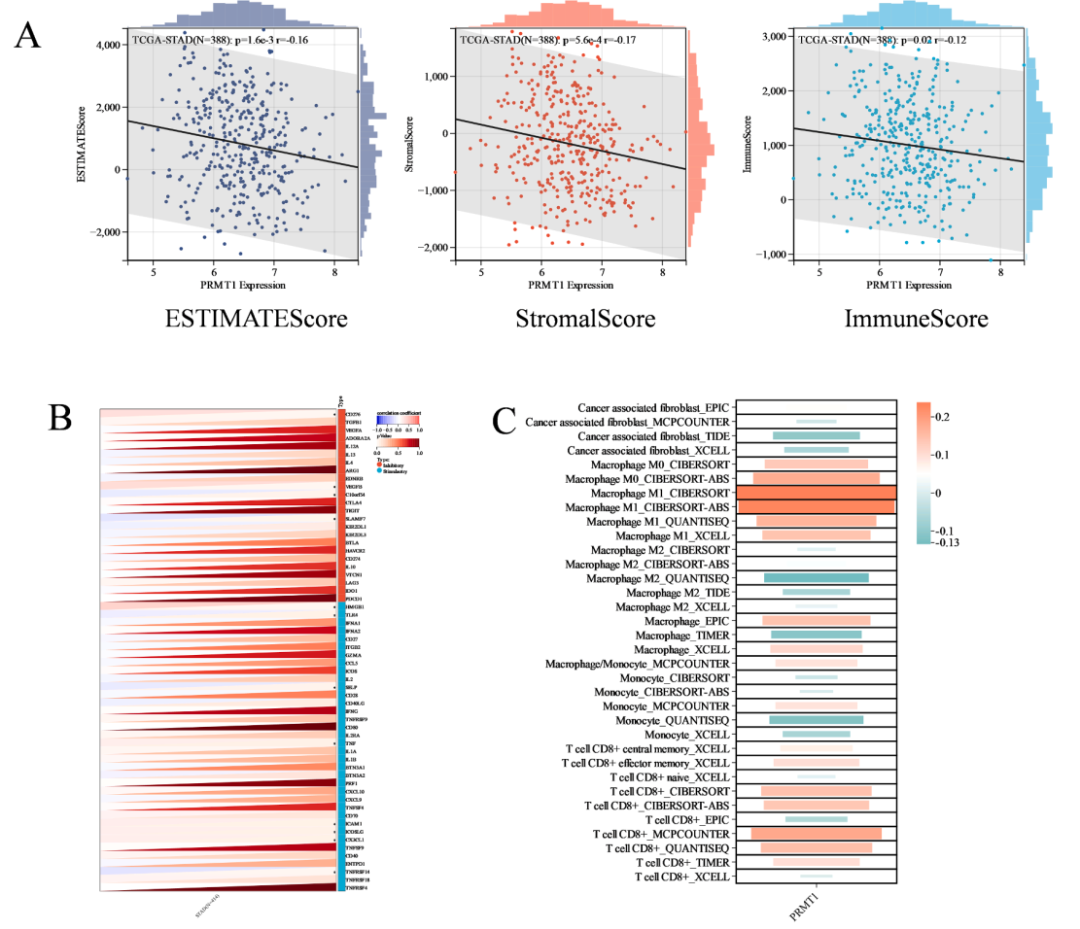
利用公共数据库分析 PRMT1 在胃癌中的表达水平、拷贝数变异及与临床病理特征的关联,以及与表观遗传调控、免疫细胞浸润等的关系。
5. 分子对接
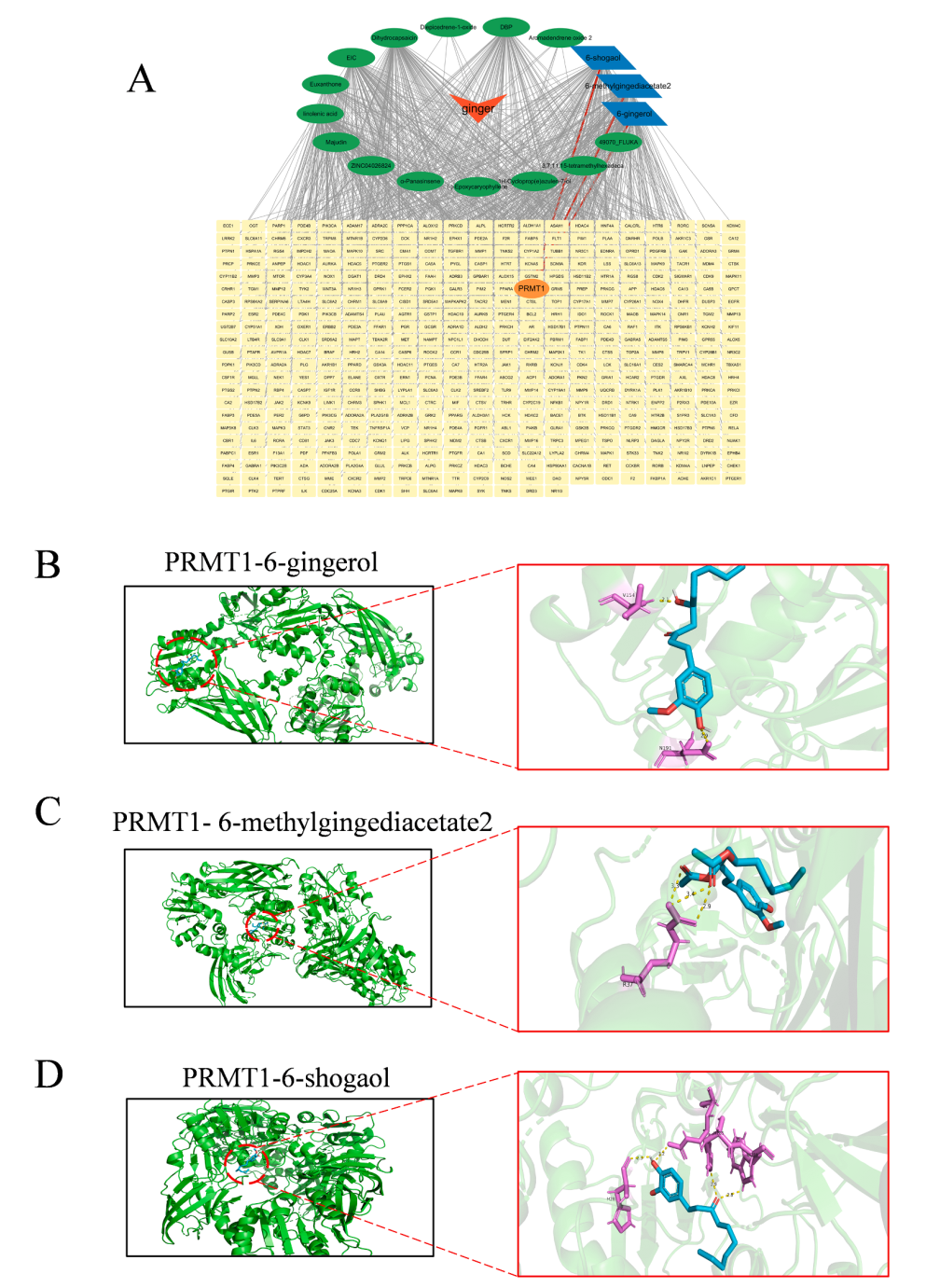
使用Cytoscape 3.9.1软件和CB-DOCK2在线分析网站,对PRMT1与生姜的活性成分进行分子对接验证。(分子对接计算量大,建议通过服务器完成,有需要欢迎滴滴馆长~)
6. 上下游分子筛选
通过数据库筛选 PRMT1 上游转录因子,预测下游蛋白。
主要结果
姜有效成分的作用靶点及GC中差异表达基因
WGCNA筛选核心基因
机器学习筛选核心基因
核心基因的临床相关性
核心基因与免疫浸润的关系
核心基因和生姜活性成分的分子对接
馆长点评
该研究结合“药食同源”这一热门选题方向,通过建立“数据库预测→机器学习算法筛选→分子对接验证→多组学关联分析”的研究流程,对传统中药姜的抗胃癌作用进行了系统研究,为药食同源植物的抗病机制研究提供了方法模板。
这篇文章整体上虽然也是网药+分子对接的研究思路,但是创新性地加入了与临床、表观遗传、免疫浸润等的相关性研究,这种多组学结合的方式不仅丰富了文章的研究内容,同时也增加了研究思路的创新性
。中医药研究思路正在不断升级,想通过中医药发文的朋友可以关注一下馆长分享的各种创新思路,选题或者分析方面遇到瓶颈的也可以直接滴滴馆长,专业团队,经验丰富,助力您早日完成KPI!
馆长会持续为大家带来最新生信思路,也可以提供特色数据库构建、思路评估、生信分析等服务,对数据库构建和生信分析感兴趣的朋友可以咨询馆长哦!